DNA甲基化是表观遗传领域的核心机制之一,是指在DNA甲基转移酶(DNMTs)的催化下,将甲基基团精准添加到DNA分子特定区域(如富含CpG二核苷酸的CpG岛)的过程,这一修饰不改变DNA碱基序列,但能显著调控基因表达。其中,胞嘧啶第5位碳原子上的甲基化修饰(即5-甲基胞嘧啶,简称5mC)是最普遍且研究最深入的类型,尽管其存在具有跨物种普遍性,但不同生物的甲基化程度差异显著:例如拟南芥中约14%的胞嘧啶发生甲基化,小鼠体内比例约为7.6%,而果蝇和果盘杆菌分别低至0.03%和0.006%,秀丽隐杆线虫与部分真菌则几乎不发生此类修饰。
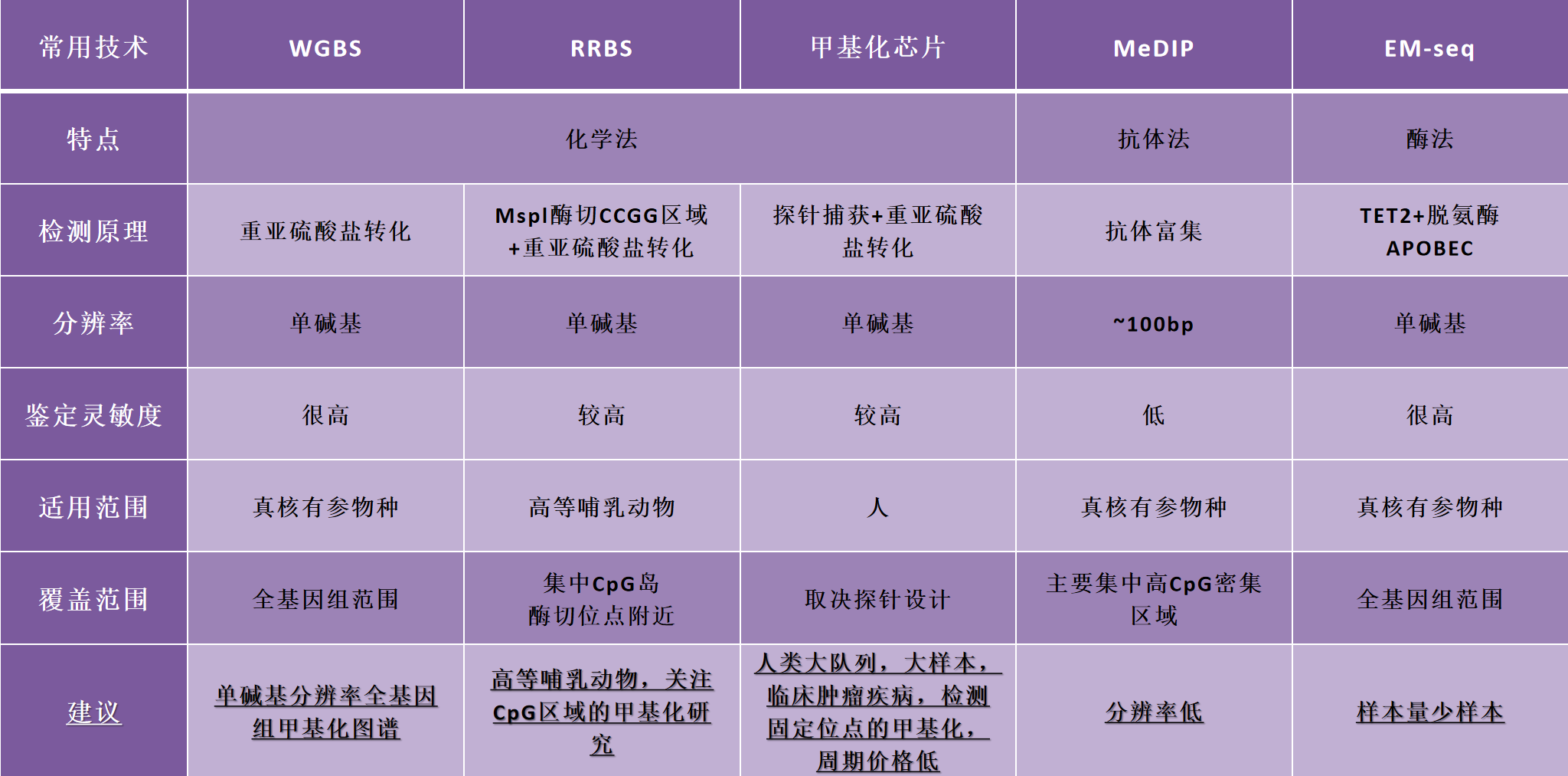
针对不同研究需求,目前有多样化的DNA甲基化分析测序技术,包括WGBS、RRBS、甲基化芯片、MeDIP和EM-seq等等。那么不同技术有什么特点呢?这篇文章给您答案。
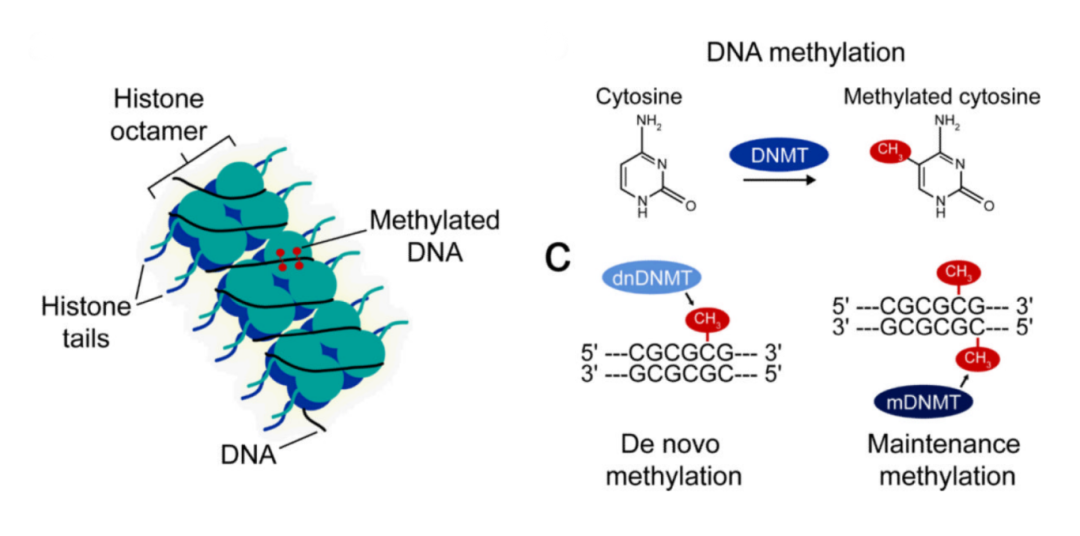
图 DNA甲基化(Day et al., 2010)。
一、全基因组甲基化测序(WGBS)
01 技术简介
WGBS是甲基化研究的“金标准”技术之一。其核心原理是利用亚硫酸氢盐处理基因组DNA,将未甲基化的胞嘧啶(C)转化为尿嘧啶(U),而甲基化的胞嘧啶(5mC)保持不变;随后通过高通量测序,结合生物信息学分析,精准区分甲基化与未甲基化的胞嘧啶,从而获得全基因组范围内单碱基分辨率的甲基化图谱。
02 技术优势
(1)覆盖范围广:可检测全基因组所有胞嘧啶位点的甲基化状态,能提供全面且详细的甲基化信息,无偏倚地呈现甲基化全景。适合挖掘新的甲基化位点及区域。
(2)分辨率高:达到单碱基水平,是当前分辨率最高的甲基化检测技术之一。能精准定位甲基化位点的具体位置,为精细解析甲基化调控机制提供基础。
(3)结果可靠:作为行业金标准,其检测结果的准确性和重复性得到广泛验证,是验证其他甲基化技术结果的重要依据。
03 技术劣势
(1)数据量大:产生的海量数据对存储设备和分析软件的要求较高,需要专业的生物信息学团队进行数据处理和解读。
(2)样本需求量大:亚硫酸氢盐处理对初始样本的DNA质量和数量要求较高,不适合应用于微量样本。
04 应用场景/适用范围
(1)基础表观遗传学研究:如解析物种甲基化图谱、探索甲基化在细胞分化、发育中的动态变化规律。
(2)疾病机制研究:筛选疾病相关的全基因组甲基化差异位点,揭示甲基化异常在肿瘤、神经退行性疾病等发生发展中的作用。
(3)标志物发现:从全基因组层面挖掘潜在的疾病诊断、预后相关甲基化标志物。
——适用于样本量充足、需全面解析甲基化模式的情况,如全基因组甲基化图谱构建、肿瘤发生机制的系统研究等。
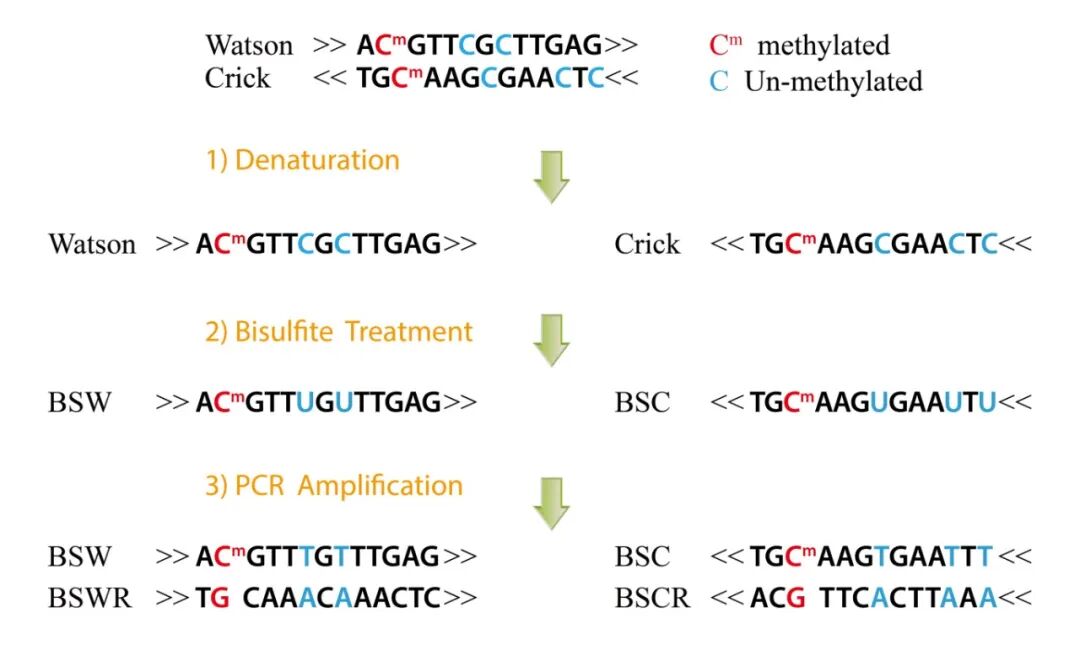
图 BS-seq实验原理。
二、简化甲基化测序(RRBS)
01 技术简介
RRBS是一种简化基因组甲基化测序技术,它利用限制性内切酶切割基因组DNA中富含CG的区域(CpG岛),通过回收特定长度的酶切片段,再进行亚硫酸氢盐处理和测序,从而聚焦于基因组中甲基化调控关键区域的甲基化状态分析。
02 技术优势
(1)成本较低:通过酶切富集CpG岛区域,大幅减少测序区域范围,降低测序数据量和成本。
(2)靶向性强:聚焦于CpG岛等甲基化调控核心区域,这些区域往往与基因表达调控密切相关,能高效挖掘关键甲基化位点。
03 技术劣势
(1)覆盖范围有限:仅覆盖酶切位点相关的CpG岛区域,无法检测全基因组范围内的甲基化位点,可能遗漏非CpG岛区域的关键甲基化信息。
(2)依赖酶切效率:酶切效率受样本DNA质量、酶活性等因素影响,可能导致不同样本间的覆盖均匀性差异。
04 应用场景/适用范围
(1)适用于大规模样本的甲基化筛查:如临床样本队列的疾病相关甲基化标志物初步筛选。
(2)聚焦CpG岛区域的研究:如基因启动子区CpG岛甲基化与基因沉默的关联研究。
——适用于经费有限、样本数量多、聚焦CpG岛等核心调控区域的情况。
三、甲基化芯片测序
01 技术简介
甲基化芯片是一种基于芯片技术的高通量甲基化检测方法。通过在芯片上固定大量针对CpG位点的探针,结合亚硫酸氢盐处理后的DNA杂交,实现对特定CpG位点甲基化水平的定量检测。
02 技术优势
(1)高通量:一次可检测数十万个CpG位点,能同时处理大量样本,检测效率高。
(2)标准化程度高:探针设计固定,实验流程标准化,不同批次、不同实验室间的结果可比性强,适合大规模队列研究。
(3)数据分析简单:无需复杂的全基因组测序数据处理流程,数据分析门槛较低。
03 技术劣势
(1)覆盖范围固定:仅能检测芯片探针设计好的CpG位点,无法检测未设计探针的区域。
(2)分辨率有限:虽能定量检测甲基化水平,但无法发现新的甲基化位点,仅适用于已知位点的检测。
(3)物种局限性:目前成熟的甲基化芯片主要针对人类、小鼠等模式生物,其他物种的芯片选择较少或需定制。
04 应用场景/适用范围
(1)大规模临床样本的甲基化标志物验证:如将WGBS或RRBS筛选到的候选标志物通过芯片在大样本队列中验证。
(2)疾病表观遗传分型:基于甲基化芯片数据对疾病样本进行分型,探索不同亚型的表观遗传特征。
(3)表观遗传关联研究(EWAS):通过大样本芯片检测,挖掘与疾病、表型相关的甲基化位点。
——适用于人类等模式生物的大规模样本已知CpG位点检测、标志物验证、疾病分型等场景,是临床研究和EWAS研究的常用技术。
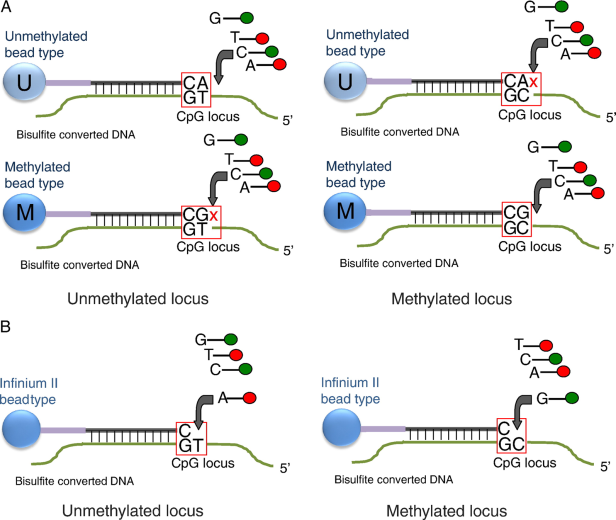
图 甲基化芯片检测原理(Ross et al., 2022)。
四、甲基化DNA免疫共沉淀测序(MeDIP-seq)
01 技术简介
MeDIP-seq是一种基于免疫共沉淀的靶向甲基化测序技术。其原理是利用特异性识别甲基化胞嘧啶(5mC)的抗体,与基因组DNA片段进行免疫共沉淀反应,富集甲基化的DNA片段,随后通过高通量测序分析富集片段的序列信息,从而确定基因组中甲基化区域的分布。
02 技术优势
(1)覆盖范围较广:可检测全基因组范围内的甲基化区域,无需提前知道甲基化位点信息,能发现新的甲基化区域。
(2)成本适中:相比WGBS,通过富集甲基化片段减少了测序数据量,成本更低;相比RRBS,覆盖区域更广泛。
03 技术劣势
(1)分辨率较低:仅能定位到甲基化区域,无法达到单碱基分辨率,难以精准确定具体的甲基化位点。
(2)富集偏倚:抗体对高甲基化区域的富集效率更高,对低甲基化区域的检测灵敏度较低,可能导致低甲基化区域的信息遗漏。
04 应用场景/适用范围
(1)新物种甲基化区域的初步筛查:如非模式生物的表观遗传研究,挖掘其甲基化调控区域。
(2)全基因组甲基化区域的差异分析:如比较疾病组与正常组的全基因组甲基化区域差异,筛选异常甲基化区域。
——适用于需全基因组范围甲基化区域筛查但无需单碱基分辨率的研究场景。
五、氧化亚硫酸氢盐测序(EM-seq)
01 技术简介
EM-seq是一种基于酶学反应的DNA甲基化检测技术,通过两步酶促反应实现单碱基分辨率的甲基化分析。首先使用TET2酶和T4-BGT糖基转移酶将5mC和5hmC氧化为不易被脱氨酶识别的形式;然后利用脱氨酶将未甲基化的胞嘧啶(C)转化为尿嘧啶(U),后续PCR扩增中U变为胸腺嘧啶(T),而甲基化胞嘧啶保持不变,最终通过高通量测序区分甲基化状态。
02 技术优势
(1)低 DNA 损伤:规避BS处理导致的DNA断裂、降解,保留更多原始片段。
(2)低起始量适配:可兼容微量样本,满足珍贵样本(如单细胞、微量活检组织)需求。
03 技术劣势
(1)成本较高:酶试剂成本较高,且操作复杂,全基因组测序成本高于传统BS-seq。
(2)不完全转化风险:少量低甲基化CpG位点可能出现假阳性(转化不完全)。
04 应用场景/适用范围
(1)全基因组甲基化图谱构建:绘制物种、组织或细胞类型的表观遗传图谱。
(2)疾病研究:肿瘤、神经退行性疾病、代谢病等的甲基化标志物筛选与机制研究。
(3)发育与分化研究:胚胎发育、干细胞分化过程中的甲基化动态变化分析。
——适用于需精准区分5mC和5hmC、样本质量有限、追求高数据质量的情况。
六、技术比较
参考文献
[1] Day J J, Sweatt J D. DNA methylation and memory formation[J]. Nature neuroscience, 2010, 13(11): 1319-1323.
[2] Ross J P, van Dijk S, Phang M, et al. Batch-effect detection, correction and characterisation in Illumina HumanMethylation450 and MethylationEPIC BeadChip array data[J]. Clinical Epigenetics, 2022, 14(1): 58.




















 2264
2264

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








