代谢物常用的数据库包括KEGG、HMDB、MONA、METLIN、MassBank、mzCloud、GNPS、LipidBlast、Lipidmaps、NIST、Fiehn等。本期伯小医为大家着重介绍KEGG数据库。KEGG全称为京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes),是整合了基因组、蛋白组、代谢组、化学和系统功能信息的综合数据库,由日本京都大学Kanehisa实验室于1995年推出。KEGG数据库是连接已知分子间相互作用的信息网络,是国际最常用的生物信息数据库之一,也是代谢组学研究中重要的一个工具网站。
01 KEGG主页介绍
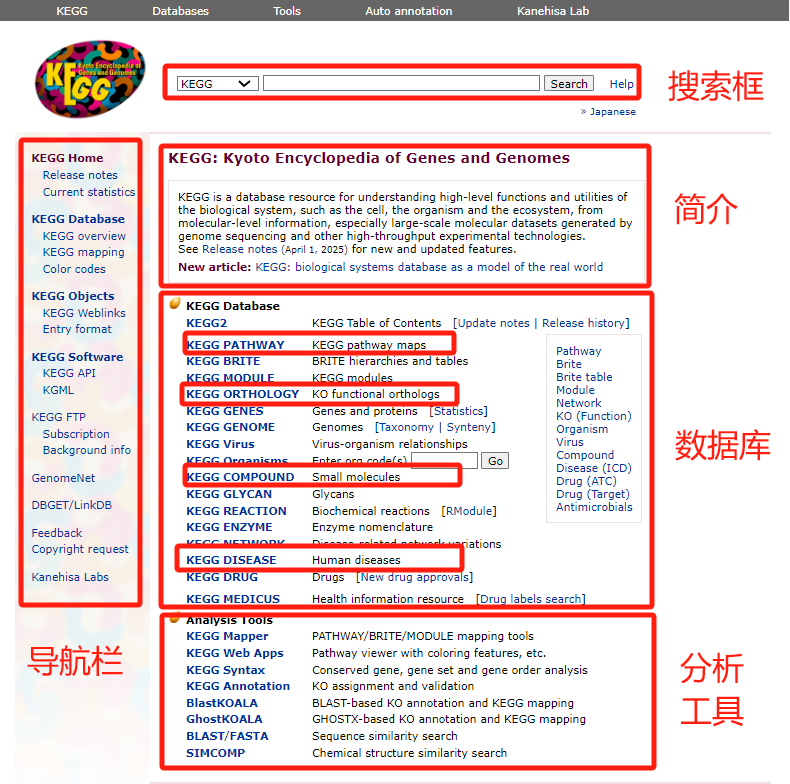
KEGG的官方网址为:https://www.kegg.jp/。进入KEGG官网,它的主页主要由导航栏、搜索框、简介、数据库、分析工具5个部分组成,其中,医学代谢组常用的4个链接分别是KEGG PATHWAY、KEGG ORTHOLOGY、KEGG COMPOUND、KEGG DISEASE,因此接下来,我们对于这四部分内容进行详细介绍。
02 KEGG PATHWAY
KEGG PATHWAY数据库广泛地被用于高通量数据的注释。KEGG PATHWAY是一系列手工绘制的代谢通路的集合,反映了人们目前对于分子间相互作用和反应网络的认识。在点击KEGG PATHWAY之后,我们进入了以下界面。KEGG PATHWAY界面由搜索框、通路图(Pathway Maps)、Pathway Identifiers(通路标识)3个部分组成。搜索框可输入代谢通路的编号或代谢物英文名称进行检索查询。
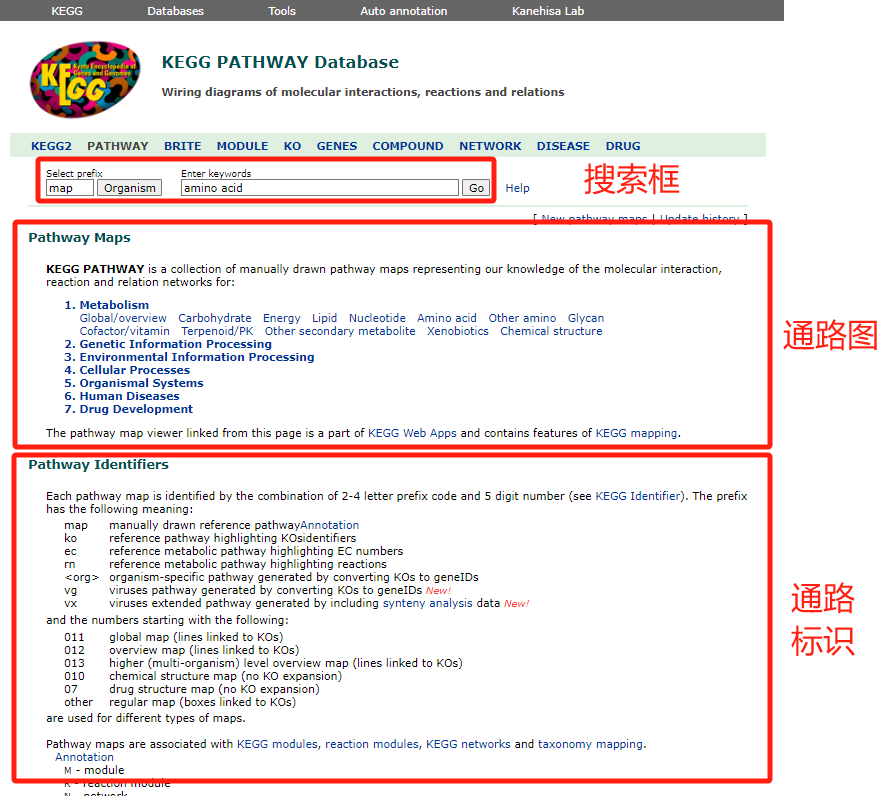
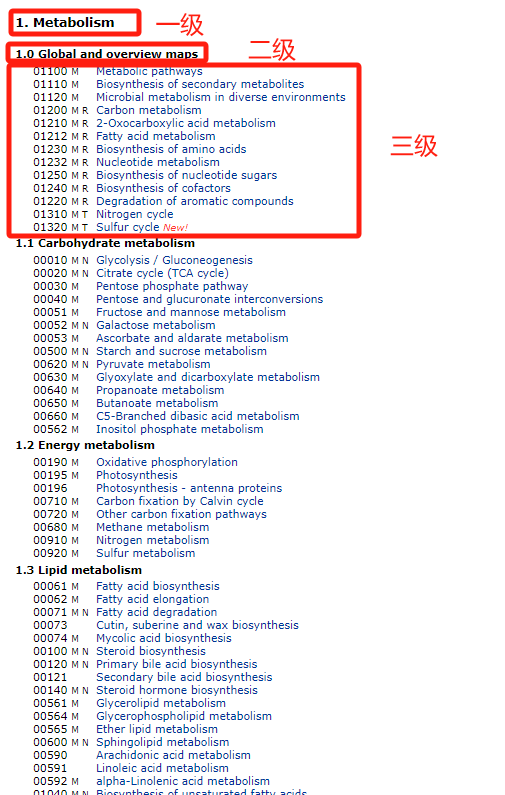
通路图可以分为三个层级,第一级包括:新陈代谢(Metabolism)、遗传信息处理(Genetic Information Processing)、环境信息处理(Environmental Information Processing)、细胞过程(Cellular Process)、生物体系统(Organismal Systems)、人类疾病(Human Diseases)、药物开发(Drug Development))7个部分。一级目录下又有多个二级目录,二级目录下有多个相关的信号通路。比如Metabolism就是一级目录,其下面包括了Global and overview maps、Carbohydrate metabolism、Energy metabolism、Lipid metabolism等十几个二级通路。而二级通路下又会包含若干个3级通路。比如图中的Global and overview maps下面又包含了Metabolic pathways、Biosynthesis of secondary metabolites、Microbial metabolism in diverse environments等13个3级通路。
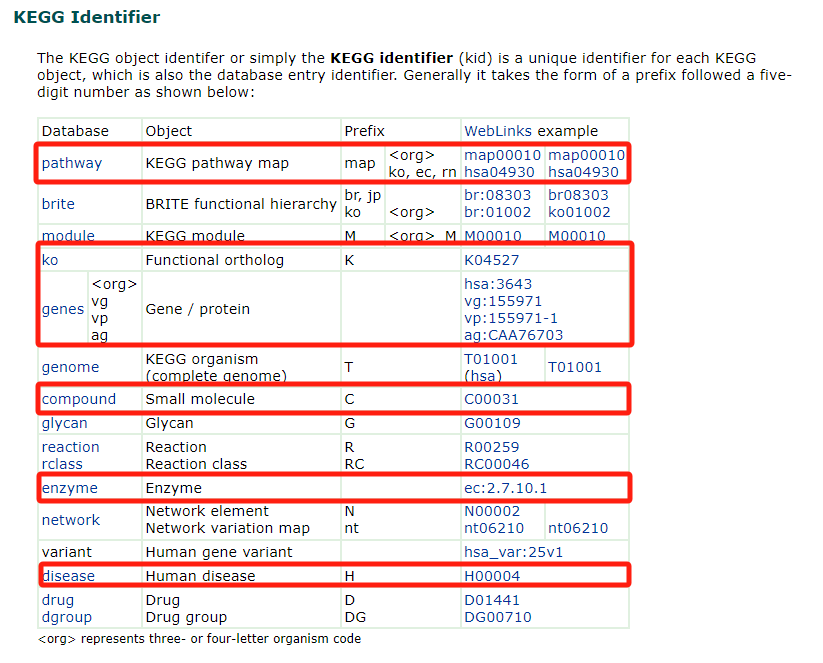
在通路标识中每个通路图由字母的前缀代码和5位数字的组合来标识,具体见下表。在代谢组、多组学、医学研究中,我们使用比较多的是pathway、ko、genes、compound、enzyme、disease这6个。
点击pathway,返回到了KEGG PATHWAY首页,然后在Enter keywords搜索框中输入map编号或者或代谢物英文名称,点击Go,即可进行检索查询。以氨基酸合成通路为案例,氨基酸合成通路为map01230,Enter keywords中输入01230,进入以下界面。该页面包括通路编号(Entry)、缩略通路图(Thumbnail Image)、通路的名称(Name)、通路的描述(Description)、对象(Object)、说明(Legend)。

点击map01230蓝色字链接,进入以下页面(下图为部分截图),该页面主要包括:通路map编号(Entry)、通路名称(name)、通路描述(Description)、通路分类(Class)、通路图(Pathway map)、模块(Module)、通路KO编号(KO pathway)。如果是其它通路还可能有其他数据库链接(Other DBs)、相关文章链接(Reference Authors Title Journal)、相关通路编号(Related pathway)等。
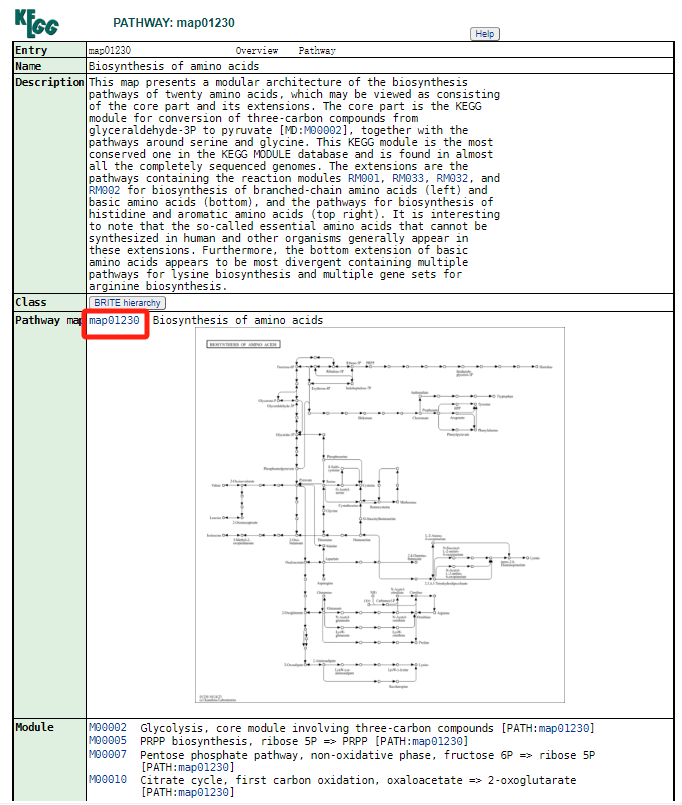
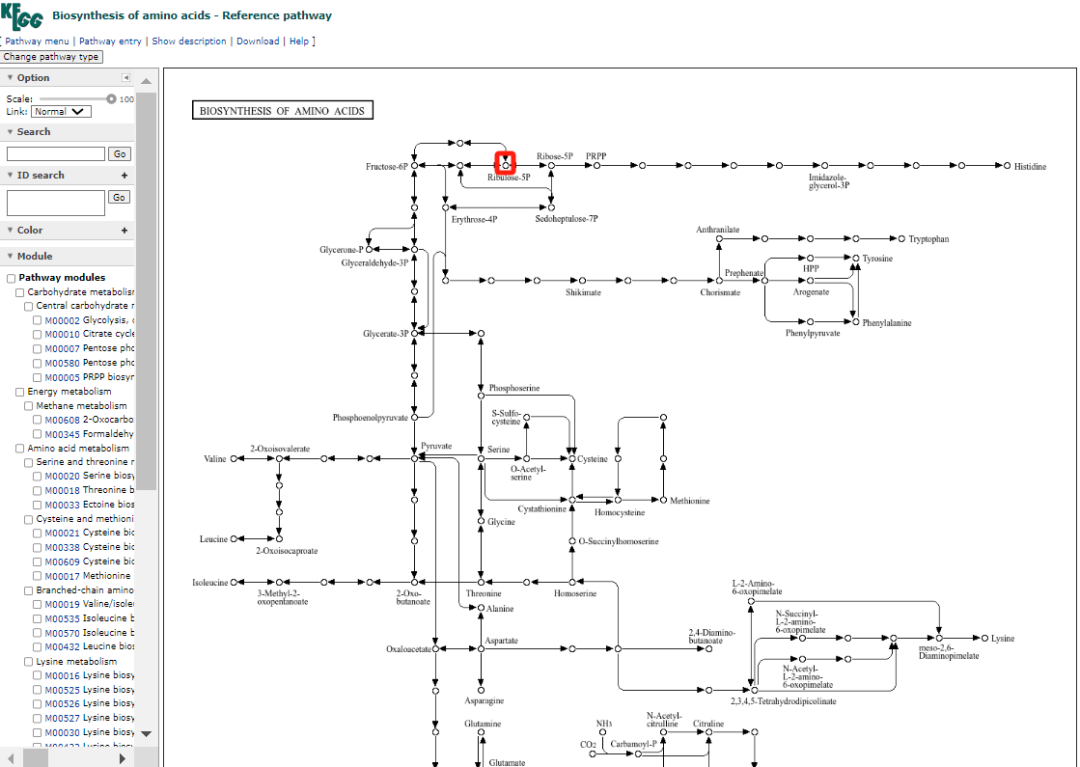
在这些模块中,我们用的最多的是Pathway map,点击map01230链接,进入该通路图。通路图主要由圆圈和箭头构成,圆圈代表的是代谢物,箭头代表代谢物之间的关系。在其它map中,还可能有长方形框,代表的是基因信息。
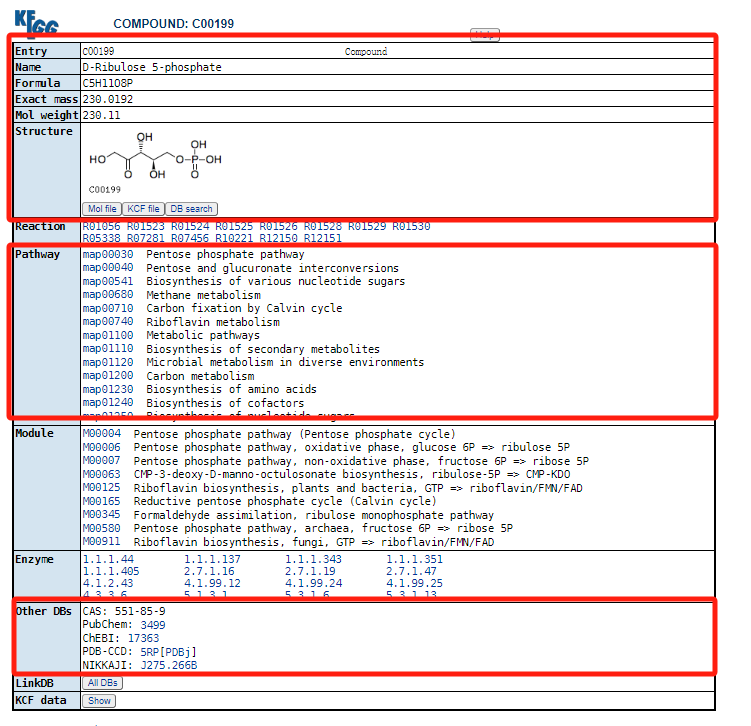
点击关注的圆圈,可进入代谢物的相关信息界面,该页面主要包括:代谢物的编号(Entry)、名称(Name)、分子式(Formula)、精确分子量(Exact mass )、摩尔质量(Mol weight)、分子结构(Structure)、反应(Reaction)、代谢通路(Pathway)、模块(Module)、酶(Enzyme)、其他数据库编号(Other DBs)、其他数据库的链接(LinkDB)、KCF数据(KCF data)。其中常用的模块如下图框出来的部分。
03 KEGG ORTHOLOGY
KEGG ORTHOLOGY(简称 KO)数据库,是一个基于同源基因具有相似功能的假设,对基因进行的分类系统。每个KO代表一个来自不同物种的直系同源基因组,这些基因在同一条通路上有相似的功能。KO帮助研究者在不同物种之间寻找并研究同源基因及其功能。在KO数据库的页面上,能够通过输入K number或者基因名称来查询相关的同源信息,并且在得到的查询结果中,还具备对不同物种进行筛选的功能,以满足更加精准的查询需求。如果我们只是关注某个KO,可以直接通过该数据库查询相关的信息。返回KEGG首页,点击KEGG Database下的KEGG ORTHOLOGY,进入KO界面,然后在for后面搜索框中输入KO编号或者或代谢物英文名称,点击Go,即可进行检索查询。
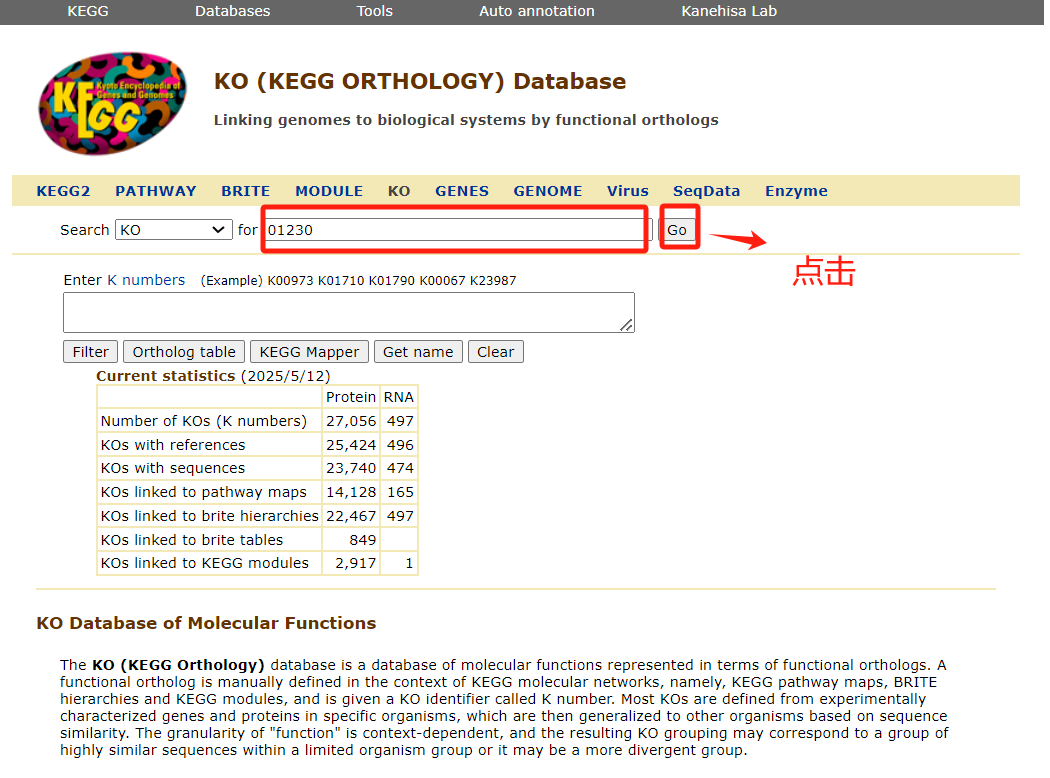
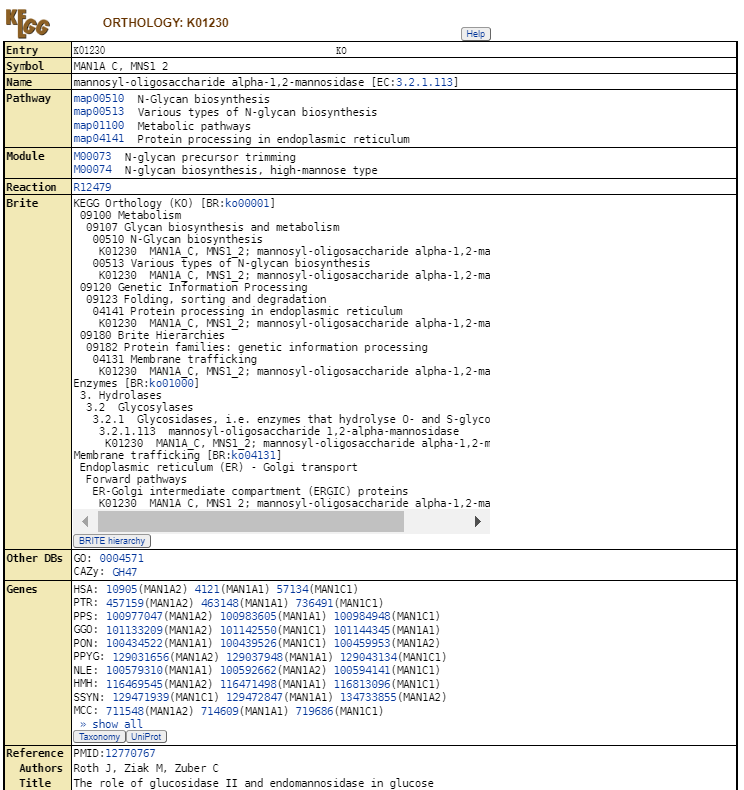
同样以氨基酸合成通路为案例,氨基酸合成通路KO号为01230,搜索框中输入01230,点击Go,然后继续点击KO01230蓝色字链接进入以下界面(下图为部分截图)。该页面主要包括:通路KO编号(Entry)、基因标识符(Symbol)、名称(Name)、通路map编号(Pathway)、模块(Module)、反应(Reaction)、层次分类(Brite)、其他数据库编号(Other DBs)、基因(Genes)、相关文章链接(Reference Authors Title Journal)、其他数据库的链接(LinkDB)。对于该页面中的蓝色字体,都可以点击进入对应的页面。
04 KEGG COMPOUND
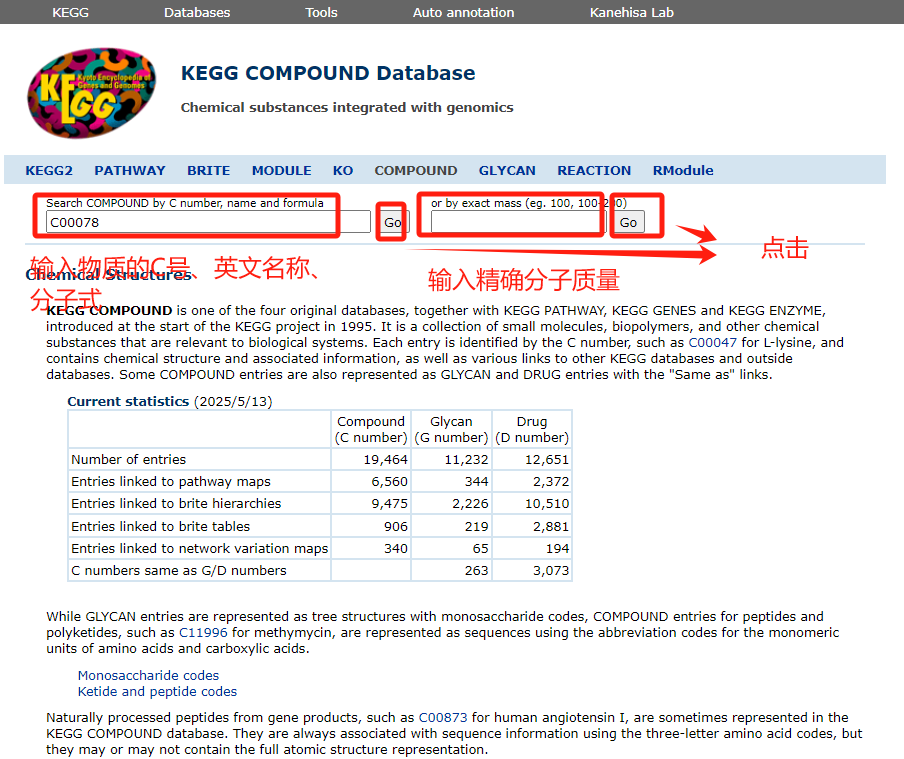
KEGG Compound是一个综合性化合物的数据库,涵盖了丰富多样的化学物质信息。该数据库详细收录了每种化合物的化学结构、分子式、分子量等基本理化性质,同时,还整合了它们在不同生物体中的代谢角色、参与的代谢反应以及与其他化合物的相互作用关系等信息。这使得科研人员能够追踪化合物在复杂代谢通路中的流动和转化,对于解析生物代谢机制、药物研发以及理解生物体的生理过程等有着不可或缺的作用,是生物信息学和系统生物学研究中常用的重要资源之一。如果我们只是关注某个代谢物,可以直接通过该数据库查询相关的信息。返回KEGG首页,点击KEGG Database下的KEGG Compound,进入以下面,然后在搜索框中输入物质的C号、英文名、分子式、精确分子质量,点击Go,即可进行检索查询。
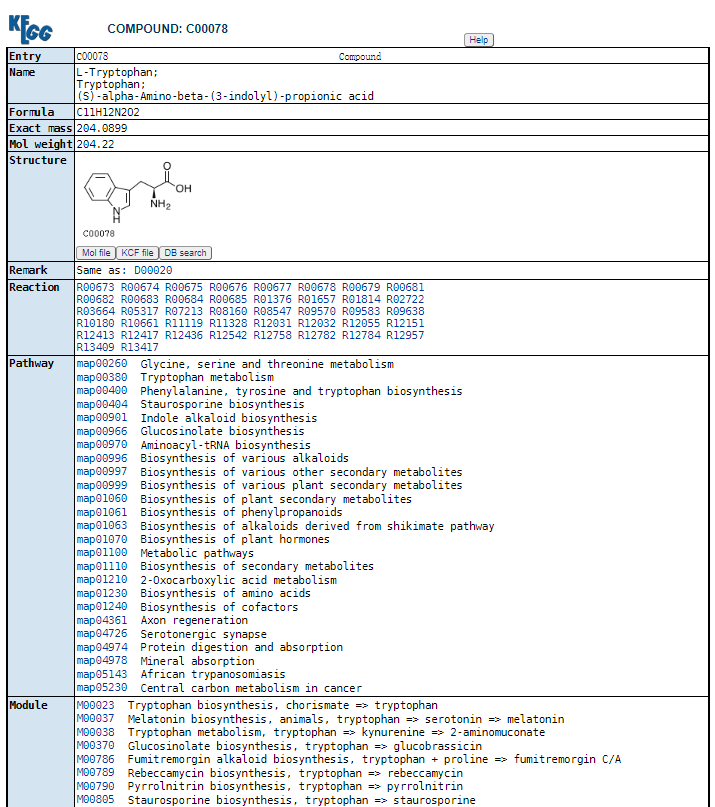
以L-色氨酸为案例,L-色氨酸的C号为C00078,英文名称为L-Tryptophan,分子式为C11H12N2O2,精确分子质量为204.0899。搜索框中输入C00078,点击Go,然后继续点击C00078红色字链接进入以下界面(下图为部分截图)。该页面和前面Pathway Map代谢物的相关信息界面包括的内容一样,这里就不再详细说明。
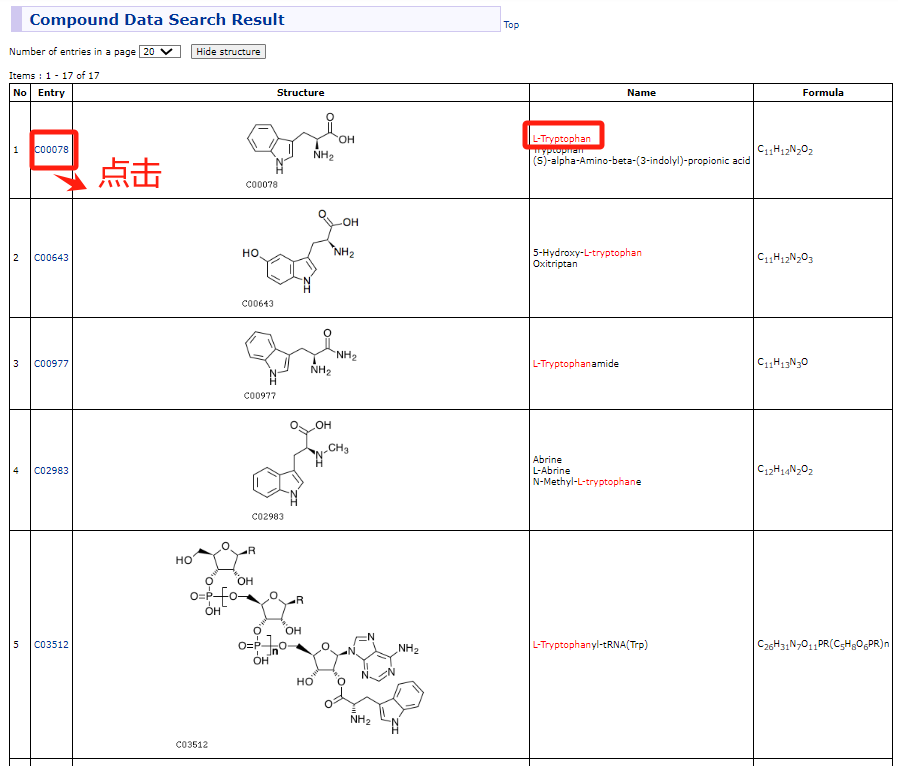
搜索框中输入L-Tryptophan,点击Go,然后进入以下界面。该页面出现多个物质,一般出现的物质包含有相同的英文文字部分,因为一个代谢物可能对应多个英文名,故优先选择名称完全一致的物质进行查询。如该案例中第一个物质英文名完全一致,故选择第一个进行查询,点击C00078链接即可进入代谢物的相关信息界面。
搜索框中输入C11H12N2O2或204.0899,点击Go,发现进入的界面和上面输入L-Tryptophan一样,会出现多个物质。这是因为同分异构体的存在,即物质组成元素和个数一致,但是其排列结构不同。这种现象出现的原因主要是碳原子可以形成四个共价键,导致多种结构异构体的存在。
05 KEGG DISEASE
KEGG DISEASE是一个综合的人类疾病数据库,旨在通过计算机处理来整合疾病信息。KEGG DISEASE提供了疾病相关的基因、分子途径和药物信息,帮助研究人员理解疾病机制。它通过整合基因和环境因素,为研究疾病提供了全面视角。此外,KEGG DISEASE还与KEGG PATHWAY等其他数据库协作,将疾病基因和药物靶点整合到通路图中,以可视化方式呈现疾病相关的分子网络。这种整合有助于发现疾病标志物和开发个性化疗法。如果我们只是关注某种疾病,可以直接通过该数据库查询相关的信息。返回KEGG首页,点击KEGG Database下的KEGG DISEASE,进入以下面,然后在搜索框中输入疾病H编号、名称、描述、类别、途径、基因,也可在KEGG MEDICUS数据库进行查询,点击Go,即可进行检索查询。
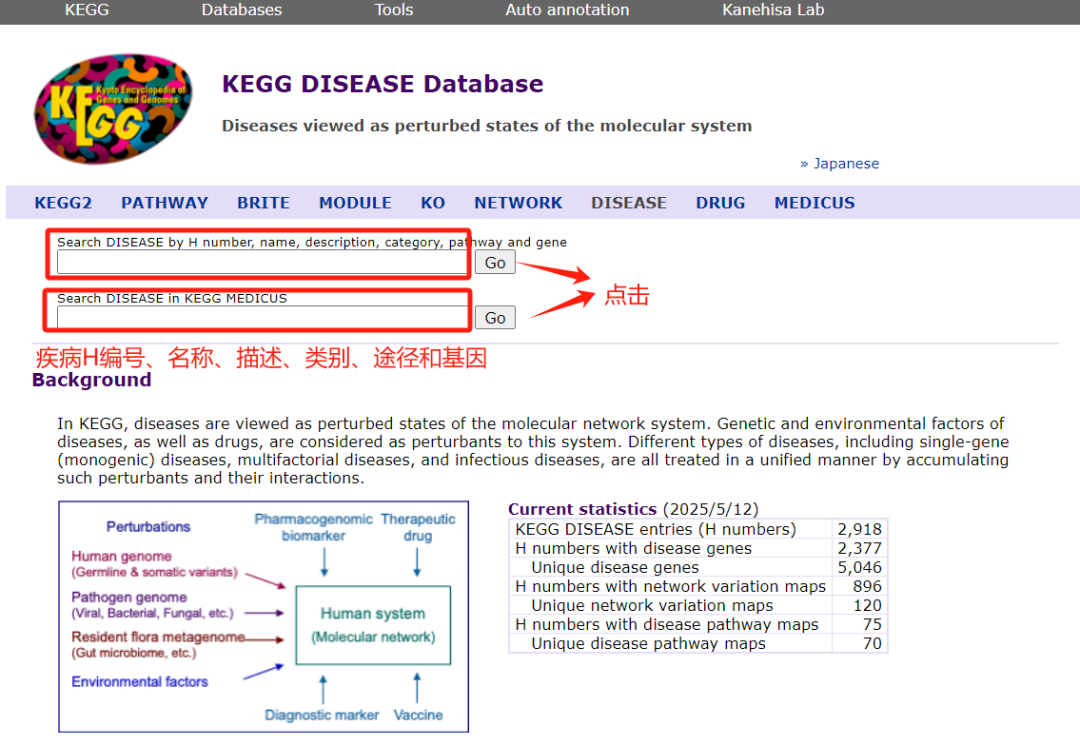
以非小细胞肺癌为案例,非小细胞肺癌的H编号为H00014,英文名称为Non-small cell lung cancer,描述根据文献等资料填写,类别为cancer,途径为hsa05223,相关基因有K05119、K05088等。搜索框中输入H00014,点击Go,然后进入以下界面。该页面包括疾病(DISEASE)、DRUG(药物)、药物分组(DGROUP)和化合物(COMPOUND)4大模块。
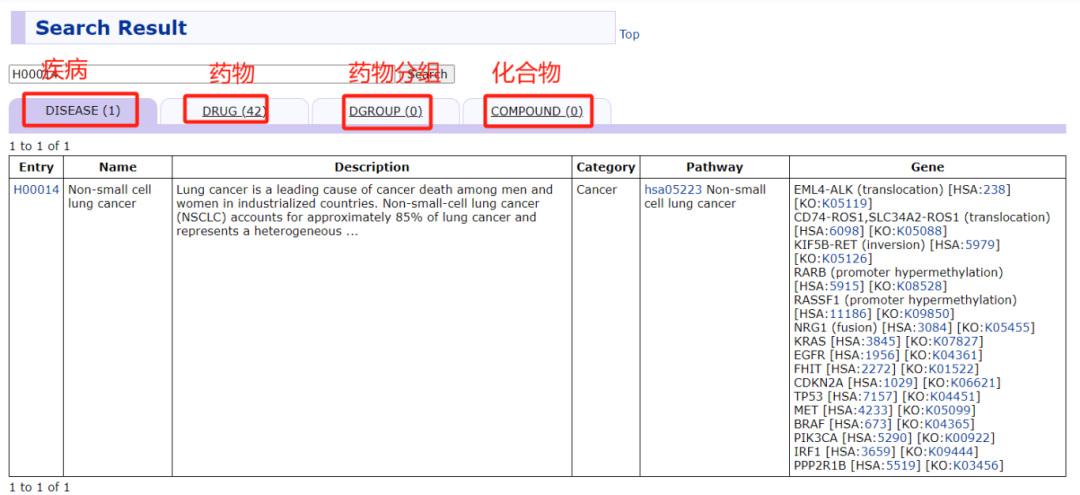
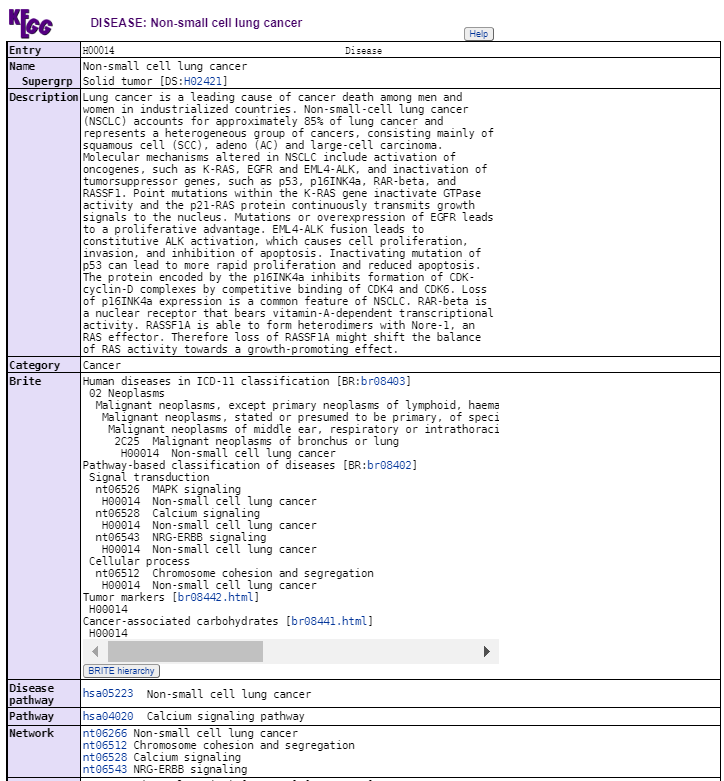
在DISEASE页面,点击H00014蓝色字链接进入以下界面(下图为部分截图)。该页面包括疾病编号(Entry)、名称(Name Supergrp)、描述(Description)、类别(Category)、层次分类(Brite)、疾病通路(Disease pathway)、关系网(Network)、基因(Genes)、药物(Drug)、其他数据库编号(Other DBs)、相关文章链接(Reference Authors Title Journal)、其他数据库的链接(LinkDB)。点击关注的蓝色字链接可进入对应的界面。
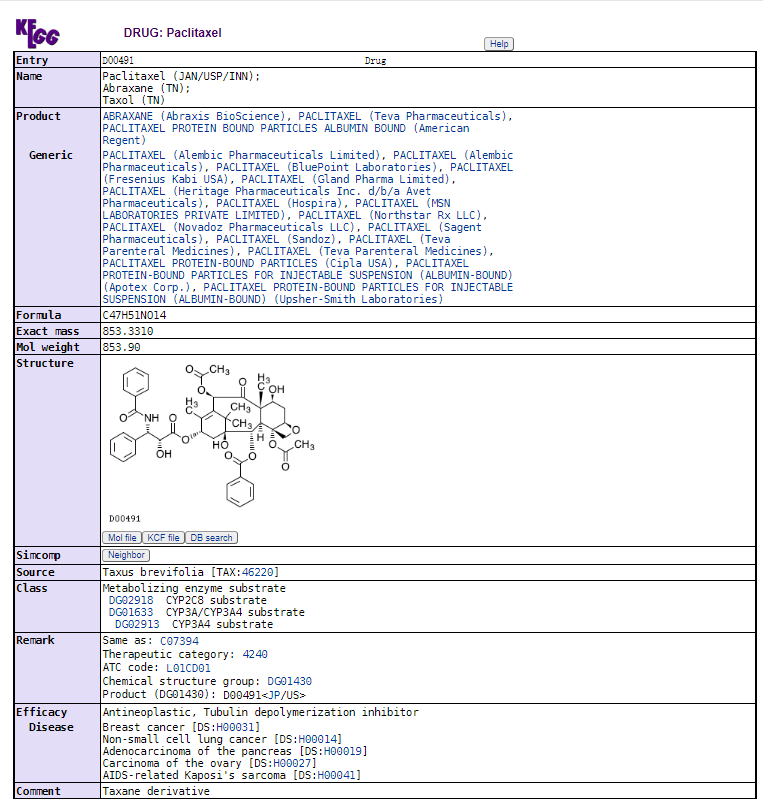
在DRUG页面,该页面出现多个药物,一个药物也对应多种疾病。然后点击关注的药物编号蓝色字链接,如点击第一个D00491即可进入以下界面(下图为部分截图)。该页面包括药物编号(Entry)、名称(Name)、产品信息(Product Generic)、分子式(Formula)、精确分子量(Exact mass)、摩尔质量(Mol weight)、分子结构(Structure)、类似化合物(Simcomp)、来源(Source)、分类(Class)、注释(Remark)、疾病疗效(Efficacy Disease)、说明(Comment)、靶标通路(Target)、代谢(Metabolism)、相互作用(Interaction)、结构通路图(Structure map)、其它通路图(Other map)、层次分类(Brite)、其他数据库编号(Other DBs)、KCF数据(KCF data)。点击关注的蓝色字链接可进入对应的界面。
DGROUP和化合物COMPOUND大多数情况下为0,这里就不再详细描述。




















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








