欢迎关注”生信修炼手册”!
回归我们并不陌生,线性回归和最小二乘法,逻辑回归和最大似然法,这些都是我们耳熟能详的事物,在生物信息学中的应用也比较广泛, 回归中经常出现两类问题,欠拟合和过拟合。
对于欠拟合,简单而言就是我们考虑的少了,一般通过在回归模型中增加自变量或者扩大样本数量来解决;对于过拟合,简单而言就是考虑的太多了,模型过于复杂了,这时候可以对已有的自变量进行筛选,在代价函数中增加惩罚项来限制模型的复杂度,增加的惩罚项我们称之为正则化,正则化常用的有L1正则化和L2正则化,
所谓正则化Regularization, 指的是在回归模型代价函数后面添加一个约束项, 在线性回归模型中,有两种不同的正则化项
1. 所有参数绝对值之和,即L1范数,对应的回归方法叫做Lasso回归
2. 所有参数的平方和,即L2范数,对应的回归方法叫做Ridge回归,岭回归
lasso回归对应的代价函数如下

岭回归对应的代价函数如下
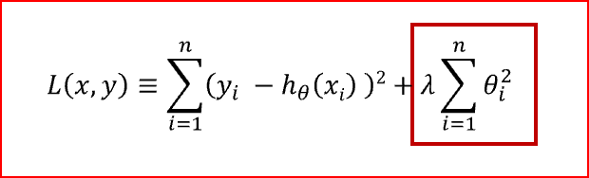
红框标记的就是正则项,需要注意的是,正则项中的回归系数为每个自变量对应的回归系数,不包含回归常数项。
在预后建模的文章中,我们需要针对多个marker基因的表达量汇总形成一个指标,使用该指标来作为最终的maker, 而这个指标在文章中被称之为各种risk score, 比如NAD+基因的预后模型,构建的maker就叫做NPRS, 全称的解释如下
The NAD+ metabolism-related prognostic risk score (NPRS) of each sample was calculated using the formula: NPRS = ΣExp (mRNAί) × Coefficient (mRNAί)
所以各种的预后建模,其实都是lasso回归技术在生物信息学领域的应用。注意观察上述的Lasso回归代价函数,,可以看到有一个未知数λ, 这个参数是一个惩罚项的系数,数值越大,惩罚项对应的影响就越大,我们求解的目标是代价函数值最小,λ = 0时,惩罚项失去意义,代价函数变成了普通的线性回归,而λ过大,惩罚项的影响被放的过大,过小时,惩罚项又失去了原本的意义,所以使用lasso回归,








 文章探讨了预后建模中lasso Cox回归的重要性,解释了正则化的概念,特别是L1(Lasso)和L2(Ridge)正则化在回归模型中的应用。通过引入惩罚项λ来防止过拟合,强调了λ值选择的策略,如10折交叉验证,并推荐了R包glmnet进行模型构建。文中还详细介绍了cox回归的正则化处理,并展示了交叉验证过程中的评价指标,如deviance和C-index。
文章探讨了预后建模中lasso Cox回归的重要性,解释了正则化的概念,特别是L1(Lasso)和L2(Ridge)正则化在回归模型中的应用。通过引入惩罚项λ来防止过拟合,强调了λ值选择的策略,如10折交叉验证,并推荐了R包glmnet进行模型构建。文中还详细介绍了cox回归的正则化处理,并展示了交叉验证过程中的评价指标,如deviance和C-index。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 2609
2609

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








