01GATK
一个专注于变异发现的基因组分析工具包。GATK是目前用于DNA和RNA数据中SNP和INDEL的行业标准。
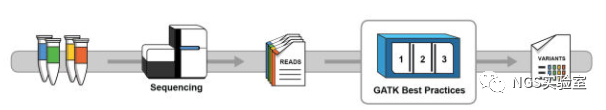
GATK目前比较通行的有两个版本,一个是最新的GATK4.0以上的版本,一个是GATK3.8版本。
两个版本结果及其接近,4.0版本相对与3.8版本的计算效率有一定程度的提高。
两个软件均由java开发完成,可以适用于linux或MacOS系统中。
03Best Practise
用GATK寻找SNP和Indel,有一个标准的分析流程叫做GATK Best Practise主要包括以下几个步骤:
数据预处理:对从测序仪下机后的数据进行质控,去除低质量的reads,将过滤后的reads比对到参考基因组上,产生BAM格式的比对文件。
寻找变异:进行variant calling,寻找SNP和Indel,将比对数据存储在VCF格式的文件中。
使用寻找出的变异位点进行后续的分析。
05变异检测流程01
本文以原始数据质控开始,生成用于bam文件
#原始数据质控fastp -i sample.1.fastq.gz -o sample.clean.1.fastq.gz -I sample.2.fastq.gz -O sample.clean.2.fastq.gz#参考基因组配置bwa index ref.fasamtools faidx ref.fasamtools dict ref.fa -O ref.dict#参考基因组比对#-R 参数及其重要:ID 编号、SM 样品名称、LB 文库类型、PL 测序平台bwa mem mem -t 8 -M -Y -R '@RG\tID:reseq01\tSM:sample\tLB:WES\tPL:Illumina' ref.fa sample.clean.1.fastq.gz sample.clean.2.fastq.gz|samtools view -Sb - > sample.bam#bam文件修饰(排序-去冗余)samtools sort -@ 8 -o sample.sorted.bam sample.bam && samtools index sample.sorted.bamsamtools markdup -@ 8 --write-index sample.sorted.bam sample.markdup.bam至此,完成了原始数据质控、比对、排序的相关过程,欲知后事如何,且听下回分解。







 本文介绍了GATK,一个用于DNA和RNA数据中SNP和INDEL分析的标准工具。GATK有GATK4.0和GATK3.8两个版本,4.0在计算效率上有提升。它遵循Java开发,适用于Linux和MacOS。GATK的最佳实践包括数据预处理、变异检测和后续分析。内容涵盖了从原始数据质控到比对生成BAM文件的过程。
本文介绍了GATK,一个用于DNA和RNA数据中SNP和INDEL分析的标准工具。GATK有GATK4.0和GATK3.8两个版本,4.0在计算效率上有提升。它遵循Java开发,适用于Linux和MacOS。GATK的最佳实践包括数据预处理、变异检测和后续分析。内容涵盖了从原始数据质控到比对生成BAM文件的过程。
















 732
732

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








