SPM 中 fMRI 的数据处理只接受多个单独的nii文件,但不知道为什么我的 SPM 中的 DICOM import 无法实现fMRI数据的转换(报错 No "Image Plane" information)。
dcm2niix 只支持多个 dicom 文件转换为一个 nii 文件(也可能是我没找到方法)。
后来发现如果将每个 dicom 文件分别放入单独的文件夹,再使用 dcm2niix 进行转换,就能实现每个 dicom 单独转换为 nii。于是写了 Python 代码实现这一过程。
文件目录如下所示:
data
├─function
│ ├─sub1
│ │ ├─dicom
│ │ └─output
│ └─sub2
└─func_out
├─sub1
└─sub2
data 为保存所有数据的文件夹,function 为原始数据存放的文件夹,其中 dicom 存放待转换的dicom 数据,output 文件夹、func_out 文件夹目前为空。
第一个 Python 代码的目标是将放在同一个文件夹 dicom 下的多个dicom数据分别移入 output 文件夹中的子文件夹。其中 'files_dir', 'new_folders_dir', 'dcm_total' 需要根据存放的位置与dicom的总数修改。'file_name'需要根据自己 dicom 文件的命名规则更改。
import os
files_dir = 'D:\\MRI_data\\DATA_fMRI\\data\\function\\sub2\\dicom' # 原始数据的位置
new_folders_dir = 'D:\\MRI_data\\DATA_fMRI\\data\\function\\sub2\\output' # 输出的位置
# 创建新文件夹
os.makedirs(new_folders_dir, exist_ok=True)
dcm_total = 640 # files_dir中dcm文件的个数
for i in range(1, dcm_total + 1):
# 创建新文件夹
folder_name = os.path.join(new_folders_dir, str(i))
os.makedirs(folder_name, exist_ok=True)
# 移动文件
file_name = str(i).zfill(8) + '.dcm'
src = os.path.join(files_dir, file_name)
dst = os.path.join(folder_name, file_name)
if os.path.exists(src):
os.rename(src, dst)
else:
print(f"文件 {file_name} 不存在")
print("文件移动完成")
运行的结果为:dicom文件夹为空,output文件夹如下:
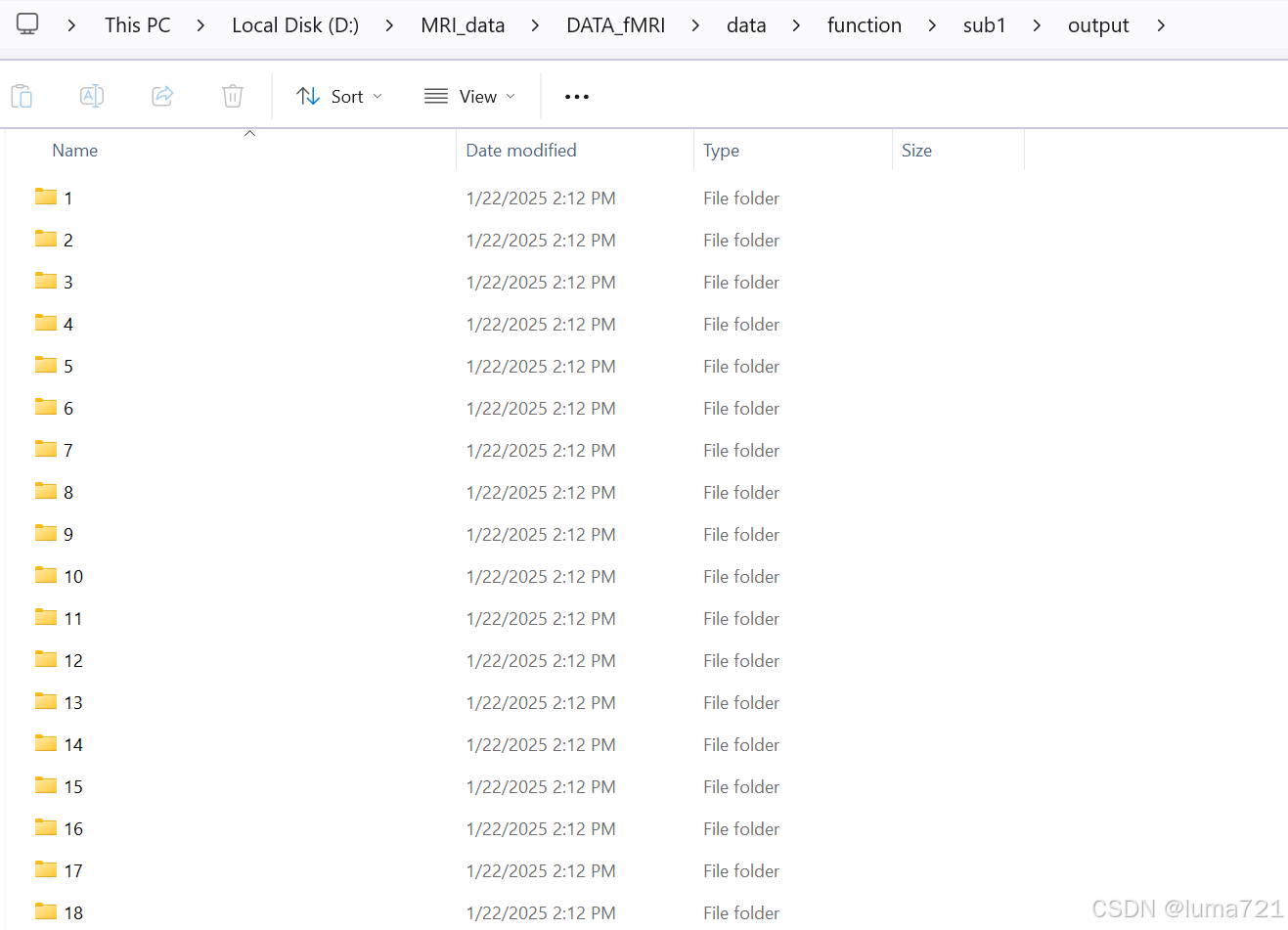
其中每个文件夹内是移入的dicom文件。
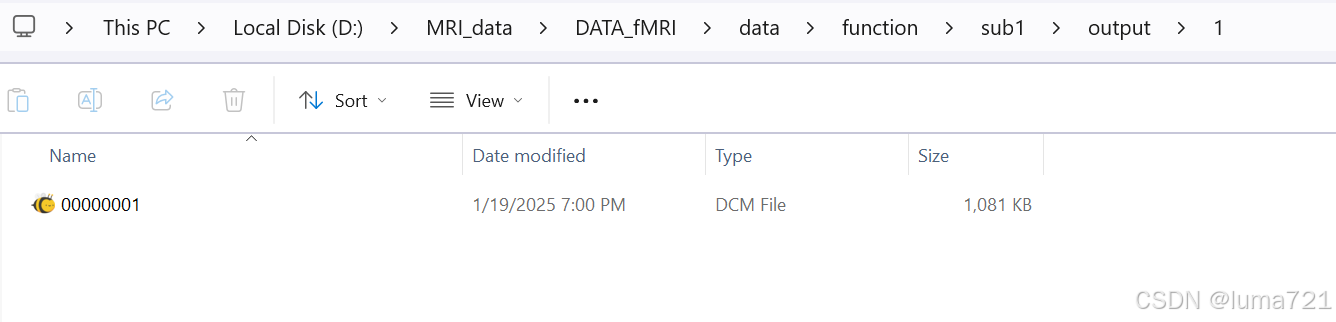
以上代码运行成功后,可以运行第二个代码。
import os
d2n = 'D:\\Tools\\MRIcron\\Resources\\dcm2niix.exe -f ' # dcm2niix.exe 所在的位置
para = ' -i y -l y -p y -x y -v y -z n -o ' # 参数设置
output_path = "D:\\MRI_data\\DATA_fMRI\\data\\func_out\\sub2"
filerootpath = 'D:\\MRI_data\\DATA_fMRI\\data\\function\\sub2\\output' # 上一段代码中的new_folders_dir变量
dcm_total = 640 # files_dir中dcm文件的个数
for i in range(1, dcm_total + 1):
outputname = str(i).zfill(8)
filepath = filerootpath + '\\' + str(i) + '\\' + str(i).zfill(8) + '.dcm'
cmd = d2n + outputname + para +'\"'+ output_path +'\" ' + '\"'+ filepath +'\"' #其中的双引号不可少
# print(cmd)
os.system(cmd)
运行结果为:
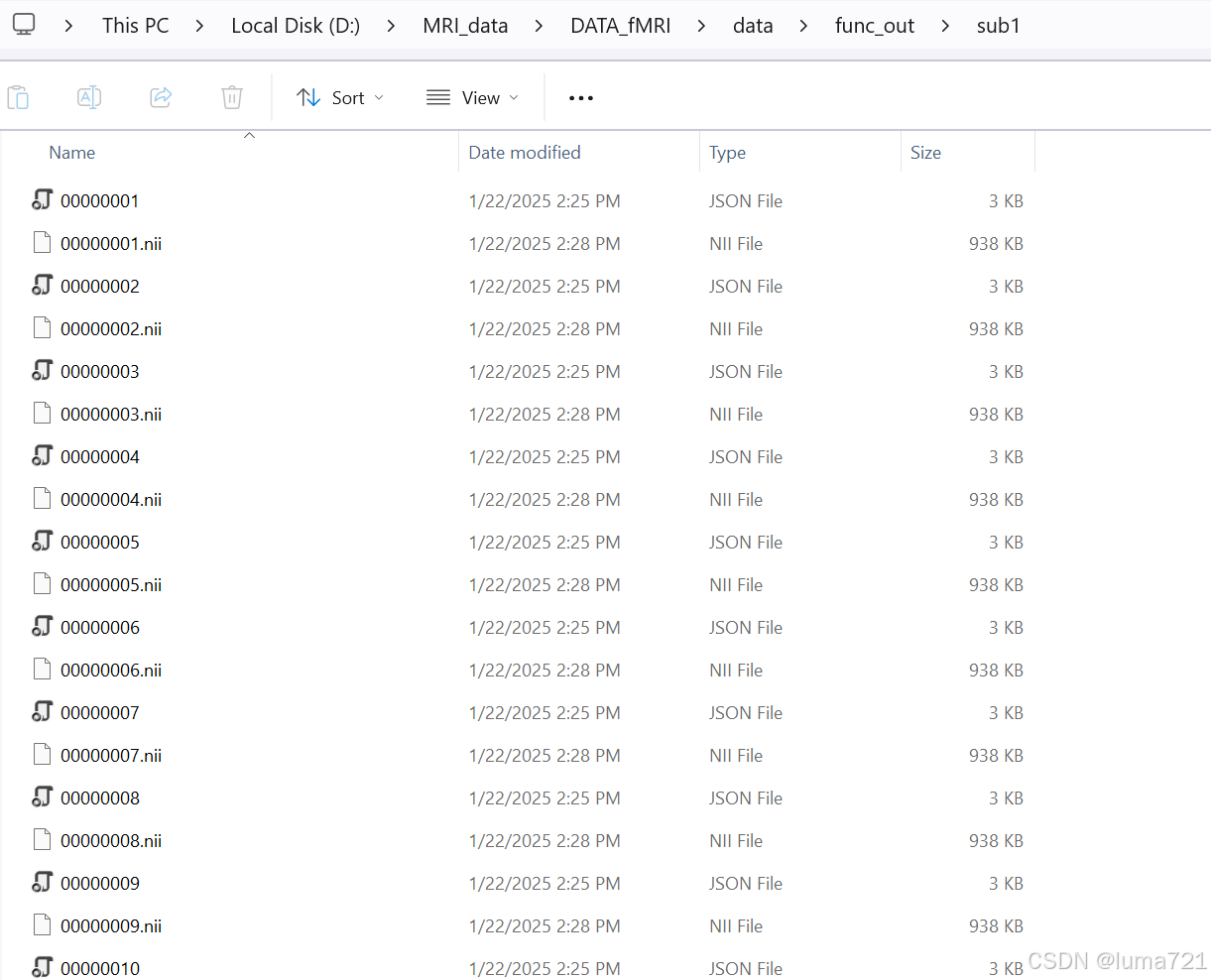




















 1814
1814

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








