文章目录
IGV——基因组可视化
Integrative Genomics Viewer (IGV) 是一种高性能、易于使用的交互式工具,用于对基因组数据进行可视化探索。IGV 支持所有常见类型的基因组数据和metadata的灵活集成,且支持各种各式的大型数据集的可视化,包括二代测序,芯片测序数据和基因组数据等。例如,查看转录组数据不同区域的Reads丰度,不同样本的基因表达差异,查找SNP位点,查看甲基化,查看基因结构信息。
官网:https://igv.org/doc/desktop/#QuickStart/
安装
IGV 可以直接下载到带桌面图像的 MAC,Linux 或 Windows 系统的本地电脑直接使用,一般情况下本地电脑即可通过简单图形界面完成 IGV 可视化,如果数据量过大,可以考虑在服务器安装,在本地查看图形界面。
下载地址:https://igv.org/doc/desktop/#DownloadPage/
方式一:服务器(Linux)安装,本地使用
(1)服务器安装与配置
考虑到大部分生信数据分析都基于服务器完成且文件较大下载到本地可能存在空间不足的问题,此处可以直接在服务器上完成安装,再配合 Jupyter 使用,建立 FTP 来通过 url 访问,即可以把可视化图像转到本地电脑屏幕。
wget https://data.broadinstitute.org/igv/projects/downloads/2.18/IGV_Linux_2.18.2_WithJava.zip
unzip IGV_Linux_2.18.2_WithJava.zip
bash igv.sh # 可能会报错 No X11 DISPLAY variable was set,但是不影响
# 使用本地屏幕可视化,需要搭配 Java11 使用
wget https://download.java.net/java/GA/jdk11/13/GPL/openjdk-11.0.1_linux-x64_bin.tar.gz
tar -xzvf openjdk-11.0.1_linux-x64_bin.tar.gz
# 设置路径
vi ~/.bashrc
# 最后一行添加以下两条命令 (根据你实际的路径)
export PATH=~/tool/jdk11/jdk-11.0.1/bin:$PATH
alias igv='bash ~/tool/igv/IGV_Linux_2.18.2/igv.sh' # 直接使用 igv 即可启用
注意:如果使用 conda 安装 igv 或者 igvtools 会自动搭配安装 java8,即使已经安装 java11 也会被强制修改,而 java8 无法启用 command line 模式。
(2)本地可视化配置(X11 安装)
服务器一般只有命令行无法完成可视化,因此需要在本地搭配可视化屏幕,从而将服务器的显示连到本地电脑上,不同的操作系统有对应不同的视窗系统软件,mac 可以使用 Xquartz,windows 系统可以另外查找和安装视窗系统,最后在服务器中用 igv 启动即可。
- mac 版本:
安装 XQuartz:https://www.xquartz.org/
配置 XQuartz:在 terminal 里修改 /etc/ssh/sshd_config 文件,将 X11Forwarding 参数改为 yes 保存退出后重启电脑激活 service ssh restart 。
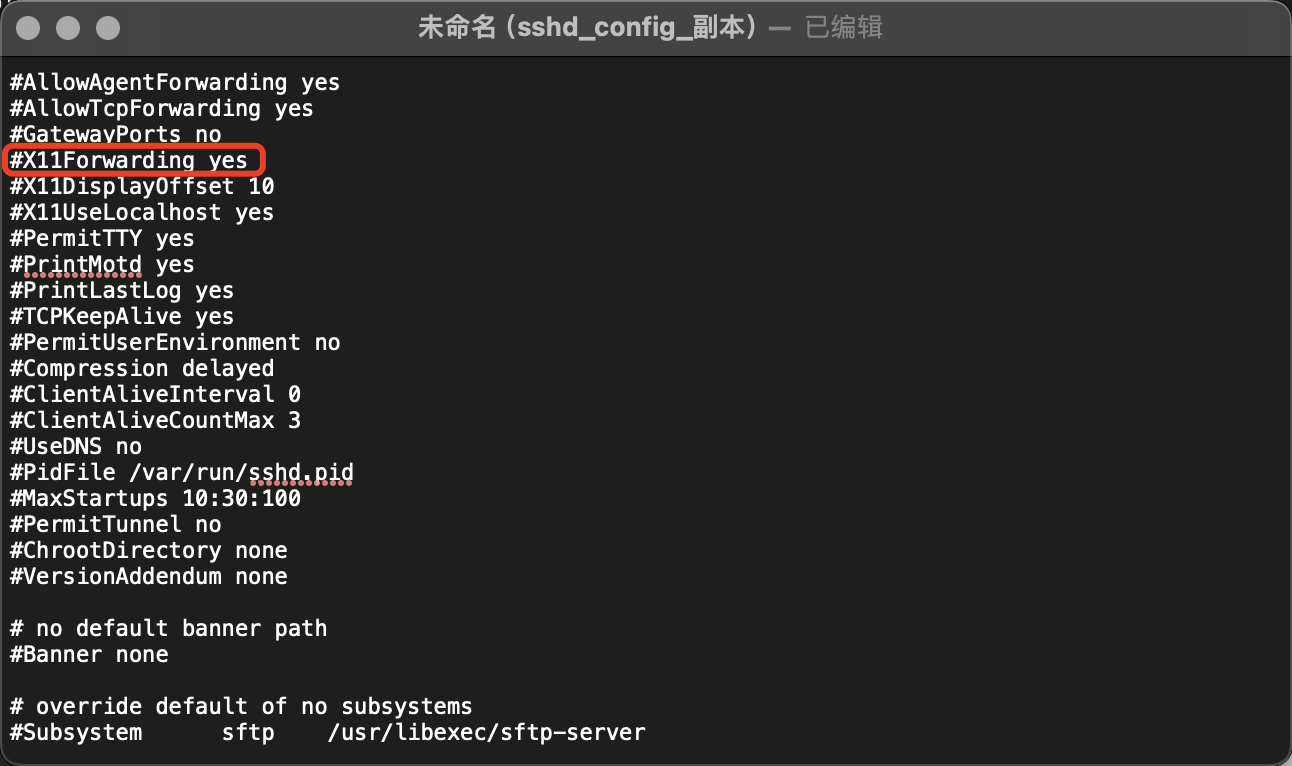
如果没有修改权限,可以运行 finder -> 前往 -> 前往文件夹 -> 输入 /private/etc/ssh/ -> 编辑另存 -> 覆盖原来文件 -> 重新启动
打开 XQuartz 后需要进行一些基本配置: 设置 -> 安全性





 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 3318
3318

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








