文章目录
ClusterGVis——时间序列聚类可视化
ClusterGVis 提供了时间序列的 RNA-seq 数据的模糊 c-means 算法和 kmeans 算法聚类的可视化,还可以实现对 WGCNA 输出的可视化,同时可以使用 clusterProfiler 的 enrichCluster 完成每个 cluster 的富集分析,直接生成发表级图形。
**官网:**https://github.com/junjunlab/ClusterGVis
安装
可以直接在 r 中完成安装,重新安装可以获取新功能。
# Note: please update your ComplexHeatmap to the latest version!
# install.packages("devtools")
devtools::install_github("junjunlab/ClusterGVis")
如果出现某些依赖项无法安装的情况,可以使用如下相应命令使用 conda 完成安装,如果仍然出现错误可以尝试创建新环境运行。
# 此处可能需要创建一个新环境才安装成功
# conda create -n demo
# conda activate demo
conda install -c bioconda r-monocle3 r-seurat
conda install conda-forge::r-magick
conda install bioconductor-org.mm.eg.db
conda install bioconductor-clusterprofiler
conda install -c conda-forge r-terra
conda install -c conda-forge r-devtools
在实际的安装中,可能会出现 jjAnno 包(https://github.com/junjunlab/jjAnno)无法安装的情况,一般情况下按上述命令完成对应依赖项安装后在 r 中重新运行如下命令即可成功安装。
devtools::install_github("junjunlab/jjAnno")
使用
1. 输入
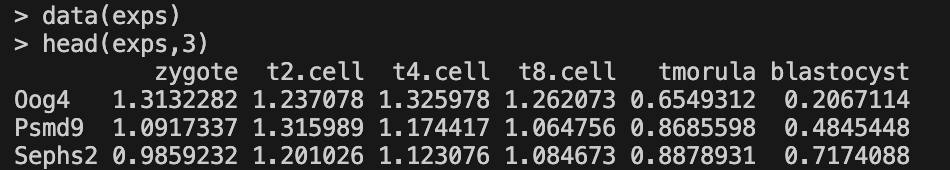
输入数据为标准化的 tpm/fpkm/rpkm 表达矩阵,结合源码和 Mfuzz 包(http://193.136.227.155/sysbiolab/mfuzz/)的文档,此处在 cluster.method == "mfuzz" 时也可以使用其他方法标准化后的数据,如 edgeR/DESeq2。

# 载入示例数据
library(ClusterGVis)
data(exps)





 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 1459
1459










