背景介绍
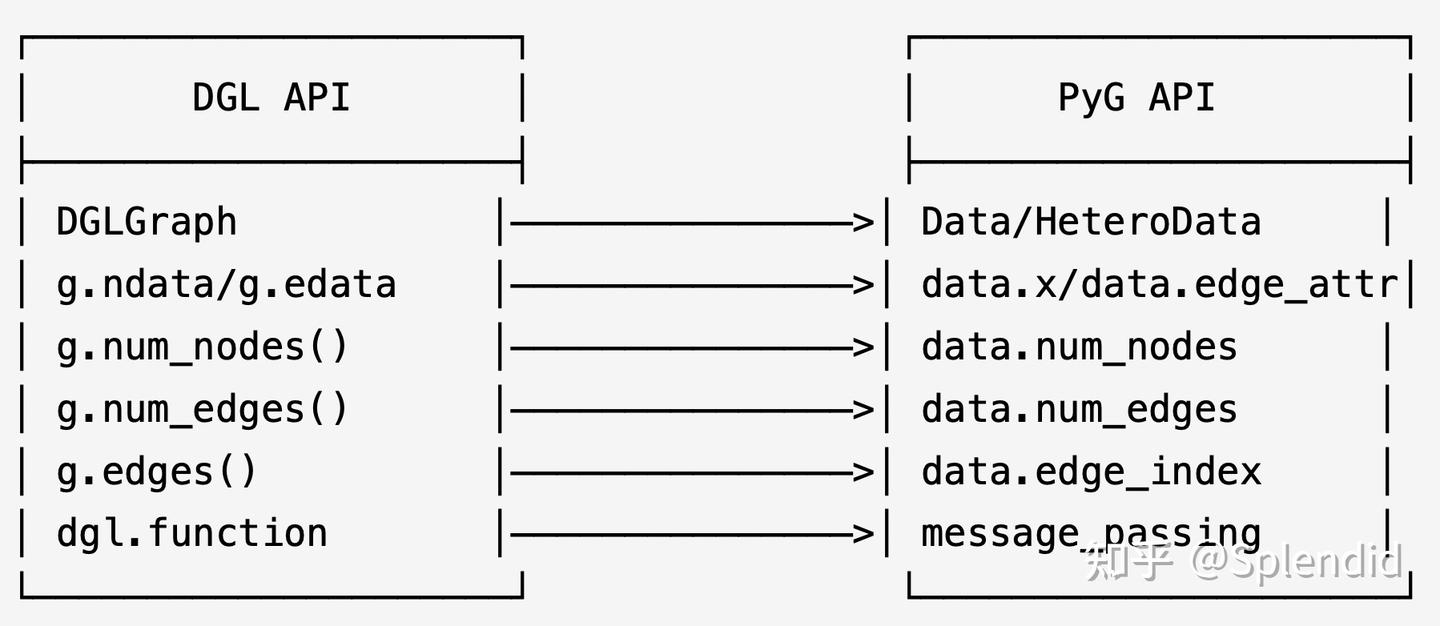
DGL (Deep Graph Learning) 和 PyG (Pytorch Geometric) 是两个主流的图神经网络库,它们在API设计和底层实现上有一定差异,在不同场景下,研究人员会使用不同的依赖库,昇腾NPU对PyG图机器学习库的支持亲和度更高,因此有些时候需要做DGL接口的PyG替换。
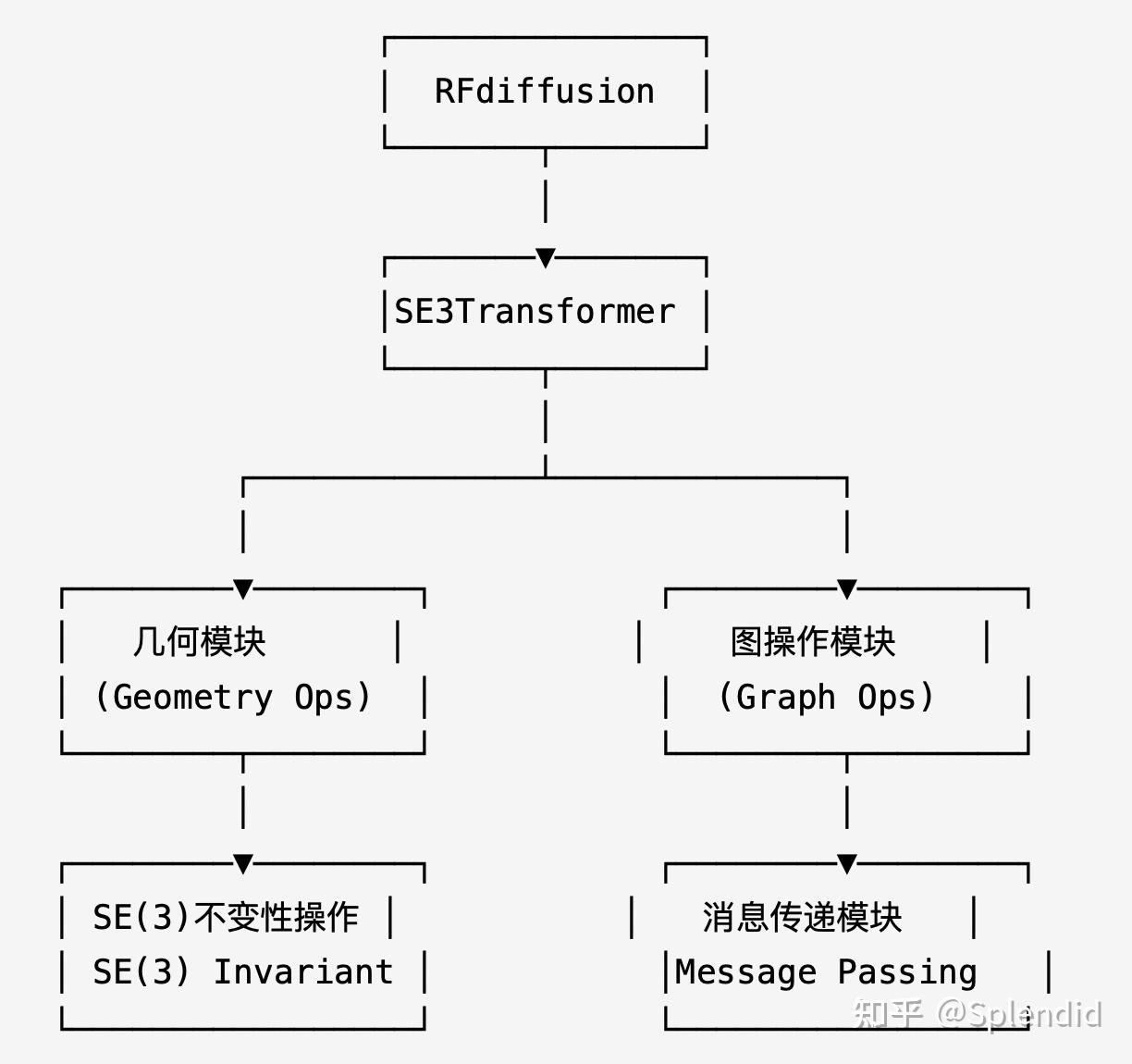
SE3Transformer在RFdiffusion蛋白质设计模型中(GitHub - RosettaCommons/RFdiffusion: Code for running RFdiffusion)作为核心组件,负责处理蛋白质结构的几何信息。其架构基于图神经网络,通过SE(3)等变性实现对三维旋转和平移的不变性特征提取。本系列以RFDiffusion模型中的SE3Transformer为例,讲解如何将DGL中的接口替换为PyG实现。在本文中,主要展示图构建结构的替换。
DGL图构建接口的PyG替换(make_full_graph和make_topk_graph)
make_full_graph 函数
位置:
- rfdiffusion/util_module.py
输入:
- xyz: 蛋白质骨架坐标,形状为(B, L, 3)或(B, L, 3, 3)
- pair: 成对特征,形状为(B, L, L, E)
- idx:残基索引
输出:
- G : DGL图
- edge_feats:边特征
调用DGL函数:
- dgl.graph:创建图结构
数学逻辑:
- 提取氨基酸相对位置
- 构建完全连接图
- 设置边特征和节点特征
PyG实现代码:
def make_full_graph(xyz, pair, idx, top_k=64, kmin=9):
B, L = xyz.shape[:2]
device = xyz.device
# 确保xyz形状正确
if xyz.dim() > 3:
xyz_flat = xyz[:,:,1] if xyz.shape[2] == 3 else xyz.reshape(B, L, 3)
else:
xyz_flat = xyz
# 计算序列分离
sep = idx[:,None,:] - idx[:,:,None]
b,i,j = torch.where(sep.abs() > 0)
# 构建PyG图所需的边索引
src = b*L+i
tgt = b*L+j
# 创建图对象
G = graph((src, tgt), num_nodes=B*L).to(device)
# 计算相对位置
rel_pos = xyz_flat[b,j,:] - xyz_flat[b,i,:]
if rel_pos.dim() > 2 and rel_pos.shape[-1] == 3:
rel_pos = rel_pos.reshape(-1, 3)
G.edata['rel_pos'] = rel_pos.detach()
# 处理边特征
edge_feats = pair[b,i,j]
if edge_feats.dim() == 1:
edge_feats = edge_feats.unsqueeze(-1)
if edge_feats.dim() == 2:
edge_feats = edge_feats.unsqueeze(-1)
# 归一化特征减少实现差异
edge_feats = torch.tanh(edge_feats / 10.0) * 10.0
return G, edge_featsmake_topk_graph
位置:
- rfdiffusion/util_module.py
输入和输出:
- 与 make_full_graph 类似,但构建k近邻图而非完全图
调用DGL函数:
- dgl.graph:创建图结构
数学逻辑:
1. 计算氨基酸之间距离
2. 选择top-k最近邻居
3. 确保每个节点至少有kmin个邻居
优化方案:
- 使用PyG的knn_graph函数简化实现函数简化实现
- 利用PyG的批处理机制处理多图




















 832
832

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








