本文主要介绍PeptideBERT模型是什么、PeptideBERT算法的基本原理、以及如何在昇腾设备上进行部署。
1. PeptideBERT介绍
PeptideBERT(Peptide Bidirectional Encoder Representations from Transforme- rs)是一种基于transformer架构,专门用于预测肽的关键性质的蛋白质语言模型,如溶血性(hemolysis)、溶解性(solubility)和抗非特异性吸附性(non-fouling)。预训练模型为ProtBERT,有12个注意力头和12个隐藏层,通过针对三个下游任务对预训练模型进行微调,PeptideBERT模型通过深度学习模型快速预测肽的功能,大幅缩短研发周期,降低实验成本,在预测溶血方面可以达到先进水平,对于确定肽诱导红细胞溶解的潜力以及非污染特性至关重要。PeptideBERT这篇工作通过前沿AI技术突破科学研究的效率瓶颈,其意义不仅在于提升肽研究的智能化水平,更在于为生命科学其他领域(如RNA、代谢物分析)提供了可迁移的方法框架,推动跨学科的下一代科学发现范式。
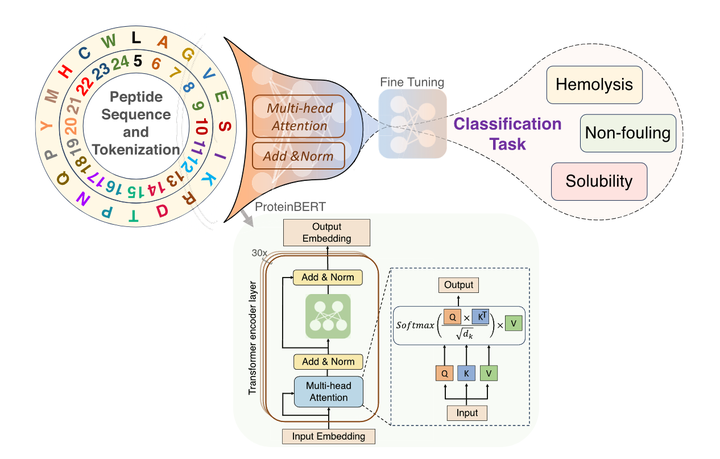
图1. PeptideBERT的模型架构。
肽序列经过标记化处理,随后通过 ProtBERT 进行处理。接着添加一个多层感知机(MLP)的分类头用于微调过程。该模型分别在溶血性、防污性和溶解性三个不同的分类下游任务上进行单独训练。
2. 算法原理
(1) 输入表示
PeptideBERT的输入为肽的氨基酸序列片段,其输入表示由三部分构成,以适配Transformer架构
-
词嵌入(Token Embeddings): 将肽序列中的氨基酸残基(如A、C、L等单字母表示)映射为高维向量。
-
物理化学属性编码 : 除字母本身,氨基酸的属性(如疏水性、电荷、分子量)也被编码为向量,与字符嵌入拼接。
-
位置编码(Positional Encoding): 通过可学习的绝对位置嵌入向序列中每个位置注入顺序信息,确保模型感知残基的一维排列结构。对于长度固定的输入(如设定最大长度50),超出部分截断,不足部分填充[PAD]标
(2) 注意力机制
-
PeptideBERT采用多头自注意力(Multi-Head Attention)计算序列内任意两残基的相互作用,而Transformer 架构的核心就是注意力机制,它能够捕捉序列中任意位置之间的依赖关系。具体来说,注意力机制通过计算查询(Query)、键(Key)和值(Value)之间的点积来确定权重,并通过 Softmax 函数进行归一化,且总和为1。
-
查询(Q)、键(K)、值(V)矩阵 对输入向量进行线性投影:
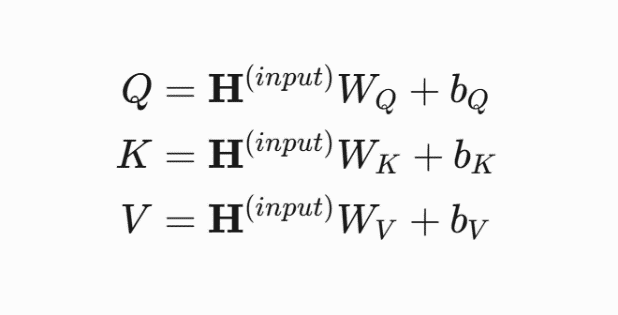
-
a. 单头注意力机制
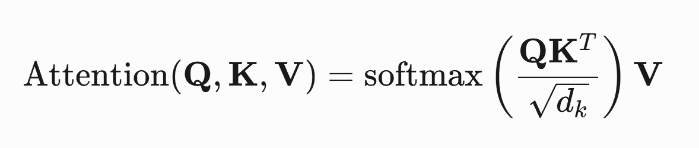
-
b. 多头注意力机制(将输入拆分为h个头,每个头单独计算后拼接)
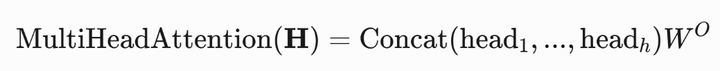
(3) 预训练目标
a. 掩码语言建模(Masked Language Modeling,MLM):随机遮蔽部分氨基酸(15%),模型需要根据上下文预测被遮蔽的位置。
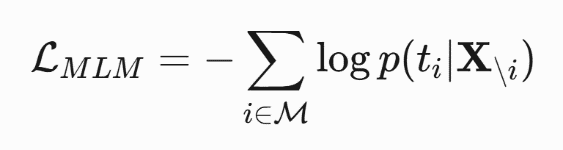
b. 下一句预测(Next Sentence Prediction,NSP):判断两个肽链片段是否属于同一蛋白质结构。
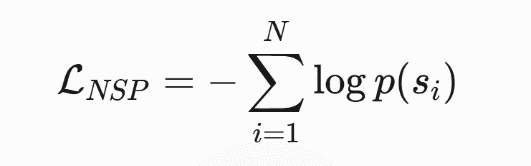
(4) 微调与输出
PeptideBERT 在预训练模型的基础上,针对三个不同的下游任务(如溶血性、溶解性和抗非特异性吸附性)对预训练模型进行微调。微调过程中,模型通过添加一个分类头(MLP)来适应这些任务,并使用特定的数据集进行训练。输出被传递到一个回归头(通常是全连接层),并经过 Sigmoid 函数处理,以确保输出值在 0 和 1 之间。最终,通过设定阈值(如 0.5)将输出值转换为二分类预测结果。
3. 部署
机器
Atlas 800T A2
组件版本
hdk:24.1.RC3
cann:8.0.RC3
torch:2.1.0
torch-npu:2.1.0.post8
环境准备
1. 创建虚拟环境
conda create -n PeptideBERT python=3.9
2. 下载源码
git clone https://github.com/ChakradharG/PeptideBERT.git
3. 安装必要的包
3.1 在PeptideBERT目录下,执行如下命令,安装依赖包
pip install -r requirements.txt
注:a.有一些包依赖于其它包,也需要安装。
b.requirements.txt里面torch的版本是2.0.1。配套的cann版本为7.0.0,由于该版本太过久远,所以这里使用的torch版本为2.1.0,配套的cann版本为8.0.RC3。
c.如果忘记安装必须的依赖包,会报错No module named……
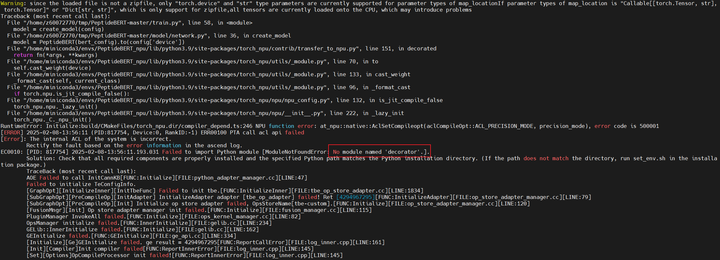
3.2 安装2.1.0版本的torch
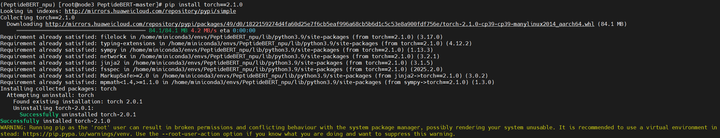
3.3 安装对应版本的torch-npu
a.下载插件包
wget https://gitee.com/ascend/pytorch/releases/download/v6.0.rc3- pytorch2.1.0/torch_npu-2.1.0.post8-cp39-cp39-manylinux_2_17_aarch64.manylinux2014_aarch64.whl
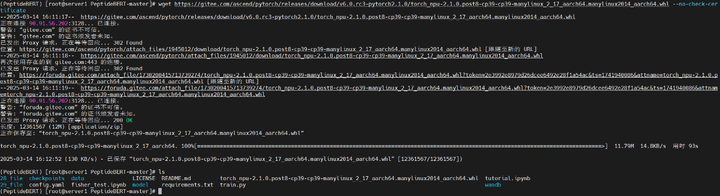
注:如果下载的时候提示证书不可信,需在命令尾部添加--no-check-certificate
b. 安装
pip3 install torch_npu-2.1.0.post8-cp39-cp39-manylinux_2_17_aarch64.manylinux2014_aarch64.whl
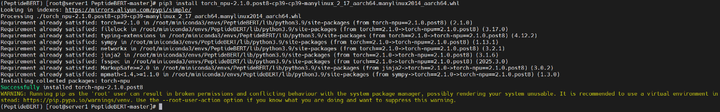
3.4 安装其它的依赖包
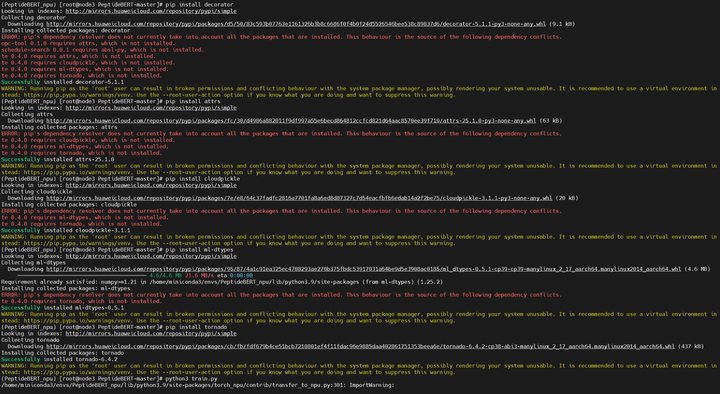
准备数据集
1. 下载数据集
如果直接执行download_data.py脚本,由于网络限制无法进行下载,会返回超时的错误,所以需要手动点击链接下载。
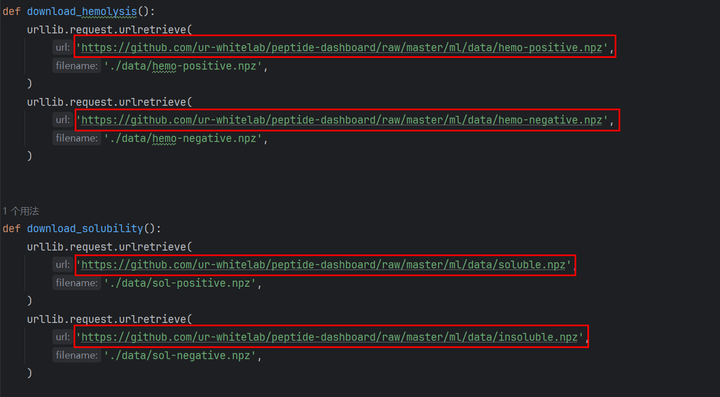
下载完成
根据脚本内的filename修改数据集名字
放至PeptideBERT-master/data/data目录
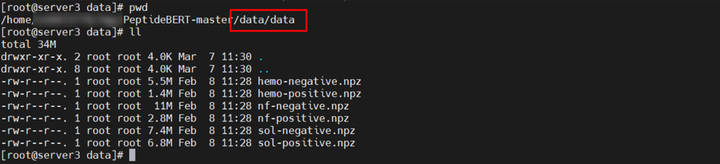
2. 处理数据集
在PeptideBERT-master/data目录下新建py文件load_data.py
文件内容为

执行脚本,会对数据集进行处理。
处理之后
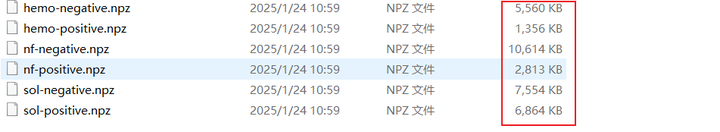
3. 划分数据集
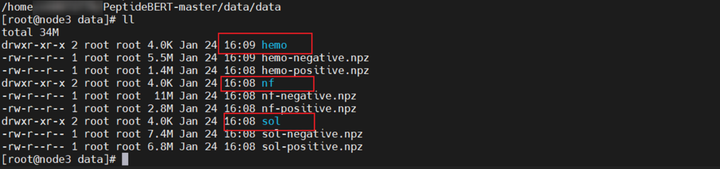
在PeptideBERT-master/data目录下执行 python split_augment.py。
执行之后
在data/data目录下多出来这三个文件夹,里面为划分好的数据集。
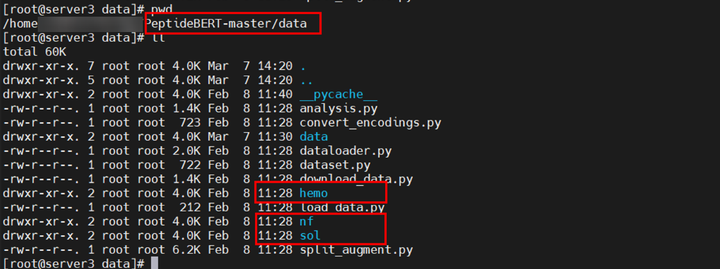
将这三个划分好的数据集移动至PeptideBERT-master/data目录下
训练
1. 开始训练前,需要先source cann

2. 验证npu是否可用

3. 在训练脚本中导入torch_npu

4. 查看配置文件,确保使用的是论文中推荐的最优超参数
数据集选择nf
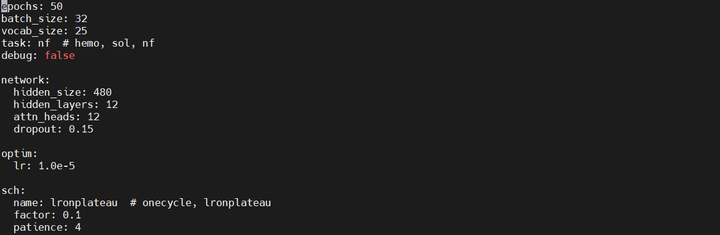
5. 执行训练
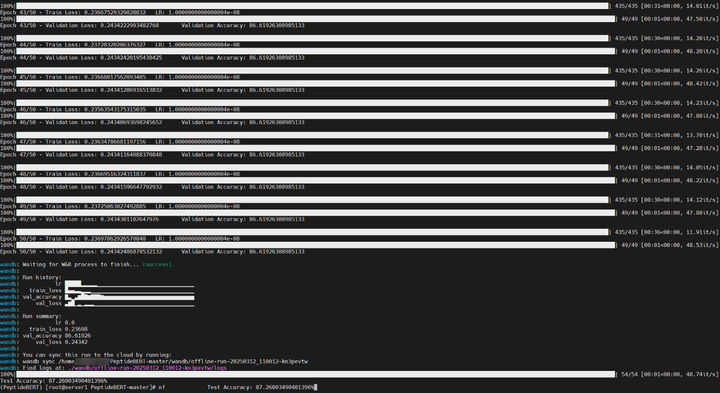
4.结论
在nf数据集上面精度为87.260% ,训练时间为26.67分钟。这一精度表明该模型能可靠的识别出有抗非特异性吸附性潜力的肽,有助于在肽的设计和应用中做出更好的决策。论文中的测试精度为88.365%,训练时间为58.28分钟,在昇腾平台上面以不到一半的训练时间可以达到论文99%的效果。该模型可以在昇腾上进行训练和推理,且性能极强。




















 17万+
17万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








