一 ProteinMPNN介绍
ProteinMPNN(Protein Message Passing Neural Network)是一种基于深度学习的蛋白质序列设计模型,核心目标是解决“逆向折叠问题”(inverse folding problem),即根据给定的蛋白质三维结构,设计出能够折叠成该结构的氨基酸序列。ProteinMPNN在计算和实验测试中都有出色的性能表现,不同位置的氨基酸序列可以在单链或多链之间偶联,从而广泛的应用于当前蛋白质设计上。ProteinMPNN不仅在天然蛋白质序列恢复率上面性能要高于传统的Rosetta方法,并且可以恢复先前设计失败的蛋白质。通过前沿AI技术突破科学研究的效率瓶颈,对于蛋白质工程、药物设计、酶设计等领域有极其重要的意义。
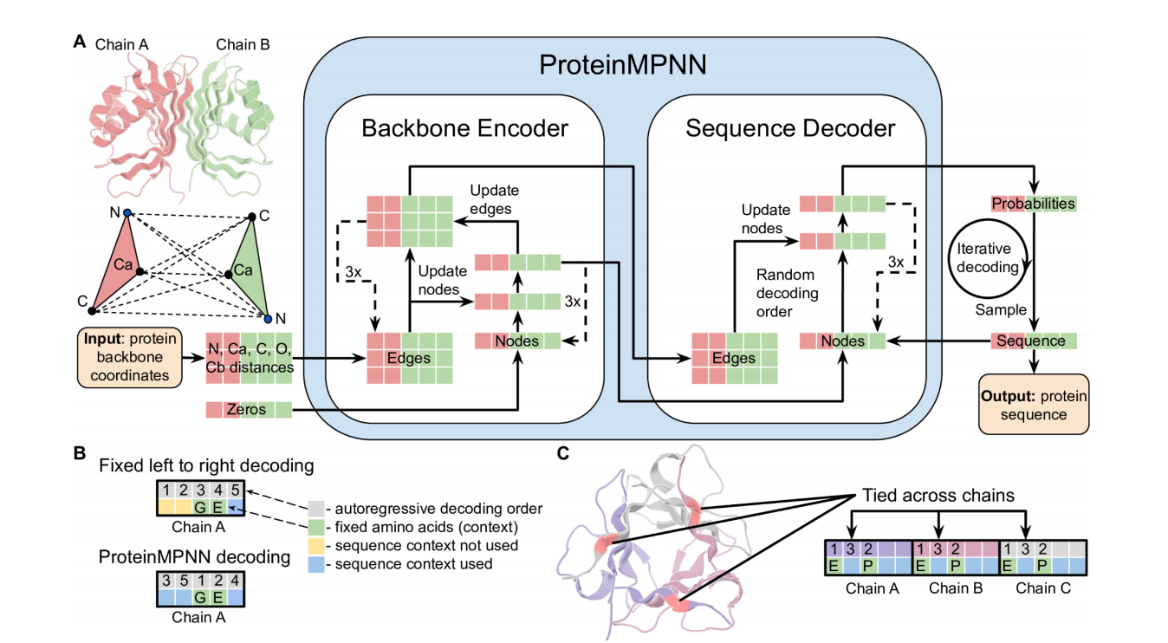
二 整体架构
2.1 编码器
2.1.1 图构建
将蛋白质结构表示为图结构,图的节点代表氨基酸残基,边代表残基之间的空间或序列关系(如距离、接触、氢键等)。
2.1.2 节点嵌入
使用可学习的嵌入层将每个残基的类型(氨基酸种类)、位置信息编码为初始节点特征。
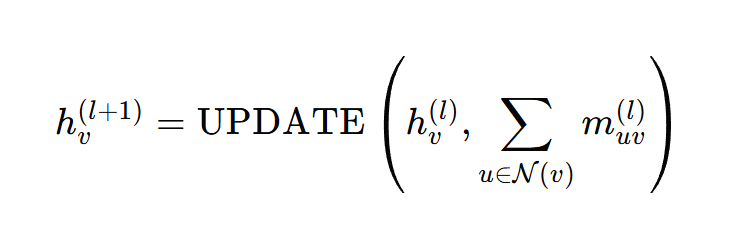
![]() 是节点v在第l层的嵌入向量。
是节点v在第l层的嵌入向量。
![]() 是节点v的邻居节点集合,是从节点u到节点v的消息。
是节点v的邻居节点集合,是从节点u到节点v的消息。
2.1.3 边嵌入
对边进行编码,边的初始特征包括残基之间对距离、角度的几何变换,或残基间的相互作用特征。
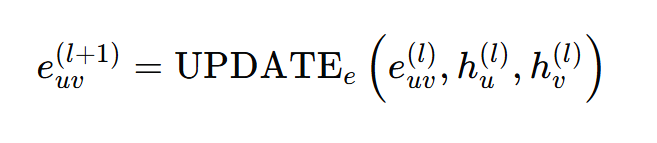
是边(u, v)在第l层的嵌入向量,
![]() 分别是节点u和节点v的嵌入向量。
分别是节点u和节点v的嵌入向量。
2.2 消息传递网络
模型通过多层堆叠的消息传递和节点更新操作迭代更新节点状态
2.2.1 多层图卷积
在每一层图卷积中,节点v接收邻居节点u的消息Muv,由边特征euv和节点特征hu计算得到:





 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 17万+
17万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








