DAP-seq(DNA affinity purification sequencing,DNA亲和纯化测序)技术是ChIP-seq的重要补充方法。通过体外表达转录因子鉴定转录因子结合位点(Transcription factor binding site,TFBS),不受抗体和物种限制,且具有高通量的优势,该技术自问世以来,已被广泛应用于转录调控和表观组学的研究。
背景说明
ChIP-seq可以在体内检测TFBS,较为依赖于抗体质量,对于没有商业化ChIP级抗体可用的物种,转基因实验周期较长,取材困难。DAP-seq通过体外表达蛋白解决了这一困扰,并且极大地提高了TFBS富集效率,在表观遗传学研究过程中表现出极大的价值。
该技术利用体外表达的蛋白和DNA进行亲和纯化,将与蛋白结合的DNA洗脱后进行高通量测序。其基本过程是将编码转录因子的CDS序列构建到含有亲和标签的载体中,构建蛋白表达载体,进行体外蛋白表达,形成转录因子和亲和标签的融合蛋白;提取样品的基因组DNA,构建DNA文库,然后将体外表达的带有亲和标签的转录因子和DNA文库进行结合,随后把结合的DNA洗脱后上机测序。
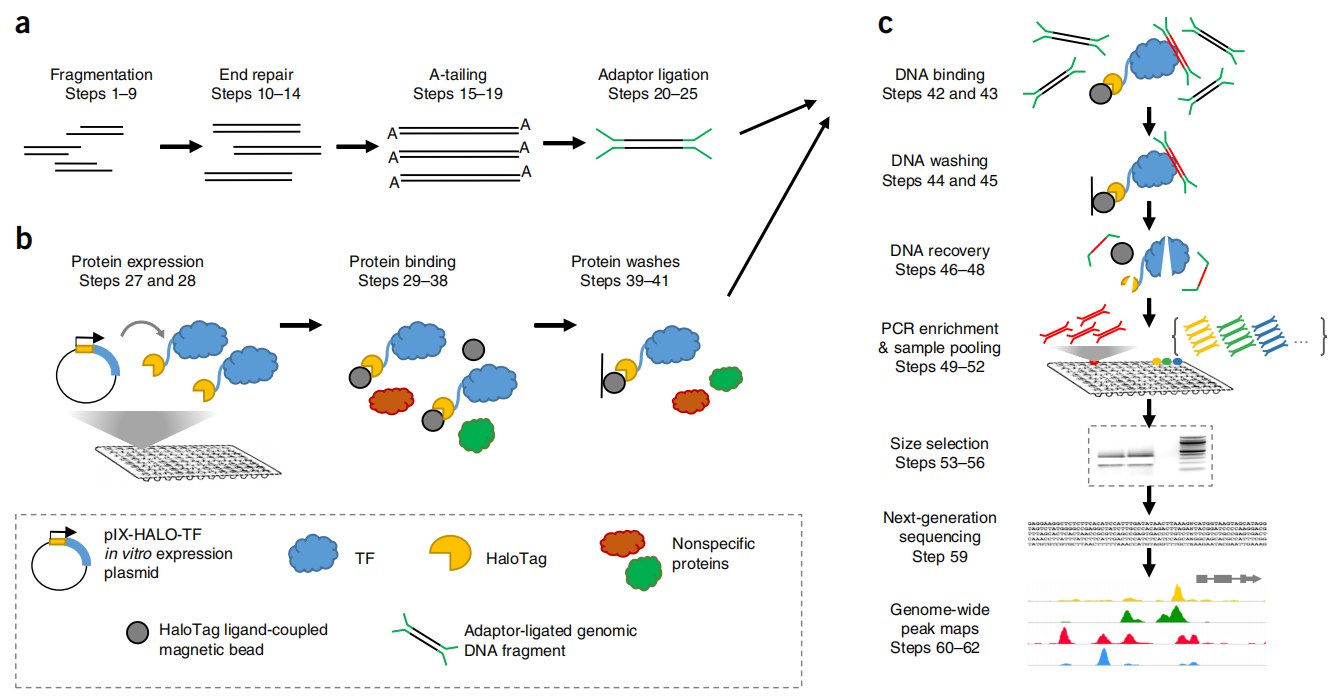
图 DAP-seq实验流程(Bartlett et al., 2017)。
服务流程
订单/实验材料确认——构建蛋白表达载体——蛋白体外表达——提取基因组DNA——DNA片段化——蛋白DNA亲和纯化——DNA文库构建——高通量测序——数据分析——结果交付
服务内容及说明
技术流程
(1)文库构建:提取样品的基因组DNA,并将其片段化,将片段化的DNA与基于Illumina的测序接头连接,构建基因组DNA文库;
(2)体外表达蛋白:将编码目的转录因子的CDS序列构建到带有亲和标签(Halo Tag)的载体中,构建蛋白表达载体,进行体外蛋白表达,形成转录因子和亲和标签的融合蛋白;
(3)亲和纯化及测序:将纯化后的转录因子和亲和标签融合蛋白与基因组DNA文库共同孵育,融合蛋白能与基因组DNA片段上的特定序列结合。通过使用Halo Tag特异性磁珠,将目的蛋白和DNA复合物进行提取和纯化,然后对捕获的DNA片段进行PCR扩增,并利用Illumina测序平台进行测序;
(4)数据分析:将测序产生的序列读数与相应的参考基因组进行比对,从而确定感兴趣转录因子的潜在结合位点。
技术应用
鉴定转录因子与基因组中特定DNA序列结合的位置。
(1)单个转录因子的结合位点鉴定;
(2)同源二聚体或异源二聚体等的结合位点鉴定。
实验周期
致电详询
客户下单及项目信息填写
在我司官网http://www.biorun.com/进行注册或登录,请客户按照页面提示填写项目名称、选择项目类型、填写个人信息及联系方式,提交项目所需要的具体信息(可联系区域销售经理),包括:
1、组织、细胞等样品来源、状态及其他相关信息;
2、参考基因组信息(提供下载链接);
3、尽可能丰富的相关资料、文献。
实验信息
实验过程中客户可以随时登入管理系统查看项目实时进展情况。实验结题时系统会通过短信自动通知客户,并发送实验报告查看网址。实验结题后,实验报告、检测结果可在线查看或打印,并永久保留。
实验交付内容
1、构建的表达质粒的测序结果和图谱文件;
2、蛋白表达的Western blot图;
3、实验报告:提供实验过程的描述包含文库质检结果;
4、生信分析结题报告。
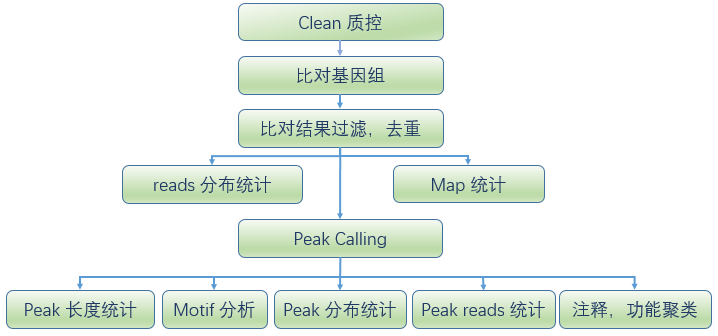
图 DAP-seq生信分析流程。
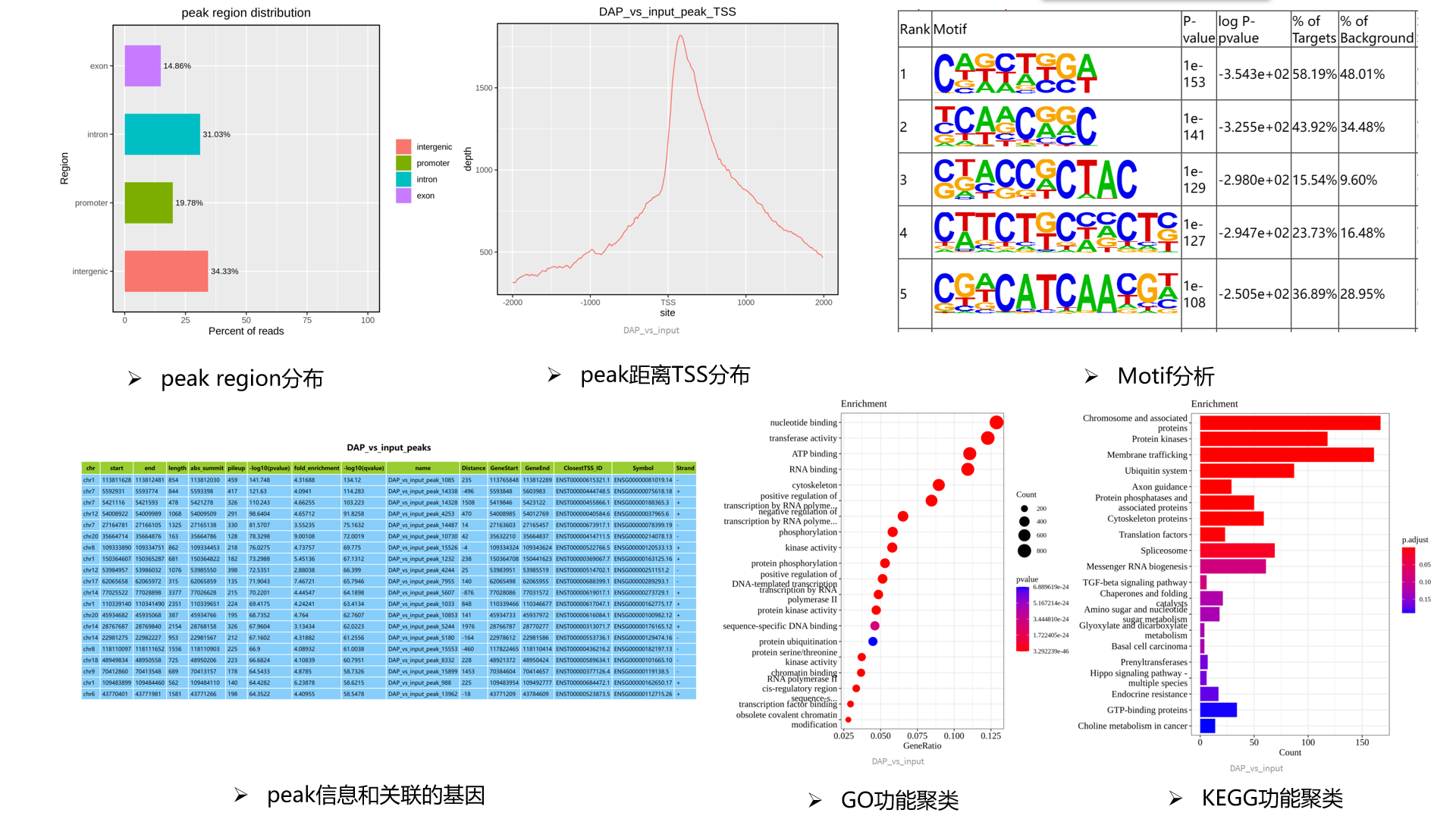
部分结果展示
参考文献
Bartlett A, O'Malley R C, Huang S C, et al. Mapping genome-wide transcription-factor binding sites using DAP-seq[J]. Nature protocols, 2017, 12(8): 1659-1672.




















 1318
1318

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








