AutoDock Vina 是一个用于进行分子对接的开源程序,由Molecular Graphics Lab的Oleg Trott博士开发。根据Oleg Trott等人测试结果,与 AutoDock 4 相比,AutoDock Vina 显着提高了结合模式预测的准确度。此外,Vina 可以利用系统上的多个CPU 内核来显着缩短其运行时间。
一、安装
1. 安装Auto Docking Vina
从http://vina.scripps.edu/download/ Auto Docking Vina下载页面下载Linux版本。我这里是Ubuntu18.0系统
解压:tar xzvf autodock_vina_1_1_2_linux_x86.tgz
得到autodock_vina_1_1_2_linux_x86文件夹,
./autodock_vina_1_1_2_linux_x86/bin/vina为执行程序,或./vina即可运行vina程序,结果如下图:
Docking之前需要对小分子和蛋白受体进行准备,包括小分子3维构象的生成,PDB文件转化为pdbqt文件,添加H原子,添加autodocking特有的AD4原子类型,甚至还包括一些键的柔性这些。这些准备过程有大量的工具可以使用,主要是openbabel和MGLtools。
2. 安装open babel -V
可以使用conda安装,也可以源码编译安装。即使是使用conda安装,安装完以后其实也不依赖于conda环境,直接在命令行中输入obabel -V也是可以运行的。如果采用Python中调包的形式,则要在特定的conda环境中。
我的安装方式:conda install -c openbabel openbabel
3. 安装mgltools
由于我们用的是没有图形界面的ubuntu系统,所以MGLtools安装命令行版本即可。从https://ccsb.scripps.edu/mgltools/downloads/ 下载mgltools_x86_64Linux2_1.5.7.tar.gz (Linux 64 tarball installer 108Mb)安装包。
注意,直接wget安装包会有问题。将安装包迁移至指定路径,然后解压,例如:
tar -axvf mgltools_x86_64Linux2_1.5.7.tar_.gz #解压
cd mgltools_x86_64Linux2_1.5.7/ #进入安装文件夹
sudo bash install.sh #安装
pwd #当前的工作路径,/home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/MGLtools-install/mgltools_x86_64Linux2_1.5.7
添加环境变量,建立alias链接,在~/.bashrc中添加(使用sudo用户):
export PATH=/home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/MGLtools-install/mgltools_x86_64Linux2_1.5.7/bin:$PATH
alias pmv='/home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/MGLtools-install/mgltools_x86_64Linux2_1.5.7/bin/pmv'
alias adt='/home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/MGLtools-install/mgltools_x86_64Linux2_1.5.7/bin/adt'
alias vision='/home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/MGLtools-install/mgltools_x86_64Linux2_1.5.7/bin/vision'
alias pythonsh='/home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/MGLtools-install/mgltools_x86_64Linux2_1.5.7/bin/pythonsh'
注:/home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/是我的安装目录,大家可以根据自己的情况自行指定。
保存后刷新initMGLtools.sh文件。刷新完以后,输入pythonsh则可以使用MGLTools的Python环境(MGLTools是自带Python环境的)。
由于MGLtools工具自带Python编译器,为了不与其他Python环境造成冲突,所以需要修改当前路径下MGLToolsPckgs/AutoDockTools/Utilities24文件夹下的py脚本。例如:
vim ./MGLToolsPckgs/AutoDockTools/Utilities24/prepare_receptor4.py
将第一行替换为:
#!/usr/bin/env /home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/MGLtools-install/mgltools_x86_64Linux2_1.5.7/bin/python
再次注明,注:/home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/是我的安装目录,大家可以根据自己的情况自行指定。
prepare_ligand4.py文件也一样修改第一行。
二、使用Vina进行分子对接(基本对接)
这里的基本对接指的是单分子的对接。而不是分子库,如果是分子库就更复杂一些,需要循环语句+多线程,如果有资源可以使用集群进行对接,可那就更复杂了,具体可以参考我以后的内容。
使用Vina进行分子对接的流程如下图。
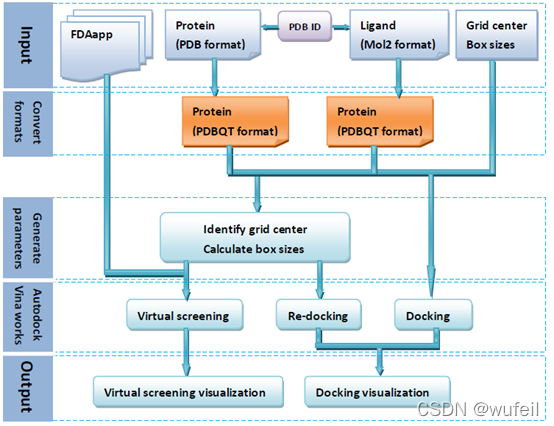
简单来说,蛋白质受体文件预处理且保存成pdbqt格式,小分子预处理且也要保存成pdbqt格式,然后对接中心需要指定。
pdbqt 格式类似于 pdb 格式,但还包含有关哪些扭转应处于活动状态以及为每个原子分配的电荷的信息。 Vina 和 Smina 不使用与 Autodock 相同的评分函数,并且在默认评分函数中根本不使用电荷。
1. 使用pymol删除不必要的水原子和重原子,添加H原子,保存成PDB文件
以1OYT蛋白为例,注:1OYT为单链分子。
在pymol的命令行下输入如下内容:
remove resn HOH #删除水分子
h_add elem O or elem N #给N原子和O原子加氢
select 1OYT-FSN, resn FSN #选择小分子
select 1OYT-receptor, 1OYT and not 1OYT-FSN #选择配体蛋白
save 1OYT.pdb, 1OYT-receptor #保存蛋白配体
save mol.pdb, 1OYT-FSN #保存小分子
2.使用MGLtools将PDB文件转化为vina需要的pdbqt文件格式
首先要先确认Utilities24文件夹的路径,我这里是:/home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/MGLtools-install/mgltools_x86_64Linux2_1.5.7/MGLToolsPckgs/AutoDockTools/Utilities24。在使用的时候可以先cd到相应的文件夹中去。然后使用如下命令将受体文件1OYT.pdb生成1OYT.pdbqt:
pythonsh /home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/MGLtools-install/mgltools_x86_64Linux2_1.5.7/MGLToolsPckgs/AutoDockTools/Utilities24/prepare_receptor4.py -r 1OYT.pdb
将小分子文件生成pdbqt文件(-l 配体文件名,支持.pdb or .mol2 or .pdbq; -F 检查并使用最大的非共价片段; -A 修补添加bonds_hydrogens, bonds, hydrogens等,需要参数,默认是不修补的)
pythonsh /home/ubuntu/wufeil/Deepchem-RDKit/Autodock_Vina/MGLtools-install/mgltools_x86_64Linux2_1.5.7/MGLToolsPckgs/AutoDockTools/Utilities24/prepare_ligand4.py -l 1a3w-mol.pdb -F -A hydrogens
3.使用openbabel将PDB文件转化为vina需要的pdbqt文件格式
Open-babel 可以快速为我们提供了两个需要的 .pdbqt 文件。例如:
obabel 1OYT-FSN.pdb -O 1OYT-FSN.pdbqt
obabel 1OYT-receptor.pdb -xr -O 1OYT-receptor.pdbqt
4.使用vina进行分子对接
对接前需要先写config.txt文件。文件中可以写入docking需要的一些参数,当然这些参数也可以在vina命令时候输入。
例如,config.txt,我写入:
center_x = 17.057
center_y = -6.468
center_z = 23.461
size_x = 15.0
size_y = 15.0
size_z = 15.0
执行命令,进行对接:
./autodock_vina_1_1_2_linux_x86/bin/vina --ligand 1OYT-FEN.pdbqt --receptor 1OYT-receptor.pdbqt --exhaustiveness 32 --config config.txt --out result.pdbqt
三、对接结果
对接结果如下图:
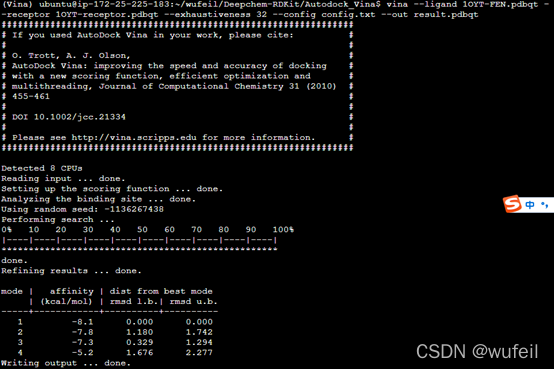
上述是对接一个分子的过程,再具体的案例中,如何对接100万个分子,如果这些分子没有生成三维构象呢?对接一个分子需要1~10秒,那么对接100万的分子,显然在单台机子上难以完成,或者耗时很长。如果有大规模对接需求可以私信我。





 本文详细介绍了如何在Ubuntu系统上安装AutoDockVina,包括openbabel、MGLtools的配置,以及如何使用Vina进行小分子与蛋白质的对接,涉及PDB文件处理和基本对接步骤。适合初学者了解分子对接流程。
本文详细介绍了如何在Ubuntu系统上安装AutoDockVina,包括openbabel、MGLtools的配置,以及如何使用Vina进行小分子与蛋白质的对接,涉及PDB文件处理和基本对接步骤。适合初学者了解分子对接流程。

















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










