本篇博客是向这篇文章学习后的一个学习记录,由于作者还在初学阶段,有些内容可能描述不清,望见谅,首先感谢这位博主的这篇文章:
关于nnunet的简介可以看博主的这篇文章:
步骤
这里以浪潮人工智能平台为例,使用nnunet网络对Synapse数据集进行分割。
1.安装nnUNet框架
一般来说可以首先创建一个开发环境,使用shell终端输入如下命令进行git,git clone其实就是把人家github上的代码克隆过来,这一步其实和直接复制粘贴代码文件是一样的。
git clone https://github.com/MIC-DKFZ/nnUNet.git
cd nnUNet
pip install -e .#最后这个点也不能忽略
但是一般会连接超时,目前我还没找到特别好的方法去解决,所以建议直接访问https://github.com/MIC-DKFZ,根据需求或者python版本问题,选择下载nnunet的v1或者v2版本,上传到服务器,然后再解压。
最后pip install -e .相当于python setup.py,也就是运行上图这个setup.py文件。它的作用如下:
(1)安装nnUNet需要的python包;
(2)向终端添加几个新命令。这些命令用于运行整个nnU-Net pipeline。您可以从系统上的任何位置执行它们。所有nnU-Net命令都带有前缀“nnUNet_”,以便于识别。
2.数据集准备
首先自行下载需要使用的数据集,但是不要着急上传,nnUNet对于要训练的数据是有严格要求的。
(1)进入名为nnUNet的文件夹,在里面创建一个名为nnUNetFrame的文件夹。
(2)在nnUNetFrame文件夹中创建一个名为DATASET的文件夹,后面会用它来存放数据。
(3)在DATASET文件夹中创建三个文件夹,它们分别是nnUNet_raw,nnUNet_preprocessed,nnUNet_trained_models。
(4)进入上面第二个文件夹nnUNet_raw,创建nnUNet_cropped_data文件夹和nnUNet_raw_data文件夹,右边存放原始数据,左边存放crop以后的数据。
(5)进入右边文件夹nnUNet_raw_data:
我所学习的文章中提到创建一个名为Task01_BrainTumour的文件夹(解释:这个Task01_BrainTumour是nnUNet的作者参加的一个十项全能竞赛的子任务名,也是我要实践的分割任务,类似的还有Task02_Heart,就是分割心脏的。)
如果想分割自己的数据集,可以Task_id从500开始,这样以确保不会与nnUNet的预训练模型发生冲突(ID不能超过999)。
(6)将下载好的公开数据集或者自己的数据集放在上面创建好的任务文件夹下。这里我命名为Task500_Synapse。
设置三个环境变量(虽然说得是永久写入,但是在开发环境中是生效的,训练环境的时候依旧要在命令模式中加入临时设置环境变量的命令,否则出错,但是这一点我还没搞清楚):
# 永久写入 ~/.bashrc
echo 'export nnUNet_raw_data_base="/xujiheng/Synapse/nnUNet/nnUNet/nnUNetFrame/DATASET/nnUNet_raw"' >> ~/.bashrc
echo 'export nnUNet_preprocessed="/xujiheng/Synapse/nnUNet/nnUNet/nnUNetFrame/DATASET/nnUNet_preprocessed"' >> ~/.bashrc
echo 'export RESULTS_FOLDER="/xujiheng/Synapse/nnUNet/nnUNet/nnUNetFrame/DATASET/nnUNet_trained_models"' >> ~/.bashrc
# 立即生效
source ~/.bashrc检查:
echo $nnUNet_raw_data_base
# 应输出:/xujiheng/Synapse/nnUNet/nnUNet/nnUNetFrame/DATASET/nnUNet_raw3.数据集转换
nnUNet要求将原始数据转换成特定的格式,以便了解如何读取和解释数据。每个分割数据集存储为单独的“任务”,命名包括任务与任务ID,即三位整数和相关联的任务名称。
重命名
首先使用命令批量对数据集重命名为指定格式,nnU-Net 要求,对于每个样本,imagesTr/<ID>_0000.nii.gz 必须对应 labelsTr/<ID>.nii.gz。以synapse数据集为例:
训练集:
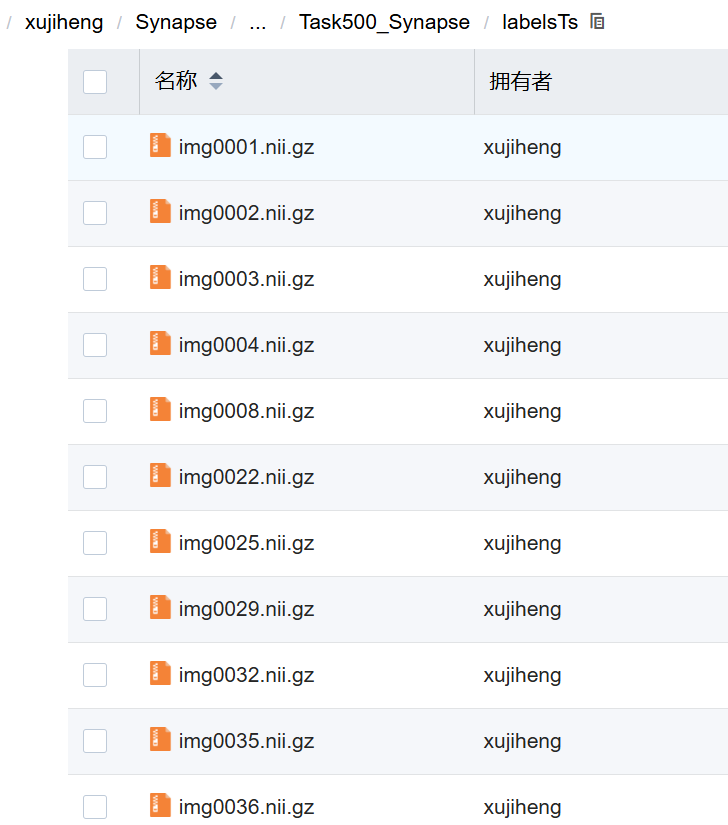
训练集标签:
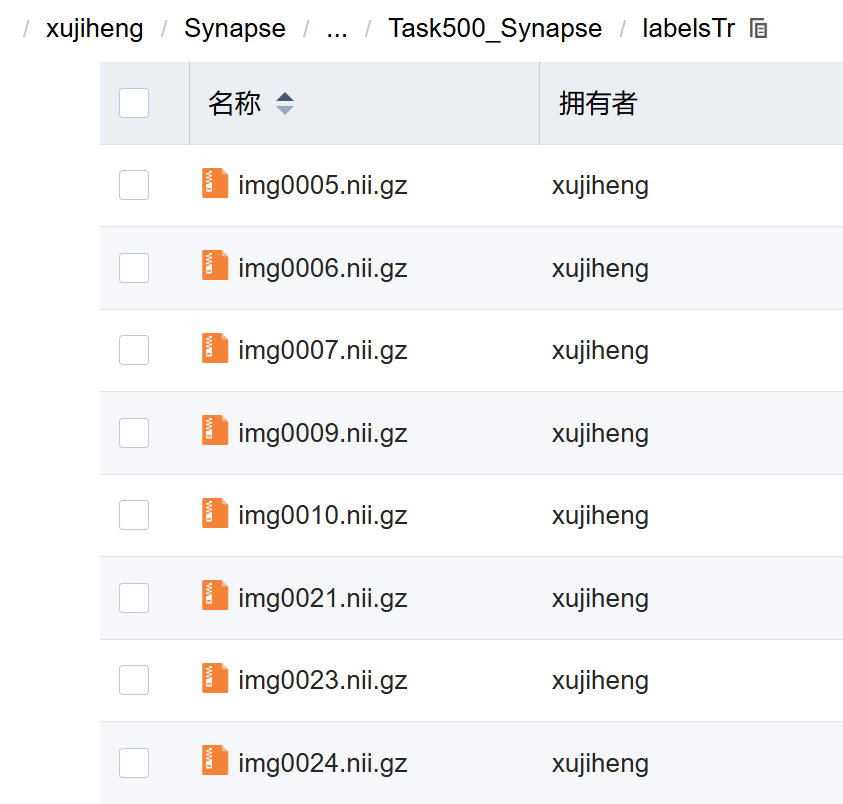
测试集:
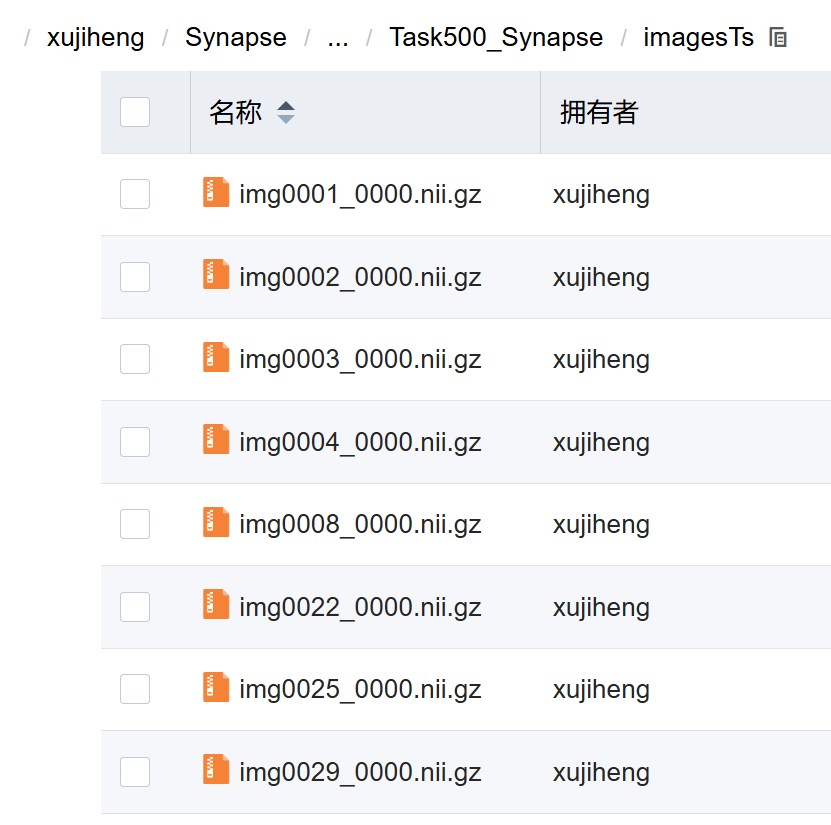
测试集标签:
生成dataset.json
作用可以概括为“扫描本地 imagesTr、labelsTr、imagesTs,把能找到的病例 ID 按 nnUNet v1 格式写进 dataset.json,并给 Synapse 八器官分配 0–8 的标签索引。”。
cd /xujiheng/Synapse/nnUNet/nnUNet/nnUNetFrame/DATASET/nnUNet_raw/nnUNet_raw_data/Task500_Synapse
python3 -c "
import os, json
task_dir = '.'
imagesTr = os.path.join(task_dir, 'imagesTr')
labelsTr = os.path.join(task_dir, 'labelsTr')
imagesTs = os.path.join(task_dir, 'imagesTs')
# Get training IDs
train_ids = []
for f in os.listdir(imagesTr):
if f.endswith('_0000.nii.gz'):
case_id = f[:-12] # remove '_0000.nii.gz'
if not os.path.exists(os.path.join(labelsTr, case_id + '.nii.gz')):
raise RuntimeError('Missing label: ' + case_id + '.nii.gz')
train_ids.append(case_id)
# Get test IDs
test_ids = []
if os.path.isdir(imagesTs):
for f in os.listdir(imagesTs):
if f.endswith('_0000.nii.gz'):
test_ids.append(f[:-12])
# Build dataset.json
dataset = {
'name': 'SYNAPSE',
'description': 'Synapse transitional zone and peripheral zone segmentation',
'reference': 'Radboud University, Nijmegen Medical Centre',
'licence': 'CC-BY-SA 4.0',
'release': '1.0 04/05/2018',
'tensorImageSize': '3D',
'modality': {'0': 'CT'},
'labels': {
'0': 'background',
'1': 'Aorta',
'2': 'Gallbladder',
'3': 'Kidney(L)',
'4': 'Kidney(R)',
'5': 'Liver',
'6': 'Pancreas',
'7': 'Spleen',
'8': 'Stomach'
},
'numTraining': len(train_ids),
'numTest': len(test_ids),
'training': [
{'image': './imagesTr/' + i + '.nii.gz', 'label': './labelsTr/' + i + '.nii.gz'}
for i in sorted(train_ids)
],
'test': [
'./imagesTs/' + i + '.nii.gz'
for i in sorted(test_ids)
]
}
with open('dataset.json', 'w') as f:
json.dump(dataset, f, indent=4)
print('dataset.json created with', len(train_ids), 'training and', len(test_ids), 'test cases.')
"预处理数据:
nnUNet_plan_and_preprocess -t 500 -tl 8预处理具体做了什么?(以 Synapse CT 为例)
对每个 imgXXXX.nii.gz(图像)和 imgXXXX.nii.gz(标签):
(1)读取原始 NIfTI 文件
(2)裁剪(Crop)
找到所有非零区域(foreground,即人体区域)
去掉大片黑色背景(节省 50%+ 空间和计算量)
(3)重采样(Resample)
将所有图像统一重采样到一个“典型 spacing”(比如 [1.5, 1.5, 2.0] mm)
保证所有样本输入尺寸一致
(4)强度归一化(Intensity Normalization)
CT 图像:截断 HU 值到 [-1000, 1000](空气到骨骼)
然后做 z-score 归一化(均值为 0,标准差为 1)
(5)保存为 .npz
比 .nii.gz 读取快 5~10 倍
直接加载为 numpy 数组,无需 nibabel 解析
检查预处理是否成功:
# 检查关键文件是否存在 + 数量是否正确
echo "=== 检查 plans.pkl ==="
ls -l $nnUNet_preprocessed/Task500_Synapse/plans.pkl
echo -e "\n=== 检查预处理数据数量(应为 18 个 .npz + 18 个 .pkl = 36 个文件)==="
ls $nnUNet_preprocessed/Task500_Synapse/nnUNetData_plans_v2.1_3D/ | wc -l
echo -e "\n=== 检查是否包含 3d_fullres 配置 ==="
python3 -c "
import pickle
with open('$nnUNet_preprocessed/Task500_Synapse/plans.pkl', 'rb') as f:
plans = pickle.load(f)
print('3d_fullres in plans:', '3d_fullres' in plans['plans_per_stage'])
print('Patch size for 3d_fullres:', plans['plans_per_stage']['3d_fullres']['patch_size'])
"
echo -e "\n=== 检查 cropped data 是否存在 ==="
ls $nnUNet_raw_data_base/nnUNet_cropped_data/Task500_Synapse/ | head -n 34.启动训练
创建训练环境,使用命令模式,输入如下命令:
注:这里是旧版nnunetv1的,v2有不同之处,因为作者现在因python版本限制,只能使用v1。
export nnUNet_raw_data_base="/xujiheng/Synapse/nnUNet/nnUNet/nnUNetFrame/DATASET/nnUNet_raw" && export nnUNet_preprocessed="/xujiheng/Synapse/nnUNet/nnUNet/nnUNetFrame/DATASET/nnUNet_preprocessed" && export RESULTS_FOLDER="/xujiheng/Synapse/nnUNet/nnUNet/nnUNetFrame/DATASET/nnUNet_trained_models" && python /xujiheng/Synapse/nnUNet/nnUNet/nnunet/run/run_training.py 3d_fullres nnUNetTrainerV2 500 all命令解释:
(1)设置三个旧版环境变量
nnUNet_raw_data_base :原始数据根目录(等价于 v2 的 nnUNet_raw) /xujiheng/Synapse/…/nnUNet_raw
nnUNet_preprocessed :预处理输出目录(等价于 v2 的 nnUNet_preprocessed) /xujiheng/Synapse/…/nnUNet_preprocessed
RESULTS_FOLDER :训练 checkpoint & 日志(等价于 v2 的 nnUNet_results) /xujiheng/Synapse/…/nnUNet_trained_models
(2)参数
3d_fullres 使用“3D 全分辨率”配置
nnUNetTrainerV2 训练器类名(旧版默认就是这个,不用改)
500 Task ID = 500
all 把 5 个 fold 全部跑一遍(等价于 fold 0 1 2 3 4 连续训练)
补充:
fold 直译是“折”,在机器学习里特指 交叉验证(cross-validation) 的“折号”。
nnUNet 说的“跑 5 个 fold”就是 5 折交叉验证:把训练集平均切成 5 份,依次拿其中 1 份当验证集、其余 4 份当训练集,循环 5 次,得到 5 个模型,最后再平均/投票得出最终性能。好处是:
(1)训练集里每一份数据都当过 1 次验证,结果更稳定;
(2)能给出 Dice 等指标的均值±标准差,方便写论文;
(3)推理时可把 5 个模型做 模型集成(ensemble),Dice 通常还能再涨 0.5–1 %。




















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








