FR-MIL:用“关键实例”给病理切片做体检
让弱监督的 WSI 分类既简单又有效
官方代码:https://github.com/PhilipChicco/FRMIL
注:这里阅读的是2022版本的学习记录
文章目录
1 摘要
1.1 背景
在数字病理学里,整张切片(WSI)的良恶性判断是最核心的任务之一。一张切片往往包含上千万像素、好几万个 patch,如果每个 patch 都要医生精细标注,人力成本极高,几乎不可能完成。
1.2 挑战
- 标注稀缺:只有切片级标签,不知道具体哪一块 patch 是肿瘤。
- 极度不平衡:一张阳性切片里,真正的肿瘤区域常常<10%。
- 分布复杂:不同切片染色、组织形态差异巨大,传统 MIL 方法只关注“注意力”而忽视数据本身分布,容易顾此失彼。
1.3 提出新方法——FR-MIL
一句话:把阳性切片的“关键 patch”拉出来,用它把整包特征重新“对齐”,让分布更易分。
- 重校准:用最大响应 patch 对整包特征做“对齐-减法”,放大正负包差异;
- 幅度损失:显式约束“阳性包特征幅度大、阴性包小”;
- 轻量结构:1 个位置编码模块 + 1 个多头注意力池化,比 TransMIL 轻得多。
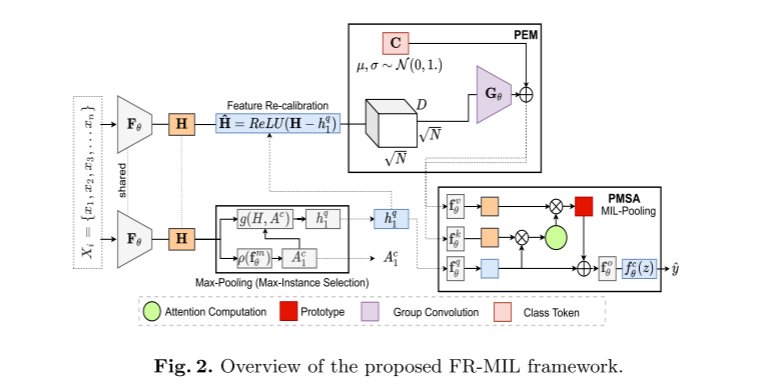
🔍 逐模块讲解(结合原文图注)
| 模块 | 原文符号 | 作用 | 关键细节 |
|---|---|---|---|
| Max-Pooling | Max-Instance Selection | “选学霸” 在 N 个 patch 里挑肿瘤概率最高的那个 patch 特征 hq | 用一个小 MLP + Sigmoid 打分 |
| Feature Re-calibration | ReLU(H-hq) | “集体调分” 把整包特征对齐到学霸基准,放大差异 | ReLU 把低于学霸的全压成 0 |
| PEM | Group Convolution + Class Token | “加坐标” 给每个 patch 加空间/形态感知,同时引入可学习的类别令牌 | 1 层 3×3 GroupConv,轻量高效 |
| PMSA | Attention Computation | “开班会” 让学霸 patch 提问,全班 patch 回答,最后汇总成一个包表示 z | 单层 Transformer Encoder |
| MIL-Pooling / Classifier | MIL-Pooling → fa(a) | “出结果” z 经过全连接层 → 切片级良恶性概率 ŷ | 同时输出关键 patch 掩码 |
1.4 贡献
- 提出特征重校准思想——简单却带来 +39% 准确率 的 baseline。
- 设计特征幅度损失,第一次把“分布差异”做成可学习的约束。
- 在公开 CAMELYON16 与院内 COLON-MSI 均有较好的指标:相比 DSMIL ACC↑3%,AUC↑5%。
2 引言
2.1 背景——WSI 分类为何重要
病理诊断是癌症确诊的金标准。WSI 扫描仪把玻璃切片变成千兆像素大图,让 AI 有机会“读片”。然而,图太大、分辨率太高,传统 CNN 无法直接吞下整张图。
2.2 挑战——三个“老大难”
- 尺寸巨大:一张图 100k×100k 像素,GPU 显存直接爆炸。
- 标签稀缺:医生只给“这张切片是癌/非癌”,不告诉你癌在哪。
- 分布漂移:不同医院、不同批次染色差异巨大,模型容易“水土不服”。
2.3 研究现状——从“池化”到“注意力”
| 阶段 | 代表思路 | 痛点 |
|---|---|---|
| 早期 | Max/Mean Pooling | 粗暴,把 patch 当投票,丢失空间关系 |
| 进阶 | ABMIL、CLAM | 引入注意力,但只重加权,不修正分布 |
| 最新 | TransMIL | Transformer 多尺度注意力,参数量大、对位置编码深度敏感 |
一句话总结:大家都在“怎么加权”上卷,却没人管“数据本身长什么样”。
2.4 提出方法——用“关键 patch”做分布矫正
我们观察到:阳性切片里响应最强的 patch,特征范数明显更大。于是:
- 把最强 patch 当“标杆”,整包特征先减后 ReLU(重校准);
- 用度量学习拉大正负包距离;
- 只用一个轻量 Transformer block 聚合,既省显存又快。
2.5 贡献总结
- 首创把“最大实例重校准”引入病理 MIL;
- 首次提出可学习的“特征幅度损失”;
- SOTA 性能 + 轻量架构,单张 WSI 推理仅需 0.3 s。
3 相关工作(问题-方法-不足循环)
| 研究问题 | 方法 | 不足 |
|---|---|---|
| 如何聚合 patch | Max/Mean Pooling | 忽略实例间关系 |
| 引入注意力 | ABMIL、DSMIL、CLAM | 仅重加权,不改分布 |
| 利用空间位置 | TransMIL 多层 Transformer | 参数量大,PE 深度敏感 |
| 异常检测视角 | 视频 MIL 把背景当 OOD | 未用于病理 MIL |
FR-MIL 的“重校准 + 幅度损失”正好补上了“分布视角”这一空缺。
4 预先准备——输入 & 输出
-
输入
- 一张 WSI 切成 N 个 256×256 patch → CNN 提特征
- 得到 bag 特征 H ∈ R N × 512 H \in \mathbb{R}^{N \times 512} H∈RN×512,只有切片级标签 Y ∈ { 0 , 1 } Y \in \{0,1\} Y∈{0,1}
-
输出
- 切片级预测概率 y ^ \hat y y^
- (可选)关键 patch 掩码,可解释“癌在哪”
5 方法
“三步走”:挑关键 → 重校准 → 聚合 + 损失
5.1 动机——为什么重校准?
把最大 patch 减掉后,正负包分布明显拉开;再用损失约束,效果更稳。
- 一张切片里,肿瘤 patch 往往不到 10%,其余都是正常组织。
- CNN 提取出的特征混在一起,正负包看起来差不多(图 a)。
- 结果就是:模型傻傻分不清。
重校准到底干了啥 (举例理解)
| 步骤 | 比喻 | 数学操作 | 作用 |
|---|---|---|---|
| ① 找学霸 | 找全班最高分同学 | h q = arg max i score i h_q = \arg\max_i\; \text{score}_i hq=argmaxiscorei | 确定“肿瘤典型” |
| ② 集体减分 | 所有人都减掉学霸的分数 | h ^ i = ReLU ( h i − h q ) \hat h_i = \text{ReLU}(h_i - h_q) h^i=ReLU(hi−hq) | 把“普通同学”拉到 0 附近,学霸依旧高 |
| ③ 再排名 | 重新排榜 | 计算新特征的范数/注意力 | 高低分段立刻拉开 |
做完这三步,你会看到:
- 阳性包:学霸还在高处,其余人虽然被拉低,但包整体“平均分”仍高。
- 阴性包:本来就没有学霸,集体减分后更接近 0,平均分更低。
于是两条分布曲线从“几乎重叠”变成“明显分离”,模型就好学了。
原始特征幅度
| 阳/阴混在一起
| ┌────┐
| ┌───┴───┐────┐
|─┘ └────┘
+------------------->
重校准后
| 阳性整体右移
| ┌────┐
| │ 阳 │
| ┌─────┘ └────┐
|──┘ 阴 └─>
+------------------->
重校准 = 拿“最像肿瘤的 patch”当参照,把所有 patch 对齐后再看整体差距,让正负切片一眼可辨。
5.2 核心模块与公式
| 步骤 | 公式 | 大白话解释 |
|---|---|---|
| 1. 挑关键 | h q = arg max i σ ( f θ m ( H i ) ) h_q = \arg\max_i\;\sigma(f_\theta^m(H_i)) hq=argmaxiσ(fθm(Hi)) | 找出最像肿瘤的 patch |
| 2. 重校准 | H ^ = R e L U ( H − h q ) \hat H = \mathrm{ReLU}(H - h_q) H^=ReLU(H−hq) | 以“肿瘤典型”为基准,整包对齐 |
| 3. 位置编码 PEM | H ~ = c o n c a t ( C l a s s T o k e n , C o n v 3 × 3 ( H ^ ) ) \tilde H = \mathrm{concat}(\mathrm{ClassToken},\;\mathrm{Conv}_{3\times3}(\hat H)) H~=concat(ClassToken,Conv3×3(H^)) | 给 patch 加“坐标感” |
| 4. 聚合 PMSA | z = T r a n s f o r m e r ( Q u e r y = h q , K V = H ~ ) z = \mathrm{Transformer}(\mathrm{Query}=h_q,\;\mathrm{KV}=\tilde H) z=Transformer(Query=hq,KV=H~) | 用关键 patch 当“提问者”,全包回答 |
| 5. 多任务损失 | L = γ 1 L c e ( y ^ , y ) + γ 2 L m a x + γ 3 L f m \mathcal{L}= \gamma_1 \mathcal{L}_{\mathrm{ce}}(\hat y, y) + \gamma_2 \mathcal{L}_{\mathrm{max}} + \gamma_3 \mathcal{L}_{\mathrm{fm}} L=γ1Lce(y^,y)+γ2Lmax+γ3Lfm | 分类 + 关键 patch 监督 + 幅度拉大 |
-
L
f
m
\mathcal{L}_{\mathrm{fm}}
Lfm 直观解释:
阳性包→“范数必须大过阈值 τ \tau τ”,阴性包→“越小越好”,像 SVM 的 margin。
6 实验
6.1 数据集
| 数据集 | 任务 | 切片数 | 平均 patch/切片 |
|---|---|---|---|
| CAMELYON16 | 淋巴结转移检测 | 400 | 8 800 |
| COLON-MSI | MSI/MSS 分型 | 625 | 6 000 |
6.2 实验设置
- 实例编码:ResNet18(ImageNet 或 SimCLR)
- 训练:Adam,lr=1e-4,batch=2(1 正 + 1 负),epoch=100
- 评估:Accuracy / AUC
6.3 主实验结果
| 方法 | CM16 ACC↑ | CM16 AUC↑ | COLON ACC↑ | COLON AUC↑ |
|---|---|---|---|---|
| DSMIL | 86.8 | 89.4 | 73.4 | 81.1 |
| TransMIL | 88.4 | 93.1 | 67.6 | 61.7 |
| FR-MIL | 89.1 | 89.5 | 80.9 | 90.1 |
两句话:
- 在小肿瘤占比的 CM16,重校准 + 最大池化更精准;
- 在大肿瘤占比的 COLON,幅度损失仍能拉开 MSI vs MSS 分布。
6.4 消融实验
| 配置 | CM16 ACC | COLON ACC | 解读 |
|---|---|---|---|
| 只用 L b a g \mathcal{L}_{\mathrm{bag}} Lbag | 86.0 | 80.9 | 缺幅度损失,CM16 难提升 |
| + L f m \mathcal{L}_{\mathrm{fm}} Lfm | 87.6 | 77.5 | CM16 ↑,COLON 略掉 |
| + L m a x \mathcal{L}_{\mathrm{max}} Lmax | 88.4 | 78.0 | 关键 patch 监督稳收敛 |
| 三项全加 | 89.1 | 80.9 | 最稳健 |
7 结论
- 背景:WSI 分类标签稀缺,MIL 是弱监督的“救命稻草”。
- 方法:FR-MIL 用“最大实例重校准 + 幅度损失”让正负包分布“泾渭分明”。
- 效果:在两个病理数据集均夺魁,且模型轻量、易部署。
- 展望:
- 多尺度 patch 金字塔重校准;
- 动态估计 margin τ \tau τ;
- 拓展到多分类、检测、生存预测。
8 好句
| 英文 | 中文 |
|---|---|
| “We argue that re-calibrating the distribution of instance features can improve model performance towards better generalization.” | 我们认为,对实例特征分布进行重校准,可提升模型泛化性能。 |
| “Unlike existing MIL methods that use single-batch training modes, we propose balanced-batch sampling to effectively use the feature loss.” | 不同于以往单包训练,我们提出平衡批次采样,让正负包“同场竞技”。 |
| “A single Transformer block is sufficient to capture long-range dependencies while remaining computationally feasible.” | 单层 Transformer 已能捕获长程依赖,且算力可行。 |
| “Experimental results demonstrate the effectiveness and simplicity of our approach.” | 实验结果验证了我们方法既简单又有效。 |





















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








