Introduction
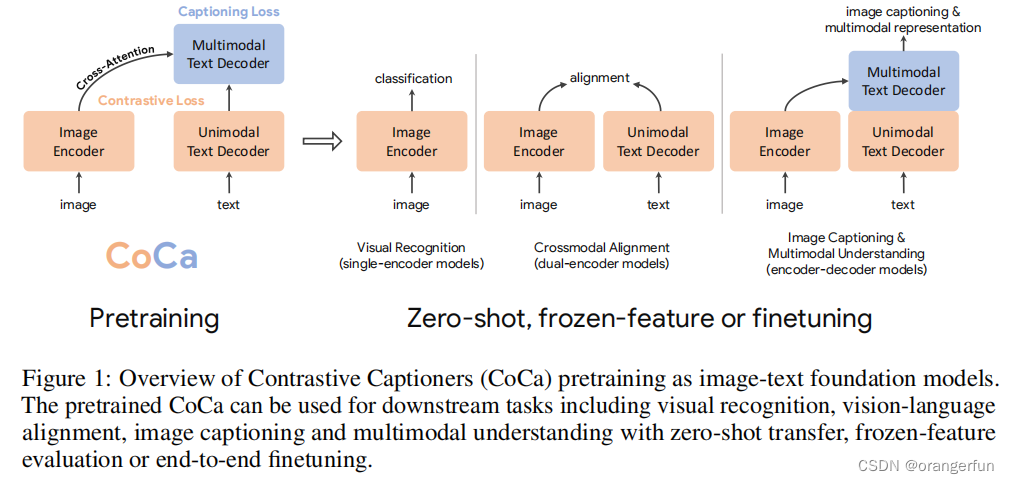
在这项工作中,我们统一了单编码器、双编码器和编码器-解码器范式,并训练了一个包含三种方法优点的图像-文本基础模型。我们提出了对比Captioner模型(CoCa),该模型采用经过对比损失和captioning损失训练的编码器-解码器架构。如图1所示,我们将Transformer解码器解耦为两部分,分别是单模态解码器和多模态解码器。我们省略了单模态解码器中的交叉注意力来编码纯文本表示,并用交叉注意力将图像编码器的输出和多模态解码器级联起来,以学习多模态图像-文本表示。
我们在图像编码器和单模态文本解码器的输出之间应用对比损失,并在多模态解码器的输出处应用captioning损失。此外,通过将所有标签简单地视为文本,我们利用有标签图像数据和带噪声图像-文本数据对CoCa进行了训练。图像注释文本上的生成损失提供了类似于单编码器交叉熵损失的细粒度训练信号,有效地将上述三种预训练范式纳入到统一的方法中。
—翻译自原文
Reated Work
CoCa模型结构整体与ALBEF很相似,此处介绍两者区别
ALBEF模型使用了双编码器结构且结合了对比损失和MLM损失,然而我们的模型能更简单更有效的训练来获得更多的能力:
(1)在每个文本-图片对的batch中,CoCa只执行一次前向和反向传播,然而ALBEF需要执行两次(一次是以随机mask token的形式输入,另一种是正常形式输入)
(2)CoCa仅通过两个目标来训练模型,而ALBEF通过预训练的图像和文本编码器初始化需要包括动量模型在内的额外训练信息
(3)使用生成损失的解码器结构更适合自然语言生成,因此可以直接用于图像描述的zero-shot学习
Approach
用不同自然语言监督的基础图像模型包括如下三种:
- Single-Encoder Classification
通过使用交叉熵分类的形式来进行预训练模型 - Dual-Encoder Contrastive Learning
使用两个编码器分别对图像和文本编码,然后利用对比学习进行联合优化两个编码器,双编码器对比学习形式的优点:能促进文本语义和图像语义对齐,可在与跨模态语义对齐中应用 - Encoder-Decoder Captioning
输入图像,然后自回归的形式生成文字,适用于通过自然语言生成的方式来进行图像描述的应用
论文提出的方法: Contrastive Captioners Pretraining
参考
[1] CoCa: Contrastive Captioners are Image-Text Foundation Models





 本文介绍了一种新型图像-文本基础模型CoCa,它融合了单编码器、双编码器及编码器-解码器的范式优势。通过对比损失和captioning损失训练,该模型能有效生成高质量的图像描述,并支持零样本学习。
本文介绍了一种新型图像-文本基础模型CoCa,它融合了单编码器、双编码器及编码器-解码器的范式优势。通过对比损失和captioning损失训练,该模型能有效生成高质量的图像描述,并支持零样本学习。

















 4396
4396










