本文作者 Taposh Dutta Roy 拥有产品、技术和战略咨询、数据科学和创业经验,是一名以消费者为中心的机器学习和数据科学极客。(我也是第一次听说还有这样的极客,小编以前一直认为极客就是黑客。)
当下深度学习的研究领域仍然停留在自然图像的层面上,本文基于python使用深度学习处理医学图像,提升医疗保健行业的服务水平。
在这篇文章中,作者会从图像处理、医学图像格式方面的基础知识为大家逐渐进行讲解。(文末有彩蛋!)
原文:Medical Image Analysis with Deep Learning — I

基本图像处理知识
开放源代码计算机视觉库(OpenCV)是图像处理库的主流,因为OpenCV有一个较大的团体支持并且其能够支持 C++、java 和 python 等多种编程语言。本文使用 jupyter notebook +OpenCV进行讲解。
先安装好开放源代码计算机视觉库(OpenCV)。
pip install opencv-python 或者直接从 http://opencv.org 官网进行安装。
打开 jupyter notebook,同时确认您可以导入 cv2。这时候,您还需要安装(pip install numpy matplotlib),然后导入矩阵运算库numpy 和画图 matplotlib 包。
(不要试图粘贴复制以下代码啦!这位极客暂时没找到相关开源code,好在简单,自己动手敲键盘吧)

通过 OpenCV 加载的示例图像(注意改下自己的jpg文件路径)

OpenCV 基本人脸检测示例
在检测脸部的过程中,我们需要使用由 Rainer Lienhart 创建的正面检测器,该正面检测器是一种基于 stump 的 20x20 gentle adaboost 算法开源可扩展标记语言。这里有一个关于 Haar-cascade 程序检测的比较不错的帖子,内容非常详细。网址如下:Face Detection using Haar Cascades。
--------------------------------
#First we need to load the required XML classifiers. Then load our input image (or video) in
#grayscale mode.(下划线表示需要注释掉的部分)
import numpy as np
import cv2
face_cascade = cv2.CascadeClassifier('haarcascade_frontalface_default.xml')
eye_cascade = cv2.CascadeClassifier('haarcascade_eye.xml')
img = cv2.imread('sachin.jpg')
gray = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY)
#Now we find the faces in the image. If faces are found, it returns the positions of detected
#faces as Rect(x,y,w,h). Once we get these locations, we can create a ROI for the face and
#apply eye detection on this ROI (since eyes are always on the face !!! ).
faces = face_cascade.detectMultiScale(gray, 1.3, 5)
for (x,y,w,h) in faces:
cv2.rectangle(img,(x,y),(x+w,y+h),(255,0,0),2)
roi_gray = gray[y:y+h, x:x+w]
roi_color = img[y:y+h, x:x+w]
eyes = eye_cascade.detectMultiScale(roi_gray)
for (ex,ey,ew,eh) in eyes:
cv2.rectangle(roi_color,(ex,ey),(ex+ew,ey+eh),(0,255,0),2)
cv2.imshow('img',img)
cv2.waitKey(0)
cv2.destroyAllWindows()
---------------------------------------
使用 OpenCV 进行脸部检测,是不是很方便。

下面网址链接中的在线文档中有很多使用 OpenCV 进行图像处理的例子:OpenCV-Python Tutorials。现在我们已经知道了图像处理的基础知识,下面 了解一下医学图像格式的基础知识吧。
医学图像数据格式
用数字成像和通信(DICOM)追踪医学图像已经成为存储和交换医学图像数据的标准解决方案。该标准的第一个版本发布于1985年,内容后来有些许的变动。该标准使用的格式包括文件格式和通信协议。
文件格式。所有病人的医学图像均以数字成像和通信(DICOM)的格式进行保存。保存的文件格式中含有关于患者的受保护的健康信息(PHI),包括姓名、性别、年龄以及其他与图像相关的数据,例如用于捕获图像的设备和一些关于医疗背景的资料等。医疗影像设备创建 DICOM 文件。计算机软件应用程序能够显示 DICOM 图像,医生可以通过使用 DICOM 查看器来查看图像并读取、诊断图像中的结果。
通信协议。DICOM 通信协议用于在档案中搜索成像研究,并将找到的成像研究恢复到工作站中并将其显示出来。连接到医院网络的所有医学成像应用程序均使用 DICOM 协议进行信息交换,主要包括 DICOM 图像,同时也包括患者以及手术信息等。此外还有更先进的网络命令,主要用于控制和追踪治疗进度、调度程序、报告状态,并且能在医生和成像设备之间实现工作负荷共享。
这里有一篇非常好的博客,其详细介绍了DICOM标准。网址如下:http://dicomiseasy.blogspot.com/。
分析 DICOM 图像
Pydicom 是一个非常好的用于分析 DICOM 图像的 Python 软件包。在本节中,我会向大家介绍如何在 Jupyter notebook 上呈现 DICOM 图像。pip install pydicom。安装 pydicom 软件包之后,回到 Jupyter notebook。在笔记本中输入 dicom 软件包和其他软件包,如下图所示。

我们还需要使用其他的软件包进行数据处理和数据分析,包括 pandas、scipy、skimage、mpl_toolkit 等软件包(需要事先安装好,这里就不再赘述)。

目前网上有很多可以免费使用的 DICOM 数据集,以下几个可以帮助您开启使用 DICOM 数据集的道路:
Kaggle 竞争数据集(Kaggle Competitions and Datasets,Your Home for Data Science):这是我个人最喜欢的数据集。该数据集能够帮助你找到肺癌方面的竞赛以及糖尿病、视网膜病变等方面的资料。
Dicom 图书馆(Dicom Library,Anonymize, Share, View DICOM files ONLINE):DICOM 图书馆是一个免费的在线医疗 DICOM 图像或视频文件共享服务数据集,主要用于教育和科研领域。
Osirix 数据集(Osirix Datasets,DICOM Image Library):提供通过各种成像模式获取的大量的人类数据集。
可视人类数据集(Visible Human Datasets,Visible Human Project Datasets):从某种角度上来看,可视人类项目的其中一些部分是可以自由获取的,但奇怪的是,其中的数据的获取既不免费也不容易。
Zubal 幻影(The Zubal Phantom,the Zubal Phantom):该网站提供了两个男人在 CT 和 MRI 中的大量的数据集,这些数据集都是免费的。
下载 dicom 文件并将其加载到您的 jupyter notebook 上。

现在,将 DICOM 图像加载到列表中。

步骤1:在 Jupyter中浏览基本 DICOM 图像

在第一行中我们加载第一个 DICOM 文件,将其命名为 RefD,我们将它作为参考以提取元数据,元数据在 lstFilesDCM 列表的文件名称是 first。

然后,我们计算 3D NumPy 数组(3D NumPy array)的总尺寸,其等于沿着笛卡尔轴 x、y、z 的数据的乘积,即“切片中的像素行数×切片中的像素列数×切片数”。最后,我们使用像素间距和切片厚度的属性来计算三个轴之间的像素间距。最终我们将 ConstPixelDims 中的数组维数和 ConstPixelSpacing 中的间距数据存储起来。


步骤2:查看 DICOM 格式的细节
CT 扫描的测量单位是 Hounsfield 单位(HU),主要用来度量辐射强度。CT 扫描仪经过精密的校准可以准确地进行测量工作。点击以下网址可以看到关于 CT 扫描仪的详细信息:https://web.archive.org/web/20070926231241/http://www.intl.elsevierhealth.com/e-books/pdf/940.pdf。
每个像素都分配有一个数值(CT 号),该数值是包含在相应像素中的所有衰减值的平均值。Godfrey Hounsfield 先生曾将分配的数值与水的衰减值进行比较,自那以后所有的数据均以 Hounsfield 为单位进行命名,并且在 Hounsfield 量表有所体现。
此 Hounsfield 量表将水的衰减值分配为零(HU)。CT 数据的范围是 2000HU,不过一些现代扫描仪的 HU 范围已经上升到了 4000 HU。每个数字代表了在光谱两端有+1000(白色)和-1000(黑色)的灰色阴影。

Hounsfield 量表[量表值:《物理学导论》(PDF).。elsevierhealth.com.]
一些扫描仪具有圆柱形扫描边界,但输出的图像是正方形的。其中,落在边界之外的像素都为固定的值,这个固定值为-2000 HU。
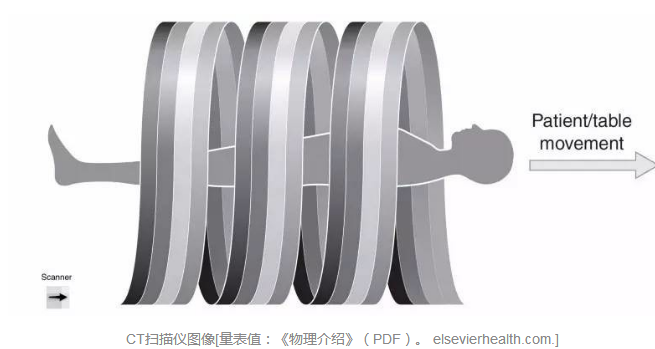
CT扫描仪图像[量表值:《物理介绍》(PDF)。 elsevierhealth.com.]
第一步通常是将这些数值设置为0。接下来,通过乘以重新调整的斜率并添加截距使我们回到HU单元(截距方便地存储在扫描的元数据中!)。
接下来的部分,作者将会运用 Kaggle 的肺癌数据集以及利用了 Keras 的卷积神经网络。欢迎关注下篇。
感谢知友,ID周生,提供的类似参考资料:Guido Zuidhof的Full Preprocessing Tutorial。





 本文介绍了使用深度学习处理医学图像的基本方法,包括图像处理基础知识、医学图像格式介绍及使用Python进行图像分析的具体实践。
本文介绍了使用深度学习处理医学图像的基本方法,包括图像处理基础知识、医学图像格式介绍及使用Python进行图像分析的具体实践。
















 1519
1519










