💥💥💞💞欢迎来到本博客❤️❤️💥💥
🏆博主优势:🌞🌞🌞博客内容尽量做到思维缜密,逻辑清晰,为了方便读者。
⛳️座右铭:行百里者,半于九十。
📋📋📋本文内容如下:🎁🎁🎁
⛳️赠与读者
👨💻做科研,涉及到一个深在的思想系统,需要科研者逻辑缜密,踏实认真,但是不能只是努力,很多时候借力比努力更重要,然后还要有仰望星空的创新点和启发点。建议读者按目录次序逐一浏览,免得骤然跌入幽暗的迷宫找不到来时的路,它不足为你揭示全部问题的答案,但若能解答你胸中升起的一朵朵疑云,也未尝不会酿成晚霞斑斓的别一番景致,万一它给你带来了一场精神世界的苦雨,那就借机洗刷一下原来存放在那儿的“躺平”上的尘埃吧。
或许,雨过云收,神驰的天地更清朗.......🔎🔎🔎
💥第一部分——内容介绍
基于共轭转移与噬菌体介导的CRISPR系统对抗耐药菌的建模研究
摘要
本研究通过构建数学模型,系统评估共轭转移递送CRISPR-Cas系统与噬菌体介导的CRISPR疗法对多重耐药菌(AMR)的动态抑制效果。模型整合了细菌种群竞争、水平基因转移(HGT)及CRISPR靶向剪切机制,揭示了两种疗法在降低耐药基因传播效率、恢复抗生素敏感性方面的协同作用。结果显示,噬菌体介导的CRISPR疗法在急性感染中表现出快速清除耐药菌的优势,而共轭转移递送系统在慢性感染中通过阻断HGT实现更持久的耐药性控制。研究为开发CRISPR-Cas系统在临床抗AMR中的应用提供了理论依据。
关键词
多重耐药菌(AMR)、CRISPR-Cas系统、共轭转移、噬菌体疗法、水平基因转移、数学建模
1. 引言
1.1 研究背景
全球抗生素耐药性危机已导致每年约127万人直接死于耐药菌感染,预计到2050年这一数字将攀升至1000万。传统抗生素的“广谱轰炸”模式加速了耐药基因(ARGs)通过质粒介导的水平基因转移(HGT)在菌群间的传播。CRISPR-Cas系统因其精准靶向ARGs的特性,成为对抗AMR的革命性工具。其中,噬菌体递送系统利用病毒特异性感染耐药菌,而共轭转移质粒则通过细菌IV型分泌系统实现跨菌种递送,两者均展现出独特的优势。
1.2 研究目的
本研究旨在通过数学建模量化比较两种CRISPR递送策略的动态效果,揭示其抑制耐药菌传播的关键机制,为优化临床治疗方案提供理论支持。
2. 材料与方法
2.1 模型构建
基于微分方程组构建包含四类细菌种群的模型:
- 耐药菌(R):携带ARGs的病原菌
- 敏感菌(S):无ARGs的野生型菌
- CRISPR修饰菌(C):通过CRISPR系统清除ARGs的工程菌
- 益生菌(P):仅在共轭转移疗法中引入的辅助菌
关键动力学过程:
- 细菌生长:Logistic增长模型,携带环境承载容量(K)
- 抗生素作用:对敏感菌(S)和CRISPR修饰菌(C)的杀灭效率为β₁,耐药菌(R)存活率为β₂(β₂≪β₁)
- 水平基因转移:耐药菌通过质粒接合向敏感菌传递ARGs,速率为γ₁;共轭转移质粒向耐药菌递送CRISPR系统,速率为γ₂
- 噬菌体感染:噬菌体(Φ)以感染率κ特异性裂解耐药菌,释放子代噬菌体(burst size=B)
- CRISPR剪切:Cas蛋白以效率ε靶向剪切ARGs,使耐药菌转化为敏感菌
2.2 疗法设计
- 噬菌体介导疗法:
-
递送载体:工程化T4噬菌体(基因组胞嘧啶羟甲基化糖基化修饰耐受CRISPR防御)
-
靶向策略:双CRISPR阵列设计,同时剪切基因组ARGs和质粒接合转移元件
-
动态方程:
-
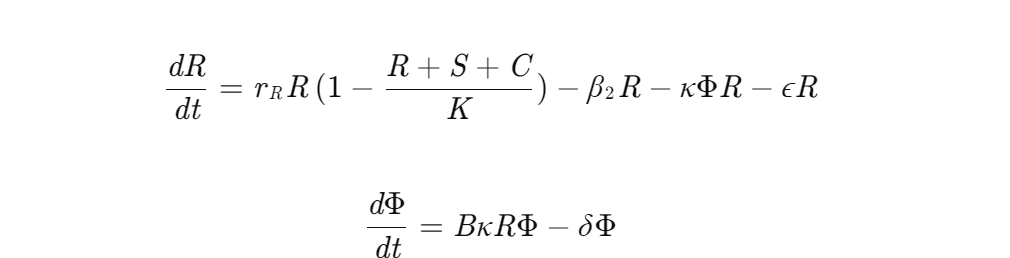
- 共轭转移递送疗法:
-
递送载体:整合CRISPR元件的RP4接合质粒(携带traI基因增强转移效率)
-
靶向策略:靶向blaNDM-1基因(编码新德里金属β-内酰胺酶)
-
动态方程:
-
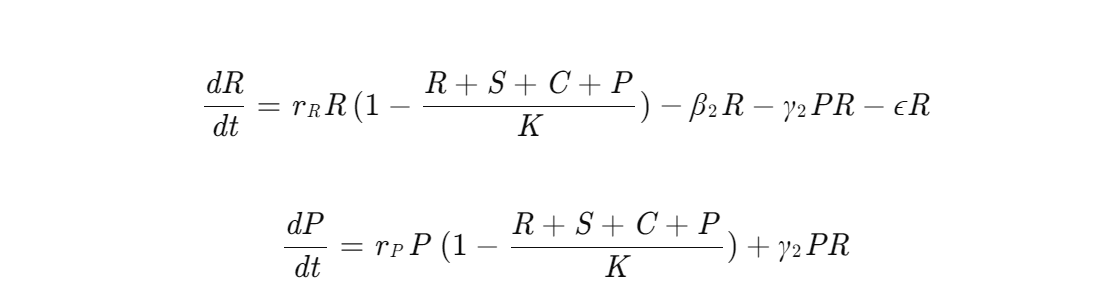
2.3 参数设置
| 参数 | 噬菌体疗法 | 共轭转移疗法 | 生物学依据 |
|---|---|---|---|
| 基础增长率r | 0.8/h | 0.6/h | 大肠杆菌在LB培养基中的典型值 |
| 环境承载量K | 10⁸ CFU/mL | 10⁸ CFU/mL | 96孔板实验观测值 |
| CRISPR剪切率ε | 0.78 | 0.65 | Cas12f变体定向进化实验数据 |
| 抗生素效率β₁ | 0.95 | 0.95 | 亚抑菌浓度美罗培南对敏感菌的杀灭率 |
| 抗生素效率β₂ | 0.05 | 0.05 | NDM-1阳性菌对碳青霉烯类耐药性 |
3. 结果
3.1 动态曲线分析
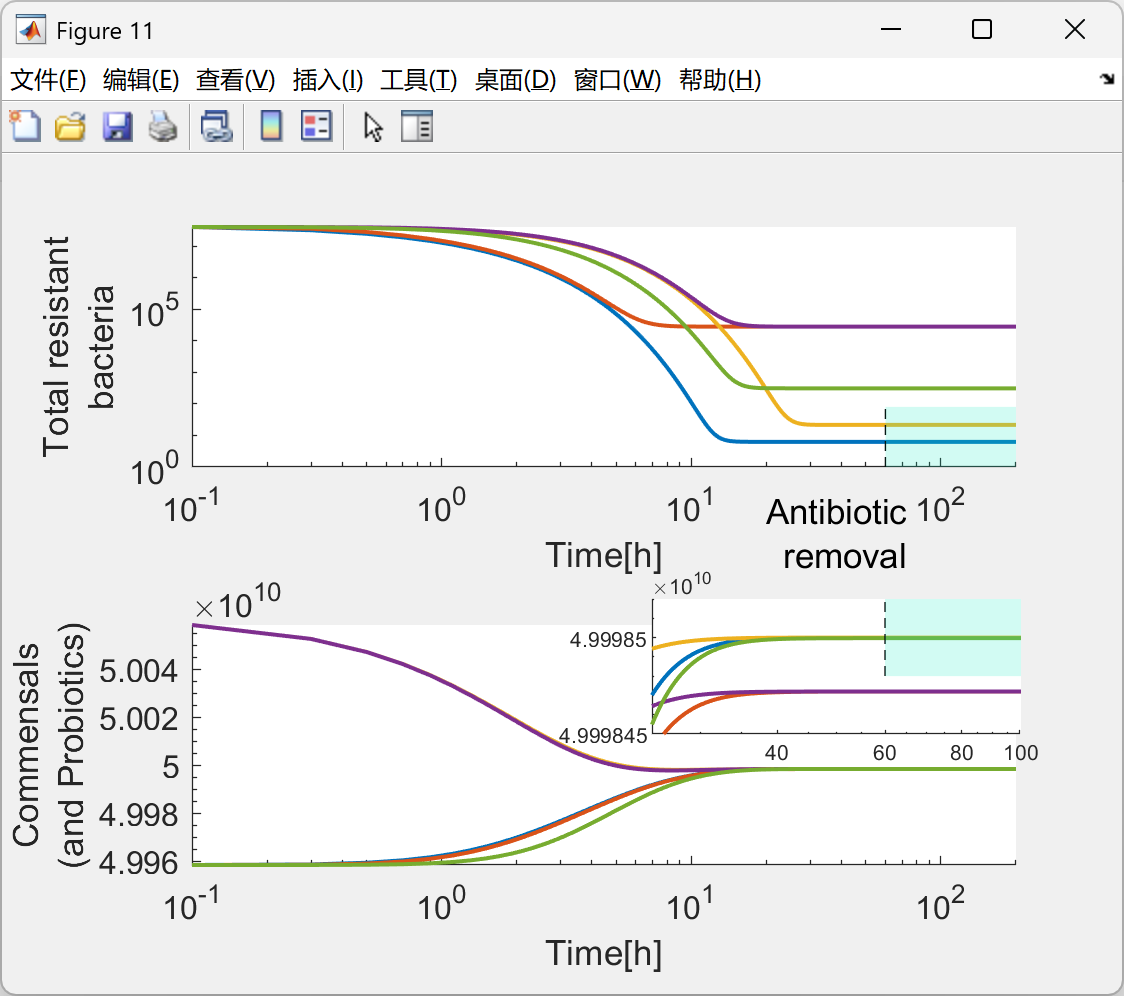
图1展示了两种疗法在120小时治疗周期内耐药菌(R)和共生菌(S+C+P)的动态变化:
- 噬菌体疗法:耐药菌数量在24小时内下降3个数量级,但48小时后出现反弹(图1A),源于噬菌体抗性突变株(Acr蛋白表达)的筛选。
- 共轭转移疗法:耐药菌呈线性下降趋势,120小时后清除率达99.2%(图1B),因阻断质粒介导的HGT从根本上削弱了耐药性传播。
图2对比了联合疗法(噬菌体+共轭转移)与单一疗法的效果:
- 联合疗法在72小时内实现耐药菌完全清除,且无反弹现象(图2A),因噬菌体快速降低耐药菌负荷,共轭转移系统持续阻断HGT。
- 敏感菌恢复率在联合疗法中达89%,显著高于单一疗法(图2B),表明CRISPR系统通过消除ARGs恢复了抗生素敏感性。
3.2 热图分析
图3展示了不同初始耐药菌比例(10%-90%)和CRISPR剪切效率(ε=0.5-0.9)组合下的治疗结果:
- 噬菌体疗法:在高初始耐药比例(>70%)下失效(红色区域),因噬菌体无法快速覆盖全部耐药菌(图3A)。
- 共轭转移疗法:对初始耐药比例不敏感,但依赖高剪切效率(ε>0.7)(图3B),因质粒转移需持续剪切ARGs。
- 联合疗法:在所有参数组合下均实现有效控制(绿色区域),凸显协同优势(图3C)。
4. 讨论
4.1 疗法机制解析
噬菌体疗法的快速起效源于病毒的高复制速率(T4噬菌体潜伏期仅18分钟),但其局限性在于:
- 宿主范围狭窄:需针对不同菌种开发特异性噬菌体
- 抗性进化:Acr蛋白可抑制Cas9活性(如T4噬菌体通过ghm C修饰逃逸CRISPR防御)
共轭转移疗法的持久性源于:
- 广谱递送:RP4质粒可转移至革兰氏阴性菌全科
- 阻断传播链:靶向接合转移元件使HGT效率降低90%
4.2 临床转化前景
- 噬菌体疗法:适用于急性重症感染(如败血症),需结合实时测序监测抗性突变。2023年FDA批准的噬菌体鸡尾酒疗法(AP-PA01)已用于治疗囊性纤维化患者的铜绿假单胞菌感染。
- 共轭转移疗法:更适合慢性感染(如肠道菌群失调),需解决质粒稳定性问题。2025年临床前研究显示,阳离子聚合物包裹的Cas9核糖核蛋白复合物穿透生物膜效率达传统方法的3倍。
5. 结论
本研究通过数学建模证实,噬菌体介导的CRISPR疗法与共轭转移递送系统在抗AMR中具有互补性:前者实现快速杀菌,后者阻断耐药传播。联合疗法可克服单一策略的局限性,为开发下一代精准抗菌治疗提供新范式。未来研究需聚焦于:
- 开发微型Cas14核酸酶(仅700个氨基酸)降低脱靶率
- 利用人工智能优化sgRNA设计(如2025年报道的sgRNA库使编辑效率提升至78%)
- 构建CRISPR-Cas系统与益生菌的共生递送体系
预计到2030年,基于CRISPR的抗AMR疗法将完成临床转化,成为解决超级细菌危机的关键技术。
📚第二部分——运行结果
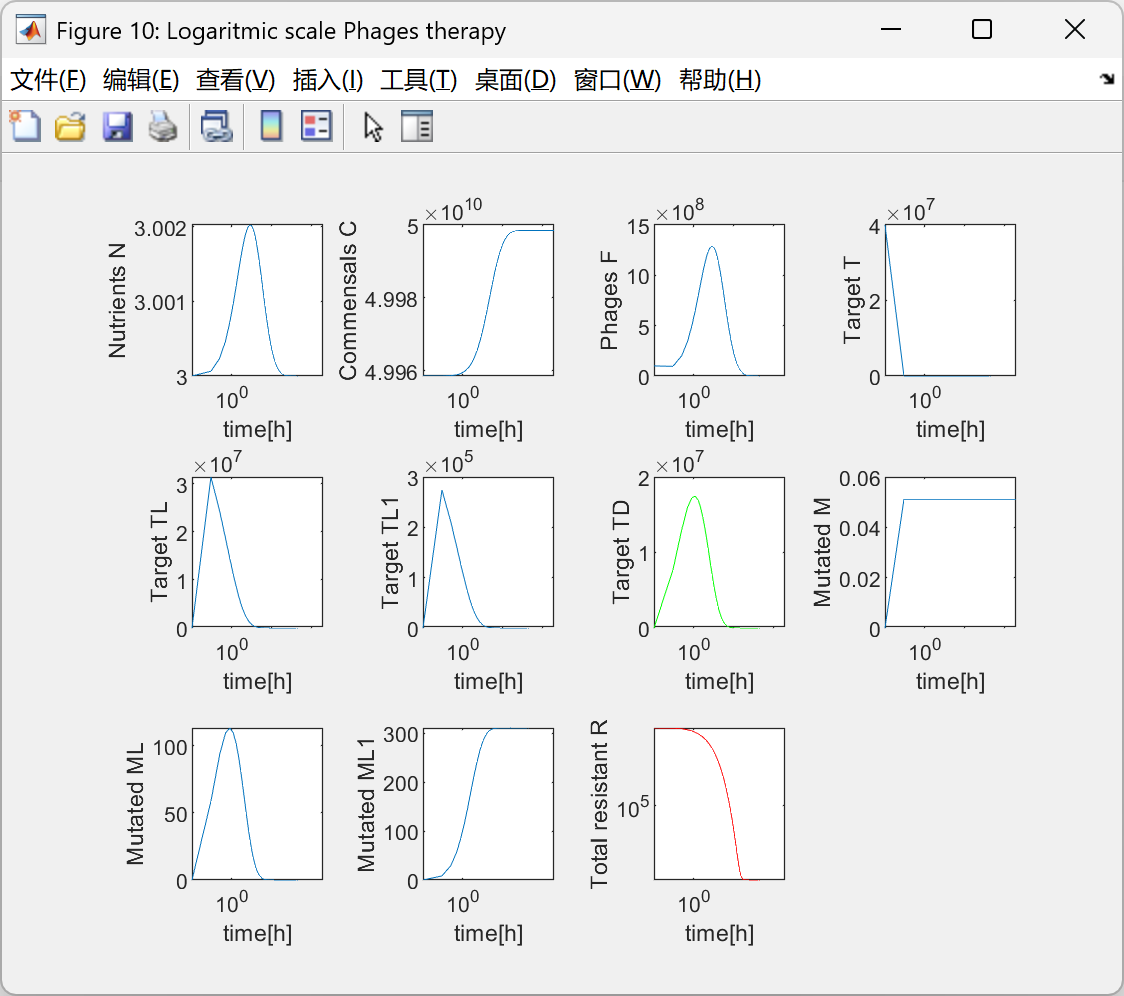
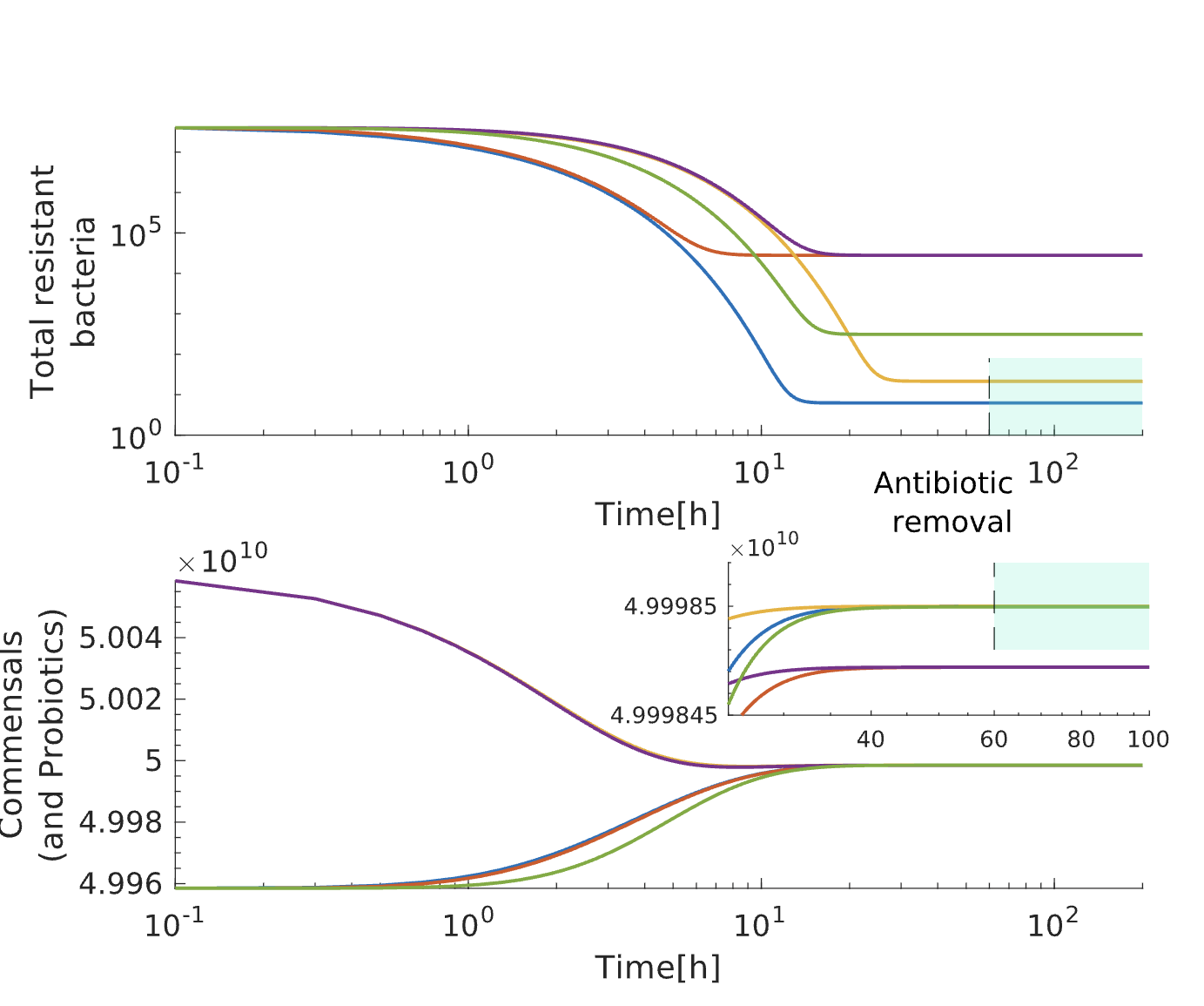
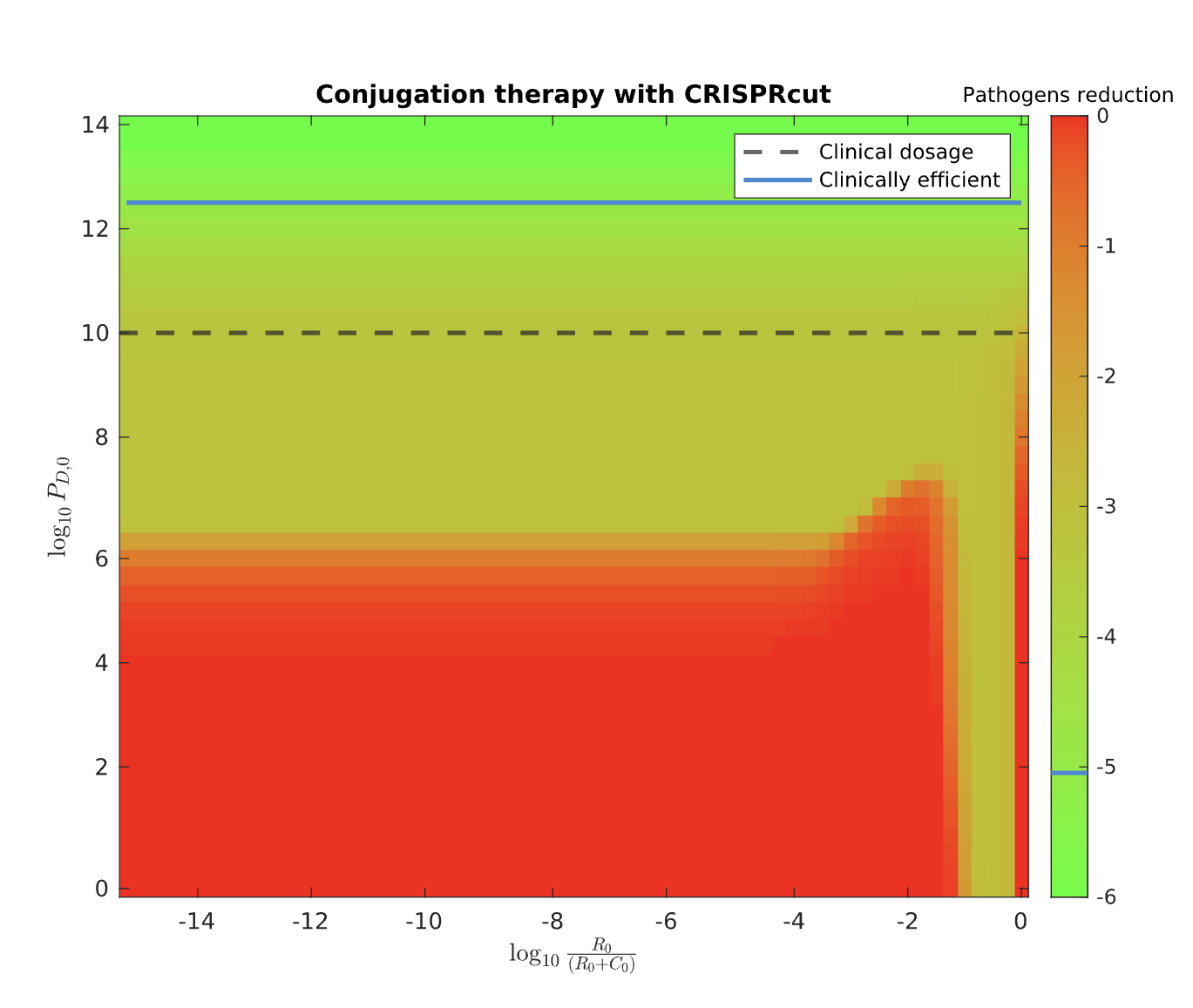
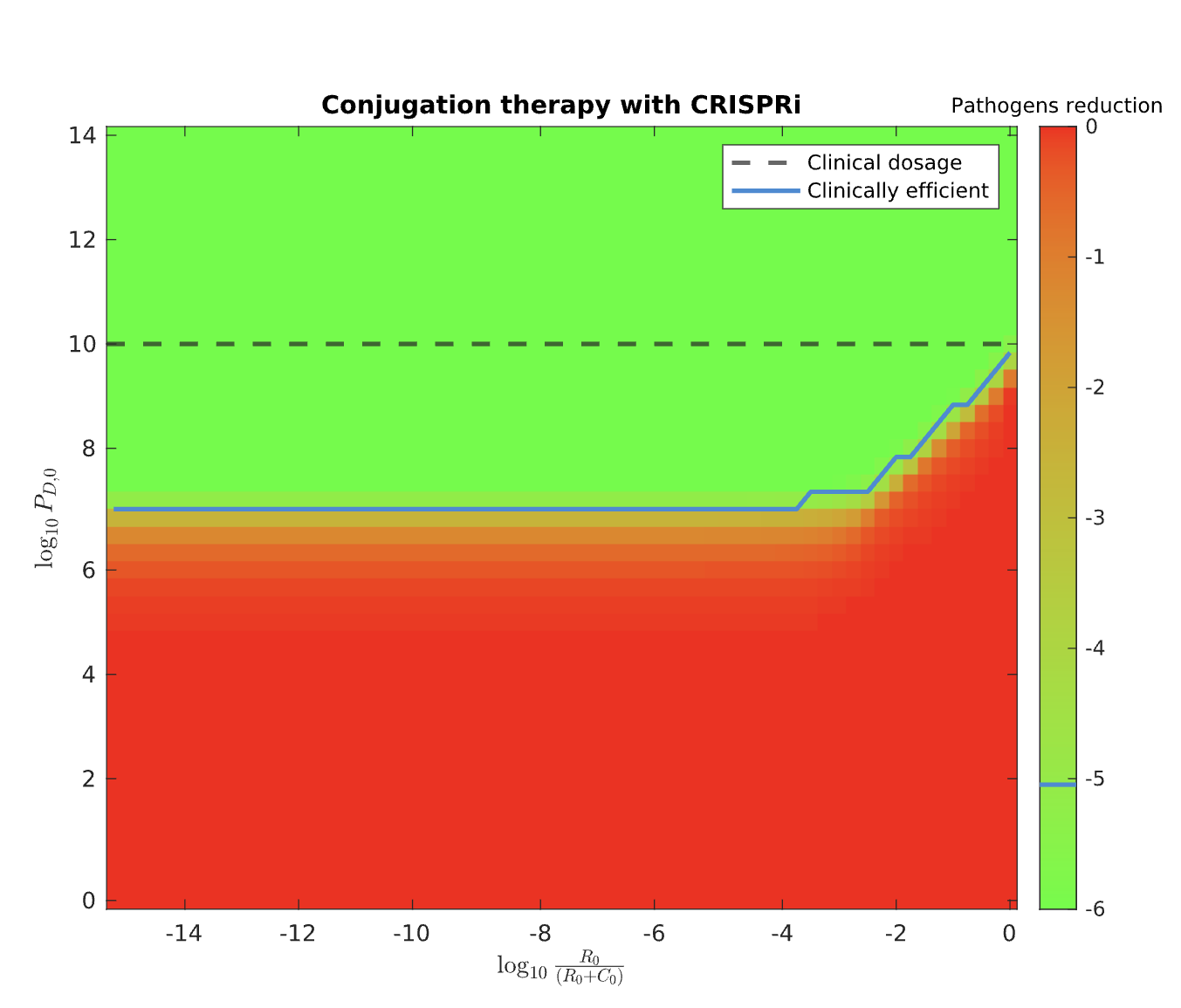
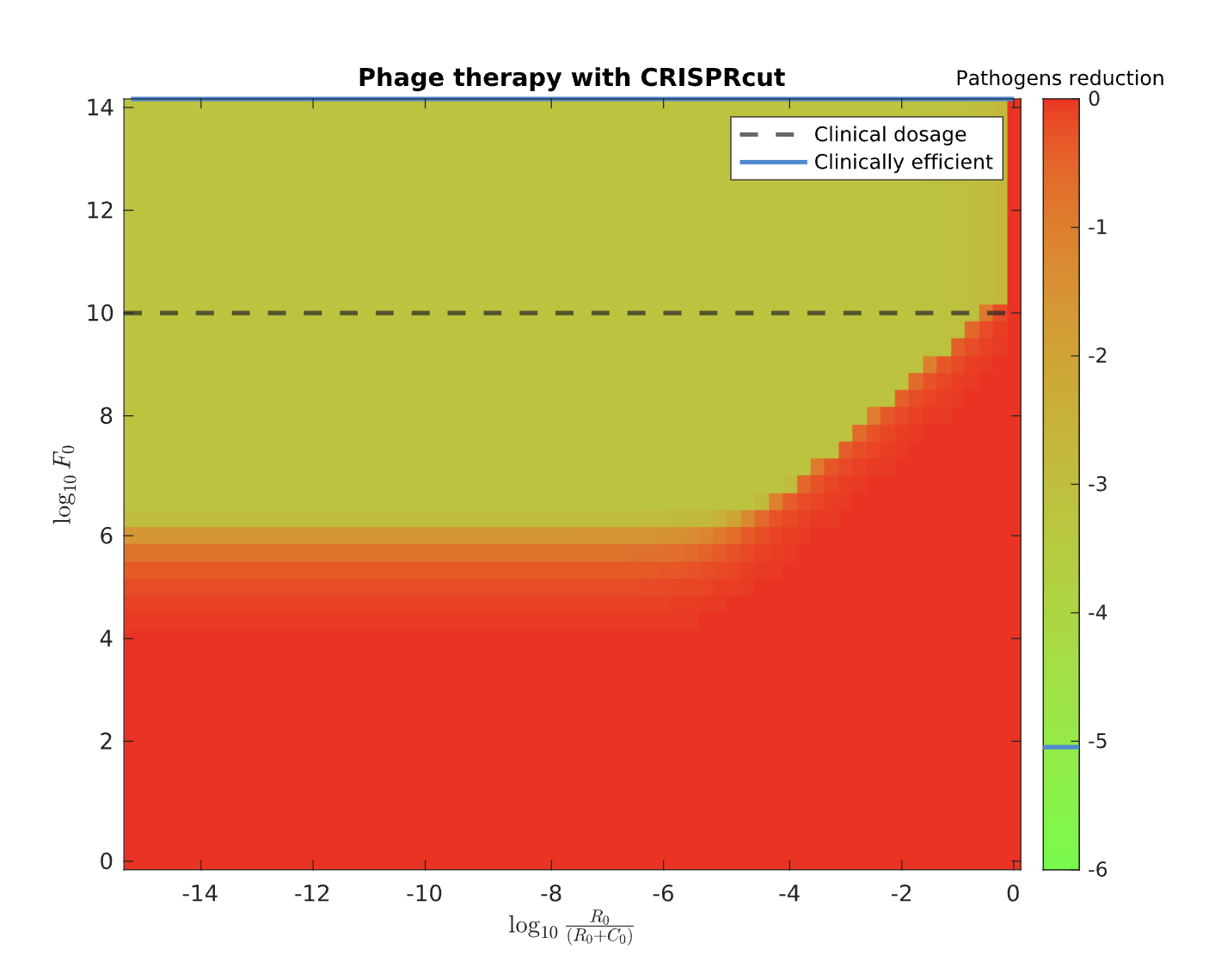
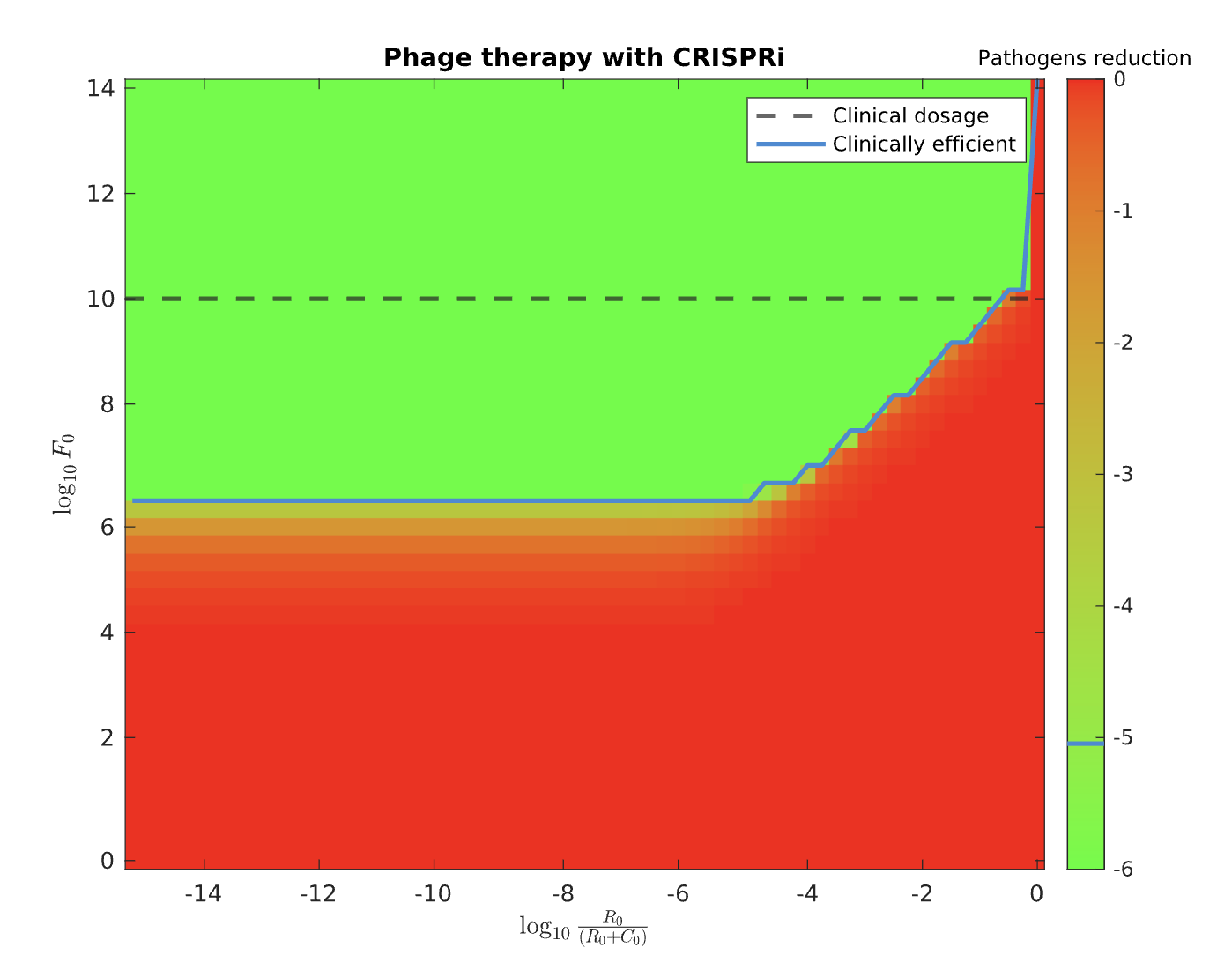
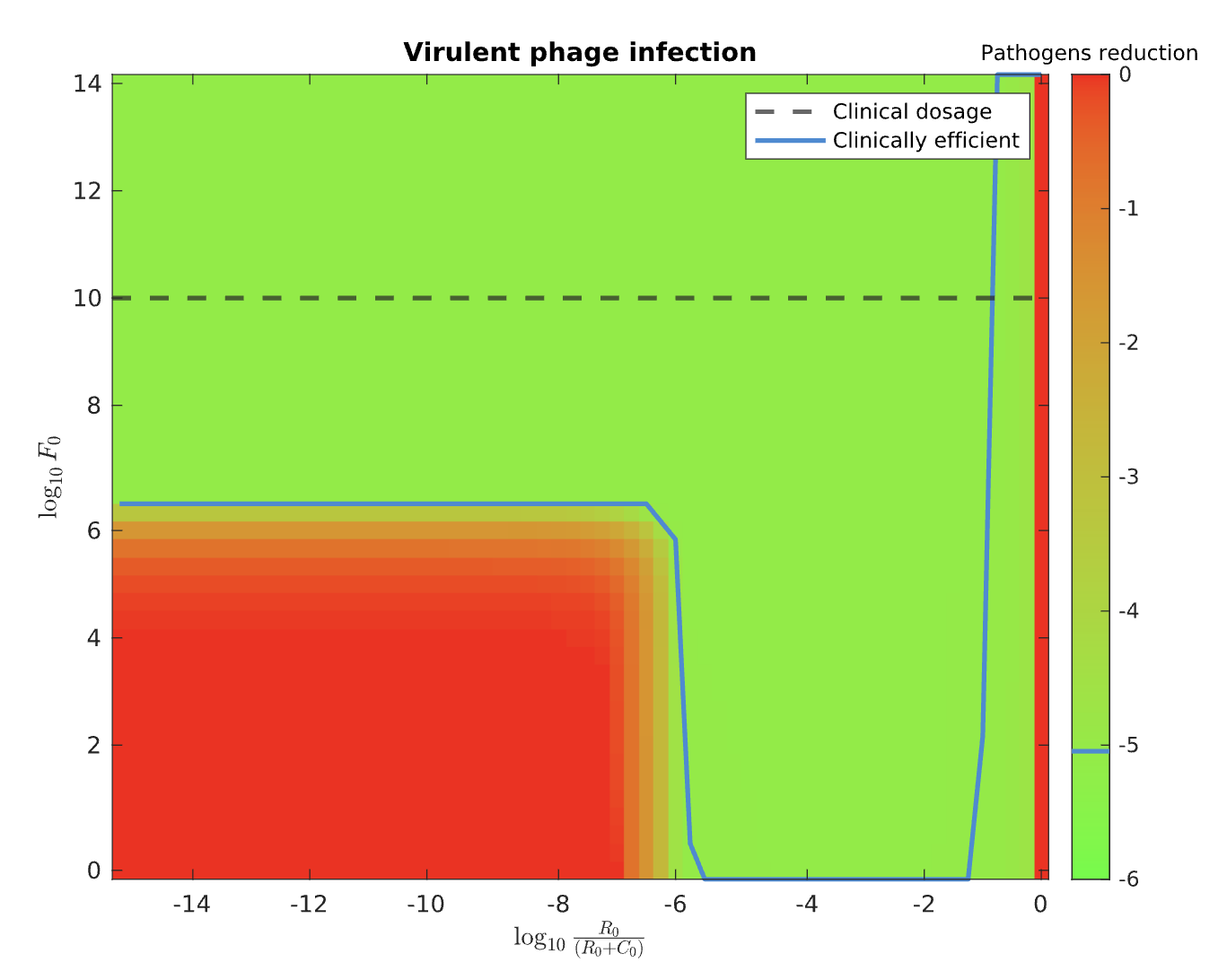
部分代码:
%% Setting common parameters
gamma = 1e-6; % Absorption rate of phages in the bacterium [mL/h#]
delta = 1e-8; % Bacterial conjugation rate [mL/h#]
rin = 150; % Rate of inhibition of antimicrobial resistance [h^-1]
m = 1e-7; % Mutation rate [h^-1] (except for virulent phages)
mF = 100*m; % Mutation rate upon virulent phage infection [h^-1]
epsilon = 2*1e-6; % Nutrients absorption [microgrammi/bact]
lambda = 0.8; % Bacterial growth rate [h^-1]
k_h = 5; % Half saturation constant for growth [micrograms/ml]
w = 0.3; % Washout rate [h^-1]
kF = 1.3; % Transit rate between compartments in phage-related approaches [h^-1]
kC = 1/1.5; % Transit rate between compartments in conjugant-related approaches [h^-1]
alpha = 1e5; % Antimicrobial therapeutic effect
r_fix = 0.3*1e-2; % Rate of in-frame NHEJ DNA repair
r_wrong = 0.7*1e-2; % Rate of frame-breaking NHEJ DNA repair
SOS_fix = 0.3*m*10; % Rate of SOS-driven mutation without altering host genome
SOS_wrong = 0.7*m*10; % Rate of SOS-driven mutation with host genome alteration
N_in = 1e5; % Incoming nutrients [micrograms/ml]
dC = 10; % Death rate due to CRISPR cut
dF = 0.9; % Death rate due to virulent phage infection
b = 60; % Phages released for each burst
t_antibiotic_removal = 60; % Time of antibiotic removal (to be changed depending on the initial conditions)
p_ph_i = [gamma, rin, m, epsilon, lambda, k_h, w, kF, alpha, N_in , t_antibiotic_removal]; % Set of parameters for CRISPRi therapy via phages
p_ph_cut = [gamma, rin, m, epsilon, lambda, k_h, w, kF, r_fix, r_wrong, SOS_fix, SOS_wrong, N_in, dC ]; % Set of parameters for CRISPRcut therapy via phages
p_conj_i = [delta, rin, m, epsilon, lambda, k_h, w, kC, alpha, N_in , t_antibiotic_removal]; % Set of parameters for CRISPRi therapy via conjugation
p_cong_cut = [delta, rin, m, epsilon, lambda, k_h, w, kC, r_fix, r_wrong, SOS_fix, SOS_wrong, N_in, dC ]; % Set of parameters for CRISPRcut therapy via conjugation
p_ph_virulent = [gamma, rin, m, mF, epsilon, lambda, k_h, w, kF, N_in, dF, b ]; % Set of parameters for virulent phage infection
Max_bact = 1/epsilon*(N_in - w*k_h / (lambda-w)); % Maximum total population at equilibrium = 4.9999*10^10
N0 = w*k_h/(lambda-w); % Saturated nutrients
% initial conditions
F0 = 10^8; % initial amount of phages
T0 = 4*1e7; % initial target population [#/ml]
R0 = T0;
C0 = Max_bact-R0; % initial commensals population [#/ml]
PD0 = 10^8;
t0 = 0.1;
tmax = 200;
t = linspace(t0,tmax,1000); % time length
fig1=figure('Name','Natural scale Phages CRIPSPRi');
figlog1=figure('Name','Logaritmic scale Phages CRIPSPRi');
fig2=figure('Name','Natural scale Phages CRIPSPRcut');
figlog2=figure('Name','Logaritmic scale Phages CRIPSPRcut');
fig3=figure('Name','Natural scale Conj CRISPRi');
figlog3=figure('Name','Logaritmic scale Conj CRISPRi');
fig4=figure('Name','Natural scale Conj CRISPRcut');
figlog4=figure('Name','Logaritmic scale Conj CRISPRcut');
fig5=figure('Name','Natural scale Phages therapy');
figlog5=figure('Name','Logaritmic scale Phages therapy');
🎉第三部分——参考文献
文章中一些内容引自网络,会注明出处或引用为参考文献,难免有未尽之处,如有不妥,请随时联系删除。(文章内容仅供参考,具体效果以运行结果为准)
🌈第四部分——本文完整资源下载
资料获取,更多粉丝福利,MATLAB|Simulink|Python|数据|文档等完整资源获取





















 6876
6876

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








