本人是win11,薛定谔版本是12.9。
官网:https://www.schrodinger.com/
本篇文章的示例大分子蛋白PDB ID为4KNN,小分子配体的MOL ID为MOL004004。
1.Protein Preparation Wizard讲解(可跳过)
在右上角的操作区的tasks搜索Protein Preparation Wizard,打开面板如下:
以下图源部分来自知乎https://zhuanlan.zhihu.com/p/416698194。

1.1.import and process


选项7:其实就是添加二硫键(共价键)。
选项9:其实就是补全残基。
选项10:对于某些结合口袋在loop区的蛋白,需要修复loop,但这一步比较耗费时间。
选项12:就是删除保守水分子之外的水分子。
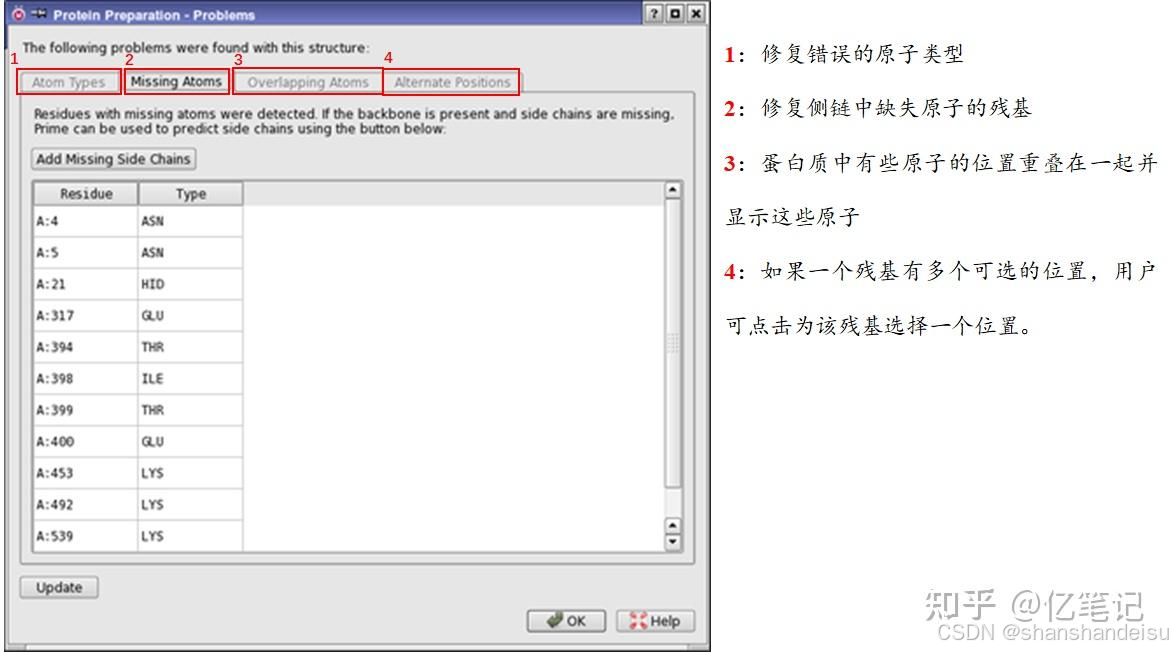
1.1.1.Problems
结束预处理这一步会弹出一个新的problems面板,告诉我们可能出现一些问题需要我们解决。分别是:
- Atom Types:原子类型错配
- Missing Atoms:原子缺失
- Overlapping Atoms:原子重叠
- Alternate Positions:原子位置交替
1.1.1.1.Atom Types
暂略
1.1.1.2.Missing Atoms

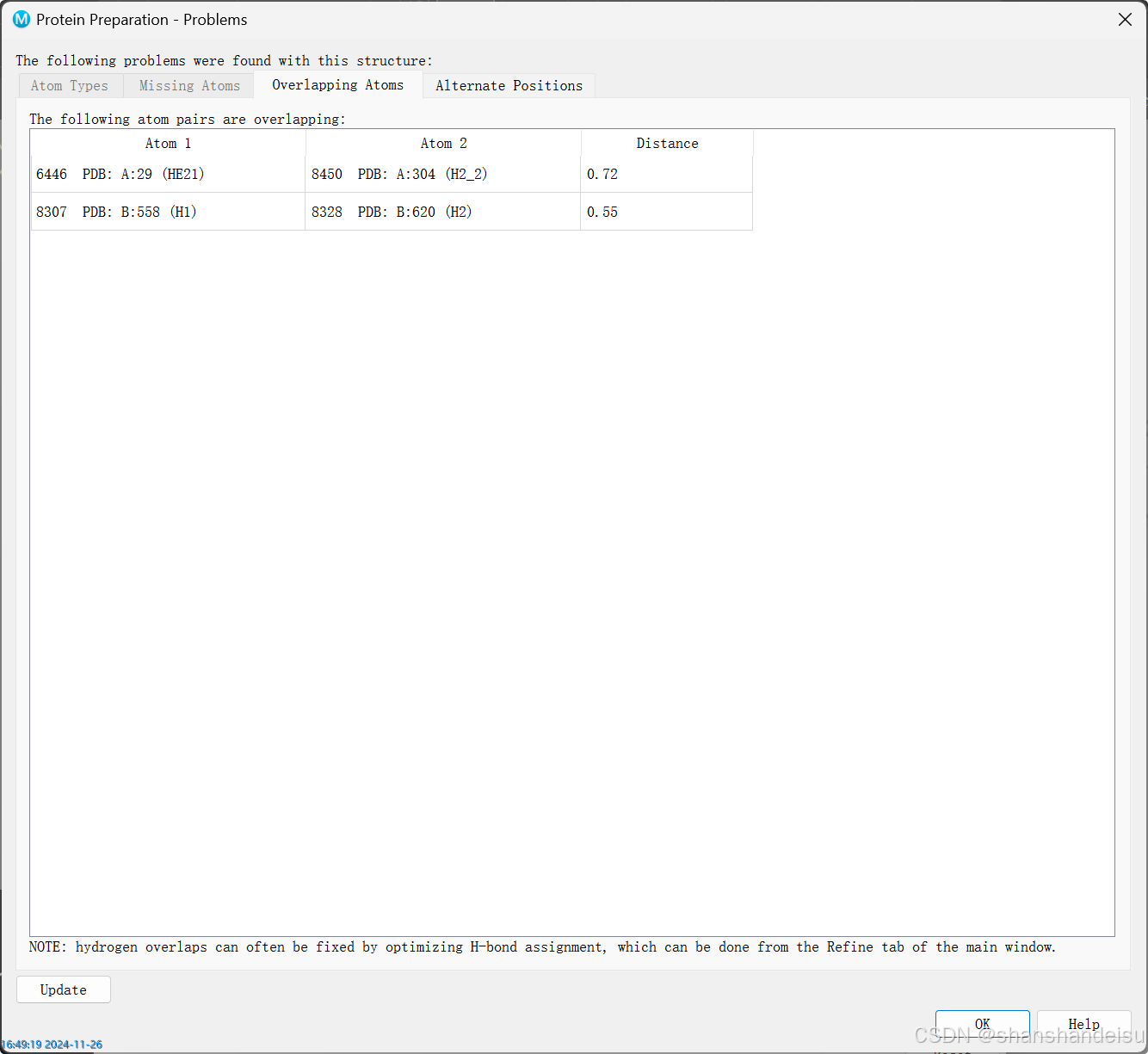
1.1.1.3.Overlapping Atoms
不用理会,后面能量最小化的时候软件会尽可能修复,直接ok即可。
氢原子之间比较容易出这个问题。
1.1.1.4.Alternate Positions
说明当前有两个残基位置不确定。其电子云密度图的occupancy不是1,所以代表着不确定性。因为溶剂中的蛋白是在运动的,所以可能在两个构象中变化。
点击next position可以跳到下个构象,但是做对接时需要确定。
我们看下离binding_pocket是否遥远,如果比较远的话就随便挑一个。点击commit之后就会发现主界面只保留了一个构象。
但是如果你有很多条目需要确定,如下,那么直接点ok就好了/(ㄒoㄒ)/~~

1.2.review and modify
一般最开始在层级结构那里将Ligands和Others和Solvents/other solvents都删除,就不会出这个问题。

1.3.refine

选项2:利用晶体结构数据(通常是利用晶体的对称性来简化计算)。
选项3:对经过修改的分子(例如添加或修改了氢原子的部分)进行氢原子位置的最小化。
点击optimize开始优化。
以下是高级设置。

点击minimize进行能量最小化。

2.Protein Preparation Wizard实战
首先在层级结构那里将Ligands和Others和Solvents/other solvents都删除(注意保留Solvents/waters,后续使用import and process自动识别保守水分子并删除)
2.1.import and process
勾选fill in missing side chains using Prime后,选择preprocess,运行结束后工作区多了一个preprocessed的条目。

2.1.2.Problems
具体见1.1.1.Problems的处理
2.2.review and modify
一般最开始在层级结构那里将Ligands和Others和Solvents/other solvents都删除,就不会出这个问题。所以掠过
2.3.refine
保持默认选项,运行Optimize后,工作区出现hbond-opt的条目。
接着运行Minimize,工作区出现minimized的条目。

2.4.总结
最后整个工作区应该有原始蛋白、preprocessed、hbond-opt、minimized四个条目。
且观察结构层级,蛋白还有哪些(我这因为没有保守水分子,所以只有蛋白的氨基酸本身)






























 8616
8616

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










