对于GWAS分析初学者Gapit包是十分简单和易学,需要提前安装好R和Rstudio.
文件准备
首先,需要提前准备好基因型文件和表型文件即可,
基因型文件准备位×××.hmp.txt的格式,表型 txt,csv,xlsx都可以;
如果基因型文件是VCF文件,可以通过Tassel软件读入,再另存为hmp格式,
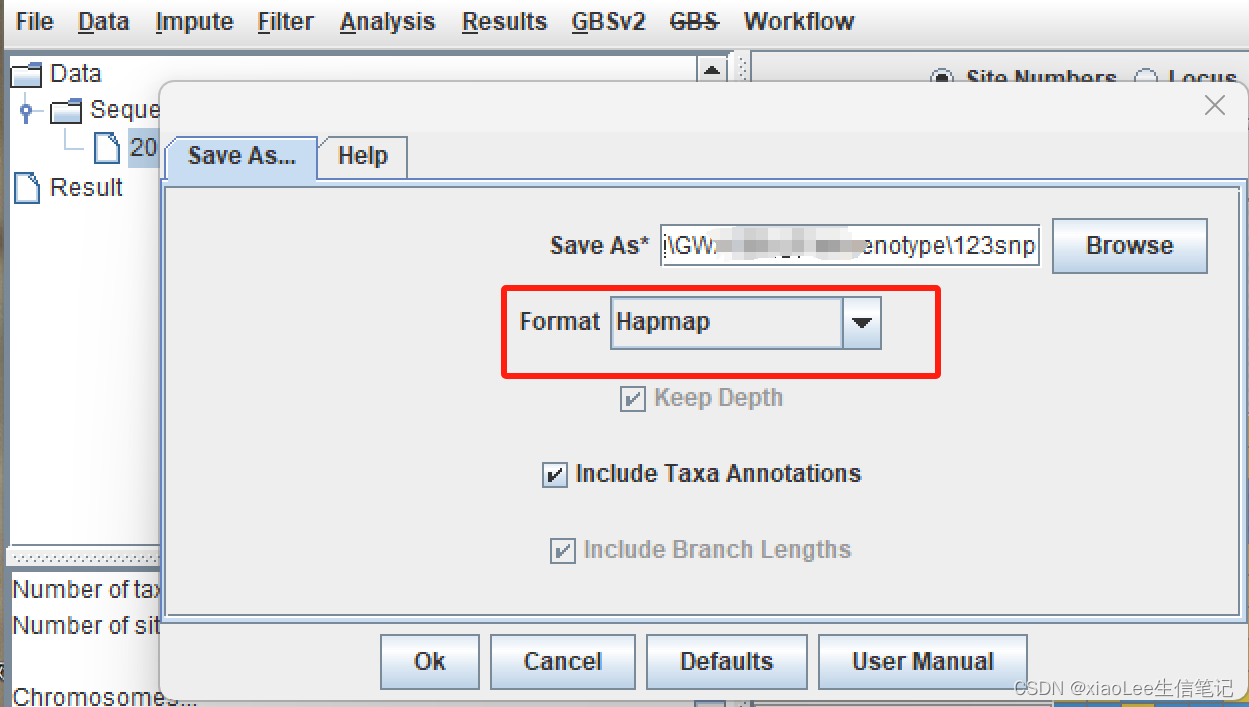
对代码文件转换不熟的均可以通过Tassel软件的另存为(File下的Save As) Format 中选择自己想要转换的格式;
GAPIT3软件安装:
install.packages("devtools")
devtools::install_github("jiabowang/GAPIT3",force=TRUE)多个模型运行:
library(GAPIT)
#Step 1: Set data directory and import files
myY <- read.table("phe.csv", head = TRUE,sep=",")
myG <- read.delim("geno.hmp.txt", head = FALSE)
# 多个模型run
myGAPIT <- GAPIT(Y=myY,G=myG,
PCA.total=6, model = c( "GLM" ,"MLM", "FarmCPU","Blink"),
Multiple_analysis = TRUE
)GAPIT可以内部自动计算kin亲缘关系,和指定个数的PCA,进行多个模型计算,或者单个模型的运行。
单个模型运行:
myGAPIT_GLM <- GAPIT(Y=myY,G=myG,model="GLM",PCA.total=5)具体步骤过程可下载说明书参考:
GitHub - jiabowang/GAPIT: Genome Association Predict Integrate Toolshttps://github.com/jiabowang/GAPIT




















 1016
1016

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








