首先基因型文件满足plink格式,生成包含有ped.map及二进制的bed.bim和fam即可;
./gemma-0.98.1-linux-static -bfile g -gk 2 -p p.txt -o gemma 亲缘关系矩阵;
./gemma-0.98.1-linux-static -bfile g -k ./output/gemma.sXX.txt -lmm 1 -p p.txt -c c.txt -o gemma_out 关联分析;
主要参考邓飞笔记 | GWAS 操作流程5-1:根红苗正的GWAS分析软件:GEMMA - 云+社区 - 腾讯云 (tencent.com)笔记 | GWAS 操作流程5-2:利用GEMMA软件进行LMM+PCA+协变量 - 云+社区 - 腾讯云 (tencent.com)两篇文章
中间出错有,采用协变量成分时如下: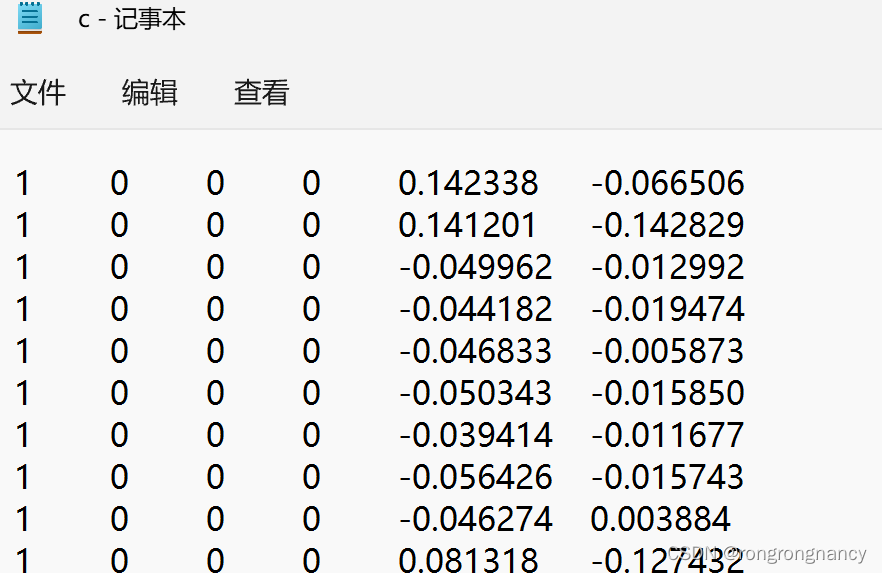
一直报错:GSL ERROR: matrix is singular in lu.c at line 266 errno 1,
出不来结果,中间无数次修改,还看到有博主说删除第一列截距就能出来结果,可我的还是不行。
再最后一遍尝试把中间的性别和世代都删除了忽然就出来结果了。
当然也不能确定结果就正确,也不太明白协变量应该留那些,目前就是留了
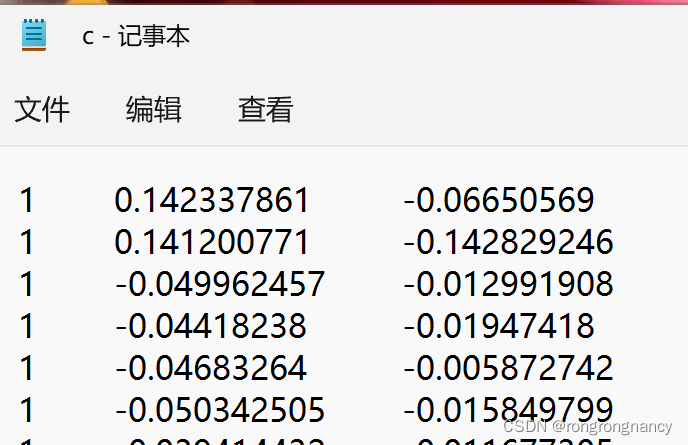
这样协变量就能做出来了。
如果有用也希望能帮助到你,有错误也希望能告诉我下。一起入门学习,借鉴经验。
参考文献:




 本文介绍了使用GEMMA软件进行基因组关联分析(GWAS)时遇到的矩阵奇异错误,并分享了解决方案。在处理协变量时,删除某些列(如性别和世代信息)后成功得到结果。作者提醒,选择协变量需谨慎,并提供了相关参考文献以供进一步学习。
本文介绍了使用GEMMA软件进行基因组关联分析(GWAS)时遇到的矩阵奇异错误,并分享了解决方案。在处理协变量时,删除某些列(如性别和世代信息)后成功得到结果。作者提醒,选择协变量需谨慎,并提供了相关参考文献以供进一步学习。
















 2594
2594










