大家都晓得 ROS,活性氧嘛......但但但但但是,它可并非是一种单分子,而是一类总称!那么,ROS 都包含哪些?当真集百害而无一利?与多种疾病相关的 ROS 又该如何检测??
Section.01
活性氧 (ROS): 一类总称
首先,大家所熟知的活性氧—— ROS,并不是单个分子的名称,而是含氧原子的高反应性化合物的总称!
如图 1 所示,ROS 可以是中性分子,如 H2O2;离子,如超氧阴离子 O2•- ;或自由基,如羟基自由基•OH;当然也可以是单线态氧 1O2 。其中,羟基自由基•OH 为自由基,其余则为非自由基[1]。不同 ROS 的形成升高导致分子损伤,称为“氧化应激”。O2•- 由 O2 还原而成,是其他 ROS 的前体。例如,O2•- 分解形成 H2O2,H2O2 与亚铁化合物和相关还原剂发生Fenton反应或Haber-Weiss反应进一步还原为・OH[2][3]。
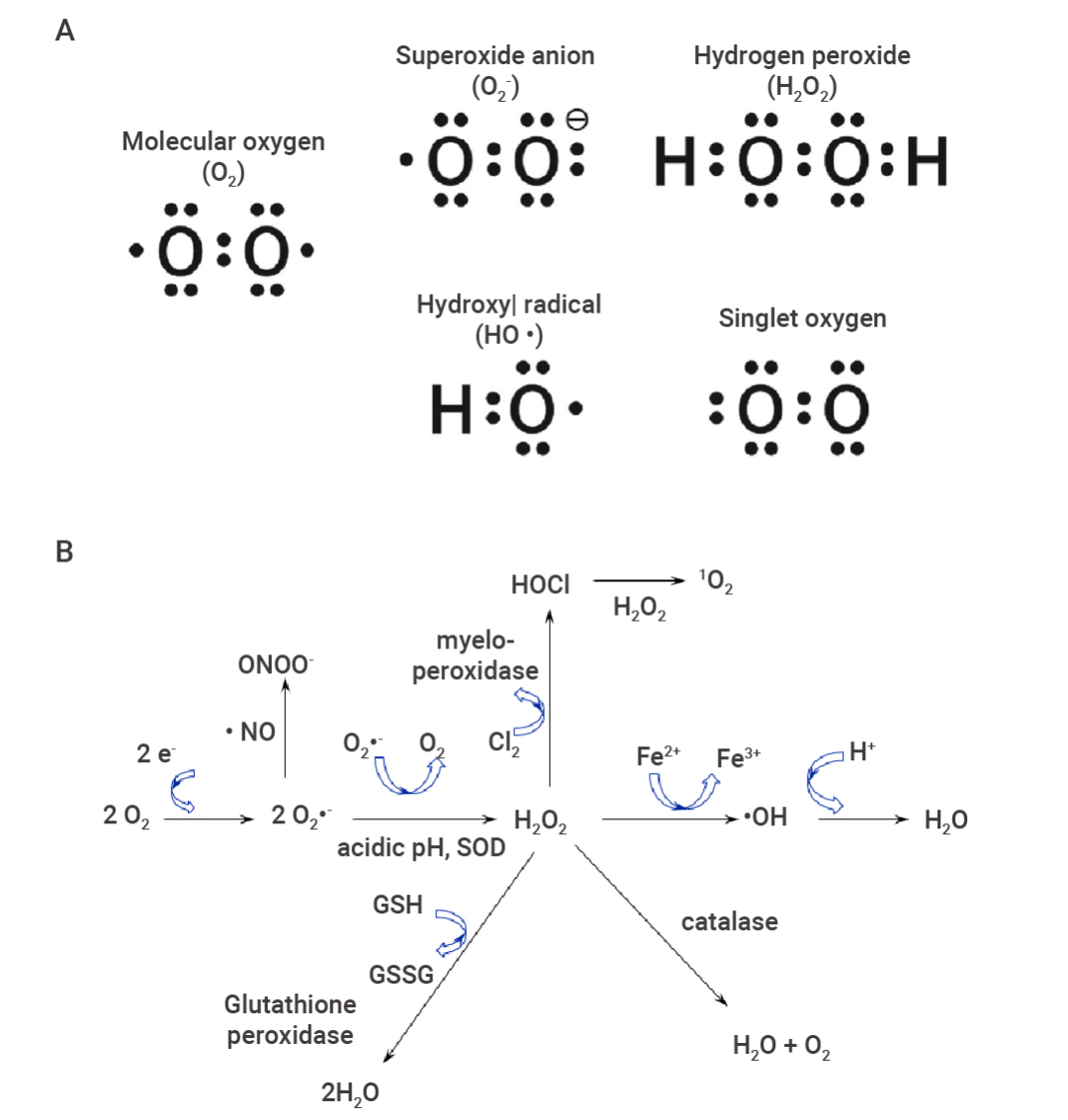
图 1. 活性氧 ROS 的电子式及转换[2][3]。
A.活性氧的电子式。超氧阴离子 (O2•-)、过氧化氢 (H2O2)、羟基自由基 (•OH) 和单线态氧 (1O2) 的电子式 (从左上到右下)。图中还显示了分子氧的电子式以供比较。B. 活性氧反应性强,可迅速转换。值得注意的是,超氧阴离子(O2•-)是不稳定的,在超氧化物歧化酶(SOD)的作用下会迅速变成过氧化氢 (H2O2)。如果阴离子与一氧化氮 (NO) 反应,则形成过氧亚硝酸盐 (ONOO-)。单线态氧(1O2)可由次氯酸(HOCI)与 H2O2 反应生成。
ROS:呼吸作用的副产物
ROS 由各种细胞区室产生,包括细胞质、细胞膜、内质网、线粒体和过氧化物酶体。在这些区室中,线粒体是 ROS 产生的主要贡献者,它们产生了大约 90% 的细胞 ROS (图 2)[4][5]。在正常的氧代谢过程中,ATP 合成会产生 ROS。因此,在大多数情况下,ROS 被认为是呼吸作用的副产物。
除此之外,内源性 ROS 产生的机制还包括内质网应激和未折叠蛋白反应、过氧化物酶体的代谢反应、NADPH 氧化酶 2 系统和酶促反应等。当然,暴露于某些有害因素,如某些外源性药物、感染因子、污染、紫外线、香烟烟雾和辐射,也可能导致 ROS 的产生。
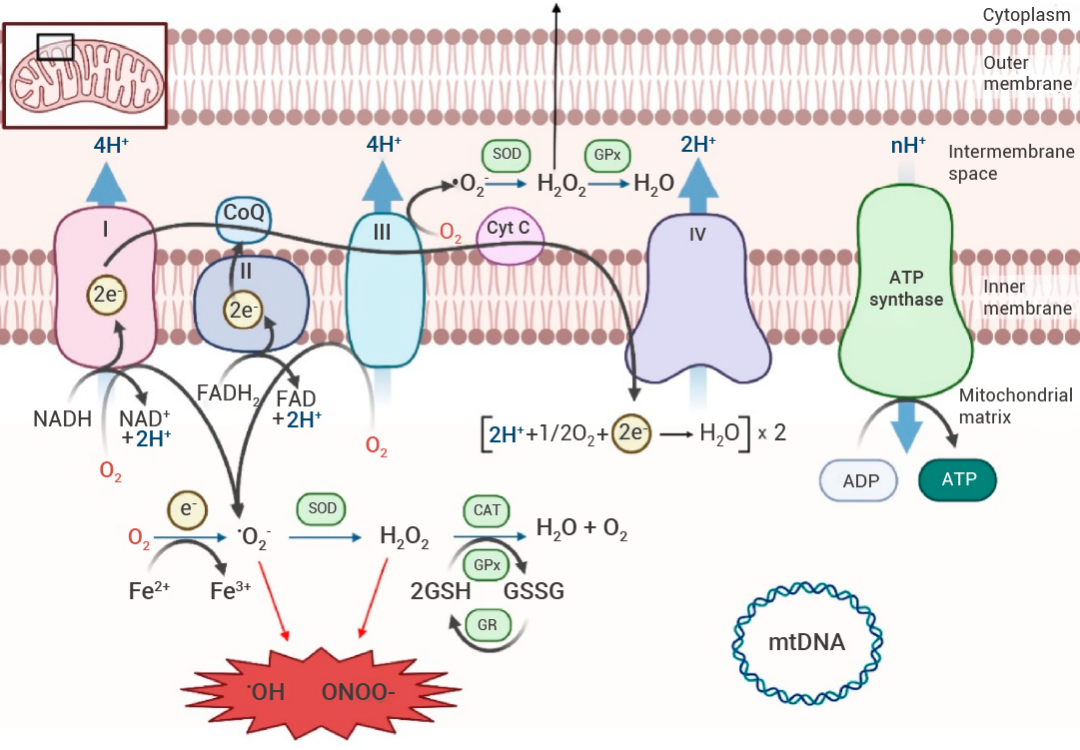
图 2. 线粒体活性氧 (ROS) 的主要来源[5]。
线粒体呼吸链 (MRC) 由四种酶复合物 (I-IV) 组成。Krebs 循环中 NADH 和 FADH 2 提供的电子分别在复合物 I 和 II 处转移,并依次转移至复合物 III 和 IV。该电子转移与跨内膜的质子转运偶联,产生质子梯度,从而允许 ADP 通过 ATP 合酶(复合物 V)进行磷酸化。氧代谢产生超氧化物阴离子 (•02− ),超氧化物歧化酶 (SOD) 将其转化为过氧化氢 (H2O2),然后在一些抗氧化酶防御机制(例如过氧化氢酶 (CAT) 或谷胱甘肽过氧化物酶 (GPx) 还原酶 (GPr) 系统)的作用下转化为水。过量的 ROS 产生会氧化蛋白质、脂质或线粒体 DNA (mtDNA),最终导致线粒体功能障碍和细胞死亡。
“双刃剑”:ROS 的两面性
你可能不了解 ROS,但你一定听过护肤品的宣传词,什么抗老抗氧化,清除自由基,新升级自由基智感防御…… blabla~~
的确!过量 ROS 对身体大有害处。然鹅!在正常的细胞环境中,ROS 对生命至关重要。
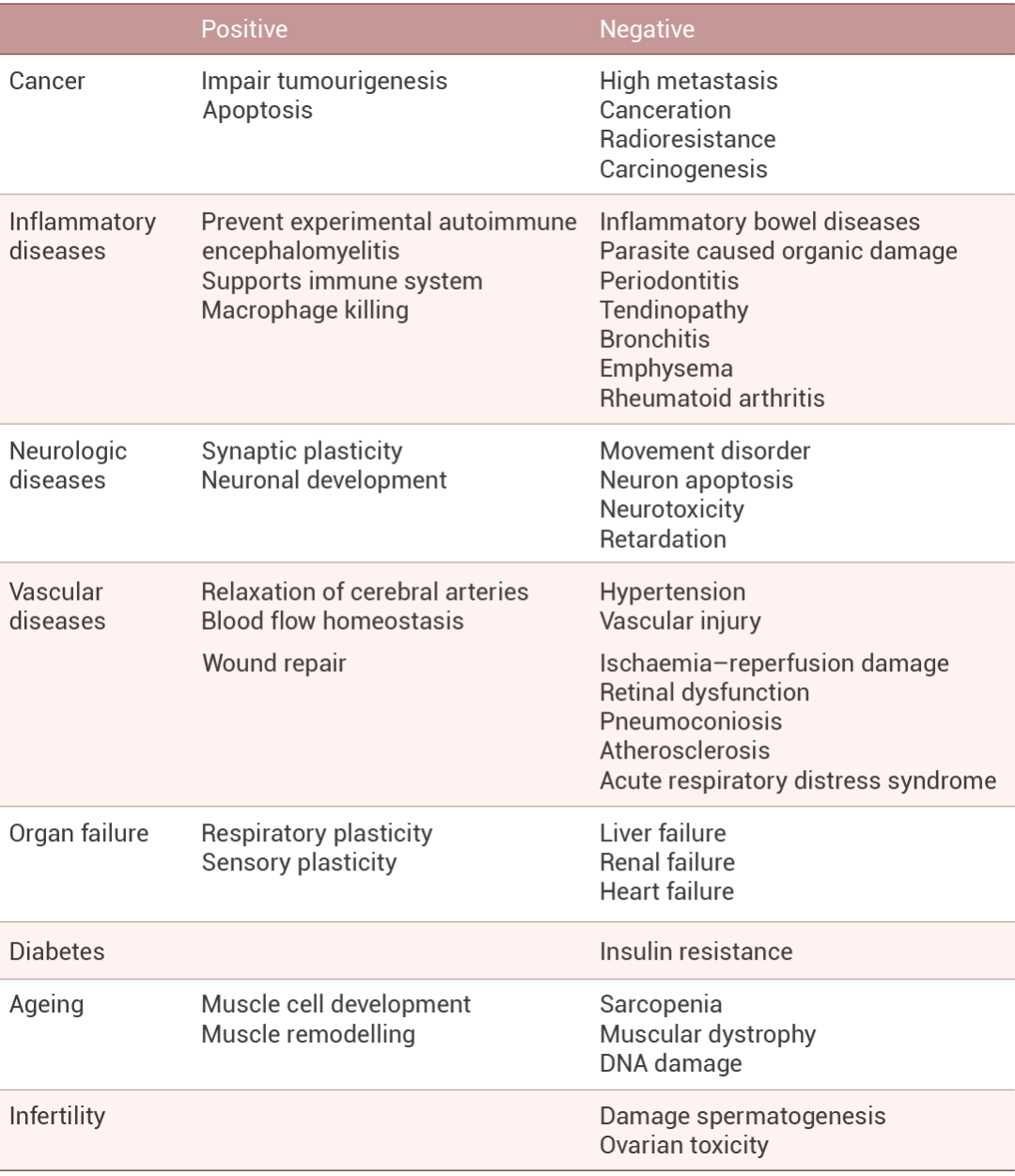
一方面,ROS 具有高反应性,很容易与几乎所有类型的生物分子发生反应。过量 ROS 通过氧化生物分子,如细胞膜脂质、组织蛋白质或酶、碳水化合物和 DNA 等引起氧化应激,导致细胞损伤。此外,ROS 可导致多种病理,癌症、心血管疾病和神经系统疾病都显示出 ROS 参与。另一方面,其参与不同的生物功能,低至中等浓度的 ROS 通过调节细胞信号级联发挥作用。例如通过氧化还原调节蛋白质磷酸化、离子通道和转录因子。某些生物合成过程也需要 ROS,包括甲状腺激素的产生和细胞外基质的交联等[1][3]。此外,神经传递、血小板聚集、血压调节、免疫系统控制、学习和记忆、细胞生长调节、重要生物化合物的合成和外源代谢都与 ROS 有关。
表 1. 活性氧在各种疾病中的作用[6]。
So!在 ROS 如此热门的研究态势下,更深入地了解其作用机制,便是重中之重。Buttttt,活性物质嘛,总是难以检测,譬如寿命很短,体内存在的抗氧化剂种类繁多等。不过!结合使用合适的荧光探针,可是测量 ROS 的一种极好的方法。
Section.02
活性氧 (ROS): 检测方案
本部分主要介绍 ROS 的荧光检测方法!
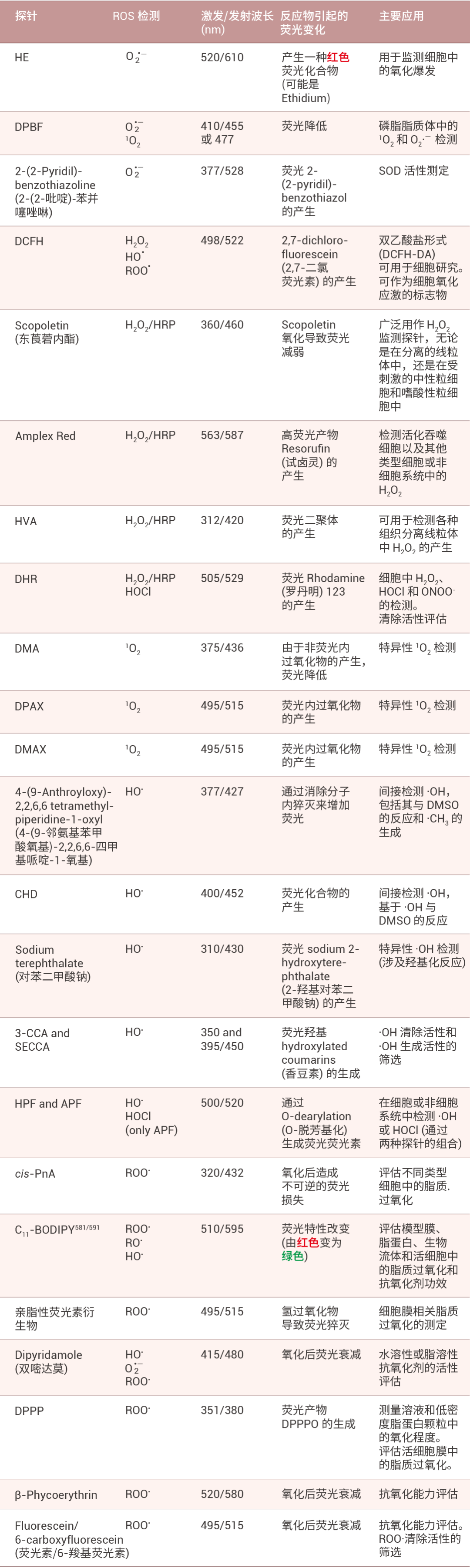
不同种类的 ROS 应该选取哪种荧光探针?激发/发射波长、反应物诱导的荧光变化和主要应用都有哪些?如表 2 所示,小 M 为大家整理了 23 种荧光探针的相关信息(可登录MCE官网、公众号后台私信或联系销售购买),大家点赞收藏,按需自取喔~
表 2. 不同荧光探针的 ROS 检测方案[1]。
超氧化物自由基(O2•-) 检测
提到超氧化物 (O2•-),就得先聊聊荧光探针Dihydroethidium(DHE,又称 Hydroethidine, HE, HY-D0079)!其主要检测 O2•-。由于 O2•- 通常是占比最高的 ROS 成分,DHE 也被广泛用于 ROS 的检测。未氧化形式下,DHE 产生固有的蓝色荧光 (Ex/Em=370/420 nm),当 DHE 被 O2 氧化时,产生了一种荧光化合物乙锭 (E+),可嵌入 DNA 的双螺旋结构中,形成乙锭-DNA 复合物,产生红色荧光[1][7]。
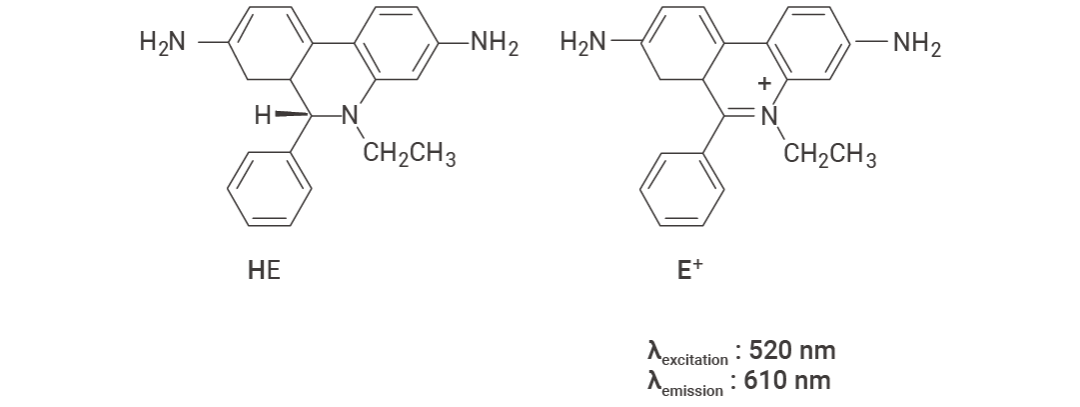
图 3. Hydroethidine (HE) 和 ethidium (E+) 的化学结构[1]。
-
DHE 实验指南
实验步骤[7]
1. 溶液配制
(1)制备储存液
用无水 DMSO 配制 10 mM 的 DHE 储备液 (1mg DHE 溶于0.317 mL DMSO 中)。
(2)工作液的配置
用预热好的无血清细胞培养基或 PBS 稀释储存液,配制成 1-10 μM 的 DHE 工作液(请根据实际情况调整 DHE 工作液的浓度)。
2. 细胞染色 (悬浮细胞)
(1)1000 g 离心 3-5 分钟,弃上清。PBS 洗涤 2 次,每次 5 分钟。
(2)加入 1 mL Dihydroethidium 工作液,室温孵育 30 分钟。
(3)4℃,400 g 离心 3-4 分钟,弃上清。
(4)PBS 洗涤 2 次,每次 5 分钟。
(5)用无血清细胞培养基或 PBS 重悬细胞,荧光显微镜或流式细胞仪检测。
3. 细胞染色 (贴壁细胞)
(1)将贴壁细胞培养于无菌盖玻片上。
(2)从培养基中移出盖玻片,吸除多余培养基。
(3)加入 100 μL 染料工作液,轻轻晃动使其完全覆盖细胞,孵育 30 分钟。
(4)吸去染料工作液,用培养基洗 2-3 次,每次 5 分钟 ,使用荧光显微镜或流式细胞仪进行观察(若需要用流式细胞仪检测,需将细胞用胰蛋白酶消化重悬后再进行染色)。
上下滑动查看更多
实验结果
图 4A 为 DHE 细胞染色结果,检测样本为 U87 细胞,使用 DHE 5 μM 避光染色 30 min[8]。 图 4B-C 为 DHE 组织染色结果,图 4B 检测样本为小鼠大脑冰冻切片,使用 DHE 10 μM 避光染色 1 h[9]。图 4C 检测样本为小鼠视网膜石蜡切片,使用 DHE 5 μM 避光染色 20 min,设置了超氧化物歧化酶处理组以验证是否存在非特异干扰[10]。
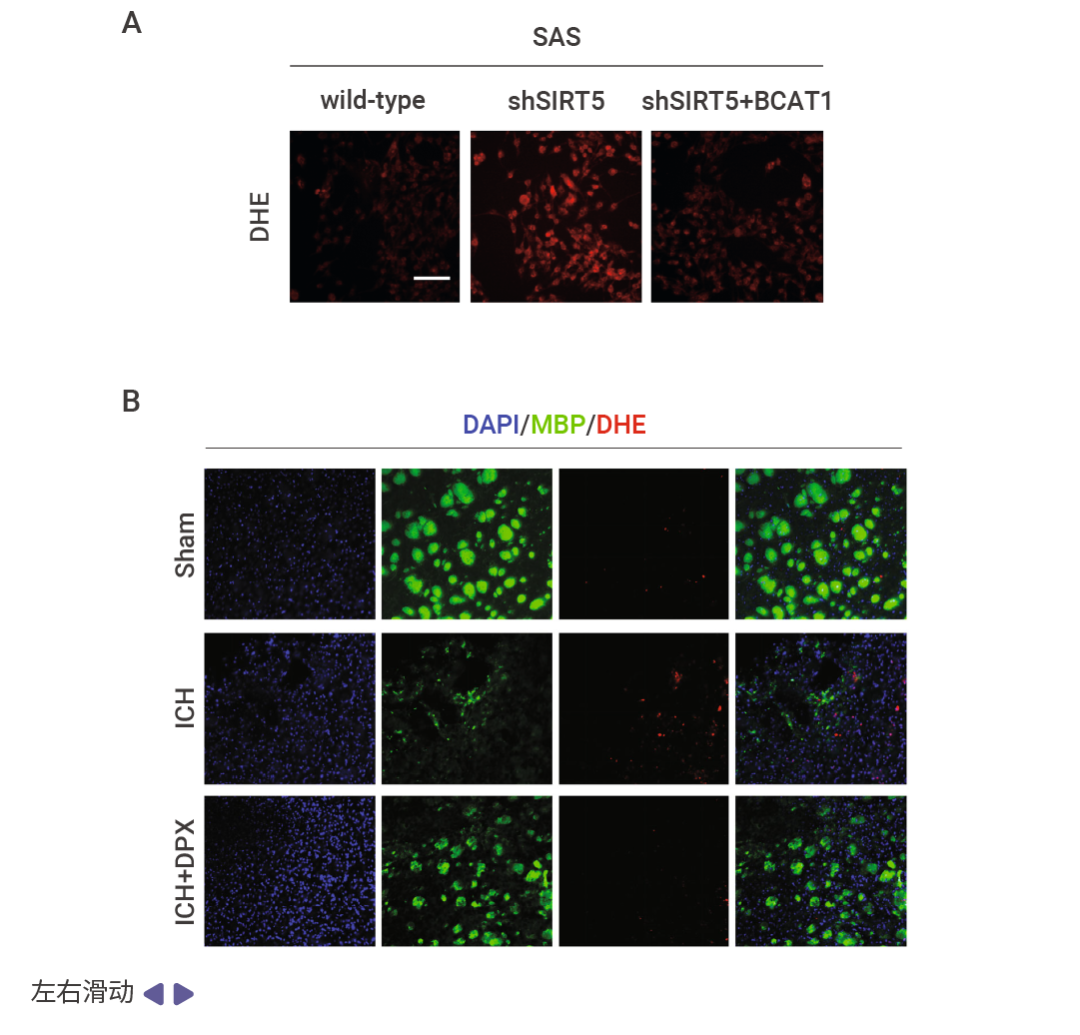
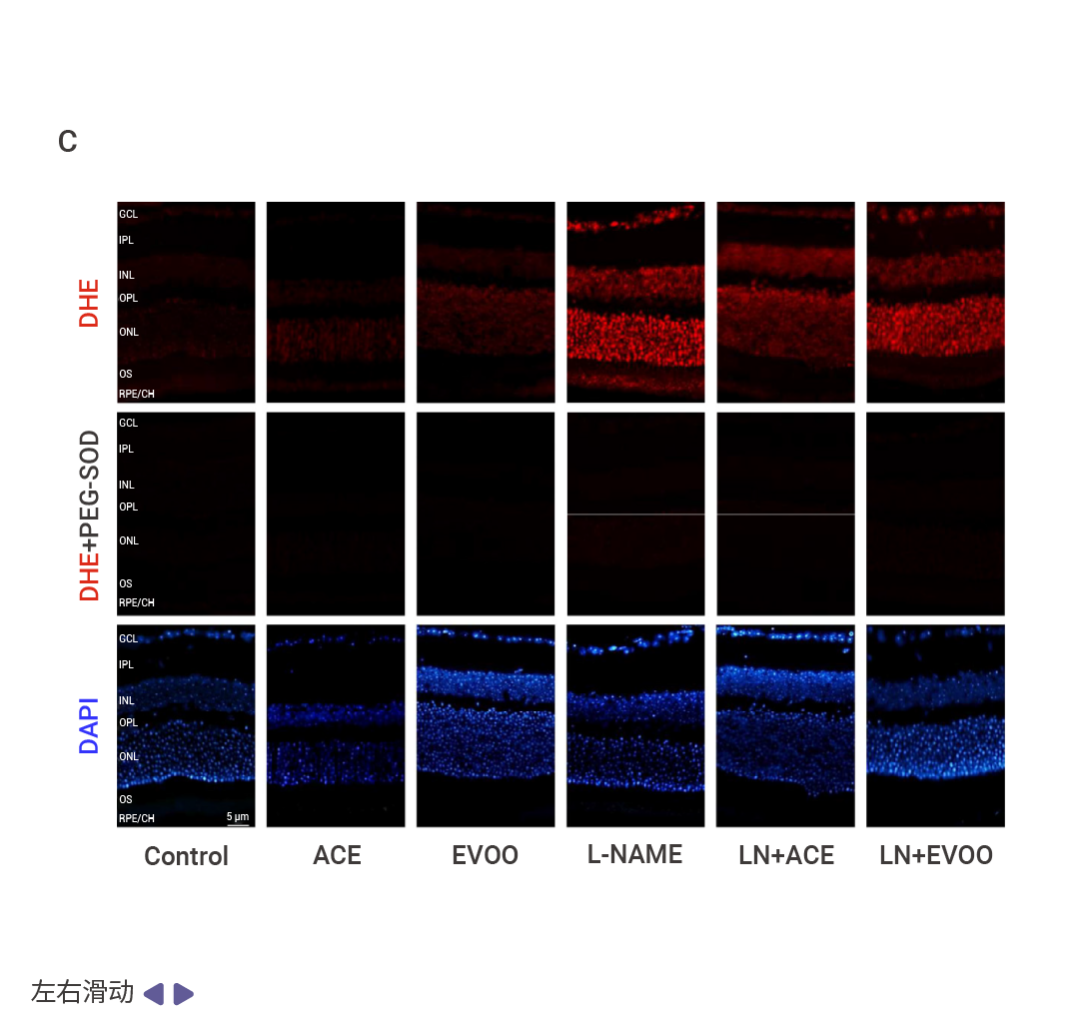
图 4. DHE 细胞和组织染色结果[8][9][10]。
荧光探针检测 H2O2
如图 5 所示,DCFH 氧化生成 DCF,一种荧光化合物,最初被认为是检测H2O2 的特定指示剂。然而,已经证明 DCFH 可以被其他 ROS 氧化。DCFH-DA(DCFH二乙酸形式, H2DCFH-DA, HY-D0940) 可以应用于细胞研究,因为它能够通过细胞膜扩散,然后被细胞内酯酶酶解成 DCFH。随后,被多种 ROS 氧化 的 DCFH 生成绿色荧光的 DCF,被广泛应用于总 ROS 的检测[1][11][12]。
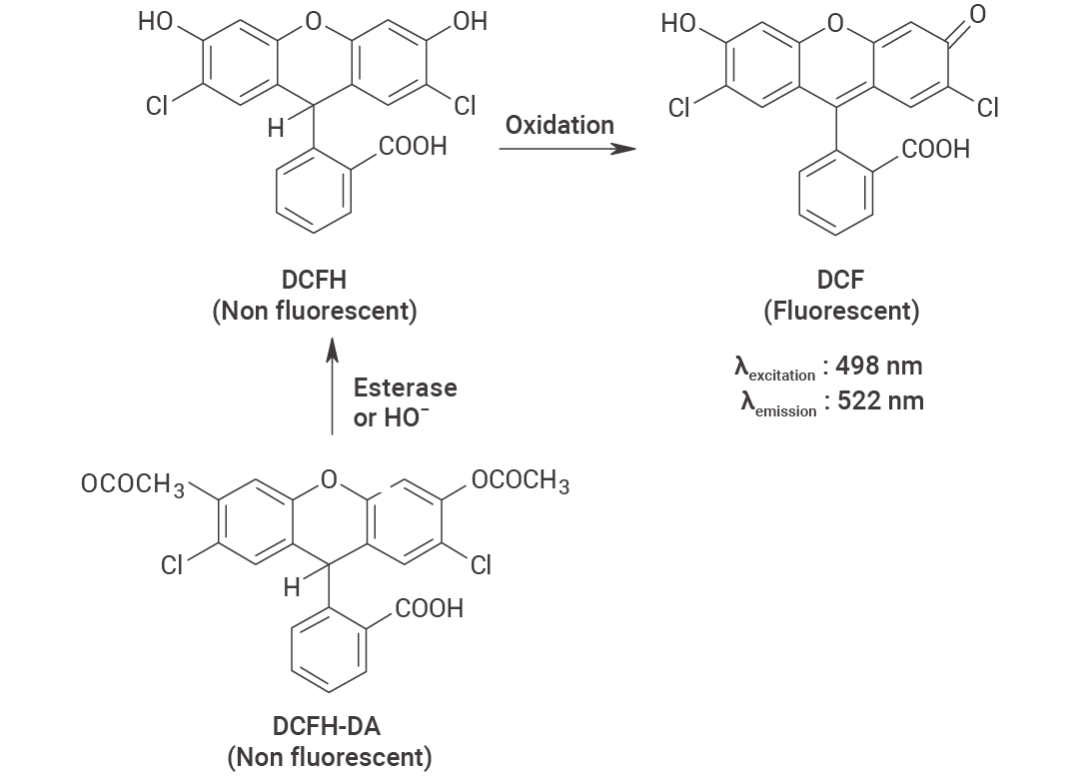
图 5. DCFH DA 脱酯化生成 DCFH,以及 ROS 和 RNS 进一步氧化生成荧光 DCF 的示意图[1]。
-
H2DCFDA 实验指南
实验步骤[7]
1. 溶液配制
(1) 制备储存液
用无水 DMSO 配制 10 mM 的 H2DCFDA 储备液 (5 mg 粉末加入 1.026 mL DMSO)。
(2)工作液的配制
用预热好的无血清细胞培养基或 PBS 稀释储存液,配制成 1-10 μM 的 H2DCFDA 工作液(请根据实际情况调整 H2DCFDA 工作液浓度,且现用现配)。
2. 细胞染色 (悬浮细胞)
(1)1000 g 离心收集细胞,加入 PBS 洗涤两次,每次 5 分钟。
(2)加入 1 mL 染料工作液,室温孵育 5-30 分钟。
(3)400 g 离心 3-4 分钟,弃去上清。
(4)加入 PBS 洗涤细胞两次,每次 5 分钟。
(5)用 1 mL 无血清培养基或 PBS 重悬细胞后,使用荧光显微镜或流式细胞仪进行观察。
3. 细胞染色 (贴壁细胞)
(1)将贴壁细胞培养于无菌盖玻片上。
(2)从培养基中移出盖玻片,吸除多余培养基。
(3)加入 100 μL 染料工作液,轻轻晃动使其完全覆盖细胞,孵育 5-30 分钟。
(4)吸去染料工作液,用培养基洗 2-3 次,每次 5 分钟 ,使用荧光显微镜或流式细胞仪进行观察(若需要用流式细胞仪检测,需将细胞用胰蛋白酶消化重悬后再进行染色)。
实验结果
图 6A 为 DHE 细胞染色结果,检测样本为 4T1 细胞,H2DCFDA 5 μM 避光染色 30 min[13]。 图 6B 为 DHE 组织染色结果,检测样本为活体海马切片,使用 H2DCFDA 10 μM 避光染色 30 min[14]。
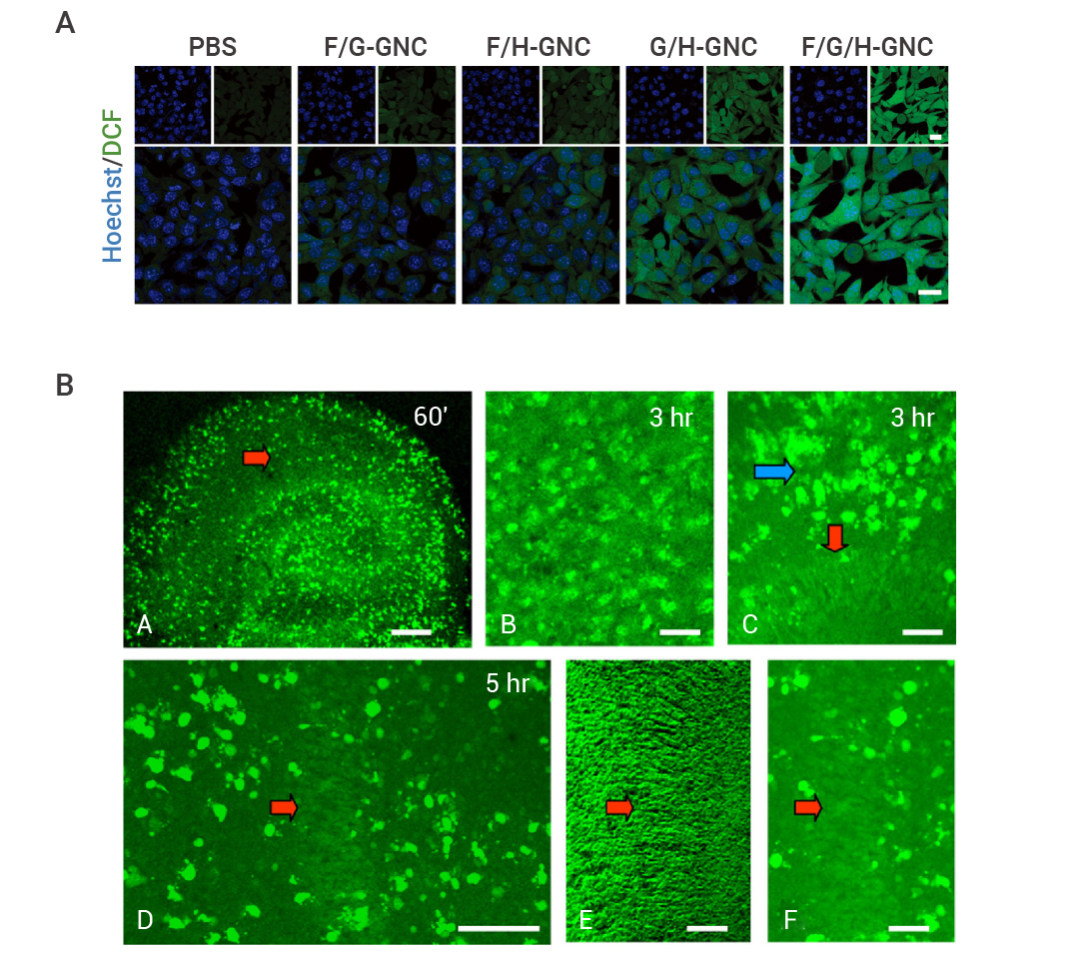
图 6. H2DCFDA 细胞和组织染色结果[13][14]。
线粒体超氧化物(O2•-) 指示剂
线粒体有氧代谢是 ROS 的主要来源之一,不过也有少量 ROS 是NAD(P)H 氧化酶、细胞色素 P450 单加氧酶等产生的。在研究线粒体相关疾病机制,或评估线粒体靶向药物效果时常需要聚焦于单独的线粒体 ROS,这该如何是好?
莫慌,MitoSOX Red(HY-D1055)就能够避免其他来源 ROS 的干扰,直接反映线粒体的超氧化物生成状态。MitoSOX Red 可以穿透细胞膜,通过线粒体膜电位依赖性的方式积聚在线粒体中,并被其中的超氧化物氧化,氧化产物与线粒体核酸结合发出强烈荧光 (Ex/Em=510/580 nm)[15][16]。
-
MitoSOX Red 实验指南
实验步骤[15][16]
1. 溶液配制
(1)制备储存液
用无水 DMSO 配制 5 mM 的 MitoSOX Red 储备液 (50 μg 粉末加入 13 μL DMSO)。
(2)工作液的配制
用预热好的无血清细胞培养基或 PBS 稀释储存液,配制成 1-10 μM 的 MitoSOX Red 工作液(请根据实际情况调整 MitoSOX Red 工作液浓度,且现用现配)。
2. 细胞染色 (悬浮细胞)
(1)4℃ 1000 g 离心收集细胞,加入 PBS 洗涤两次,每次 5 分钟。细胞密度为 1×106/mL。
(2)加入 1 mL 染料工作液,室温孵育 5-30 分钟。
(3)4℃ 400 g 离心 3-4 分钟,弃去上清。
(4)加入 PBS 洗涤细胞两次,每次 5 分钟。
(5)用 1 mL 无血清培养基或 PBS 重悬细胞后,使用荧光显微镜或流式细胞仪进行观察。
3. 细胞染色 (贴壁细胞)
(1)将贴壁细胞培养于无菌盖玻片上。
(2)从培养基中移出盖玻片,吸除多余培养基。
(3)加入 100 μL 染料工作液,轻轻晃动使其完全覆盖细胞,孵育 5-30 分钟。
(4)吸去染料工作液,用培养基洗 2-3 次,每次 5 分钟 ,使用荧光显微镜或流式细胞仪进行观察(若需要用流式细胞仪检测,需将细胞用胰蛋白酶消化重悬后再进行染色)。
实验结果
图 7 为 MitoSOX Red 细胞染色结果,检测样本为 Huh7 细胞,使用 5 μM MitoSOX Red 避光染色 30 min[17]。
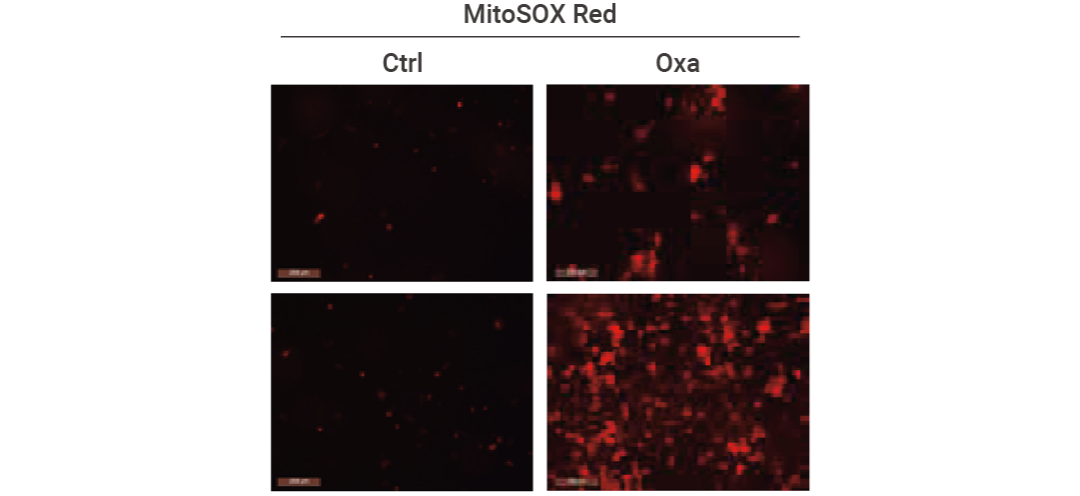
图 7. MitoSOX Red 细胞染色结果示例[17]。
注意事项:
-
避光操作:DHE 储存液、H2DCFDA 储存液、MitoSOX Red 储存液建议分装后在 -20℃ 或 -80℃ 避光保存,避免反复冻融。同时,实验全程也需要避光操作,溶解、孵育、洗涤和成像需要在暗环境中进行,必要时可用铝箔等包裹样品。
-
含核酸样本:DHE 的荧光依赖于与核酸的结合,所以不能用于不含核酸的溶液样本的 ROS 检测。
-
及时观察:H2DCFDA 和 MitoSOX Red 在染色后需要尽快观察,并且染色后也不可固定细胞。
-
活细胞染色:H2DCFDA 和 MitoSOX Red 只适用于活细胞染色,DHE 推荐用于未固定的新鲜切片或活细胞(虽然 DHE 可以尝试用于固定切片的染色,但是活细胞染色 DHE 后不可固定细胞)。
也有部分文献将 DHE 用于石蜡切片,因为固定操作对 ROS 有一定的影响,固定时间、染色浓度等条件都需要根据实际样本情况做调整,总体的染色效果往往也不如未固定的样本,也有可能难以做出理想的效果,实在有固定切片染色需要的小伙伴可以参考上面图 4 的组织染色示例摸索一下,并且建议设置超氧化物歧化酶处理组等实验组来排除可能存在的非特异染色干扰~
Section.03
小结
好啦,本期干货较多,需要的小伙伴们记得点赞收藏~
此外,许多小伙伴会问到关于组织切片的染色问题,需要注意固定的操作可能破坏细胞内 ROS 的分布,小 M 优先建议使用无固定的冰冻切片或活体切片,在切片后尽快进行染色观察,组织染色的实验难度相对较大,需要摸索的条件 (切片条件、染色浓度时间等) 相对较多,可以参考细胞染色的浓度和时间摸索一下切片染色的效果,并且做好无处理对照减少自发荧光等因素的干扰~

[1] Gomes A, et al. Fluorescence probes used for detection of reactive oxygen species. J Biochem Biophys Methods. 2005 Dec 31;65(2-3):45-80.
[2] Kakizawa S. Involvement of ROS signal in aging and regulation of brain functions. J Physiol Sci. 2025 Mar;75(1):100003.
[3] Brieger K,et al. Reactive oxygen species: from health to disease. Swiss Med Wkly. 2012 Aug 17;142:w13659.
[4] Tirichen H,et al. Mitochondrial Reactive Oxygen Species and Their Contribution in Chronic Kidney Disease Progression Through Oxidative Stress. Front Physiol. 2021 Apr 23;12:627837.
[5] García-García, Francesc Josep et al. Nutrition, Bioenergetics, and Metabolic Syndrome. Nutrients vol. 12,9 2785. 11 Sep. 2020.
[6] Yang S, Lian G. ROS and diseases: role in metabolism and energy supply. Mol Cell Biochem. 2020 Apr;467(1-2):1-12. doi: 10.1007/s11010-019-03667-9. Epub 2019 Dec 7. Erratum in: Mol Cell Biochem. 2020 Apr;467(1-2):13.
[7] Zielonka J, et al. Global profiling of reactive oxygen and nitrogen species in biological systems: high-throughput real-time analyses. J Biol Chem. 2012 Jan 27;287(5):2984-95.
[8] Wang T, et al. SIRT5-mediated BCAT1 desuccinylation and stabilization leads to ferroptosis insensitivity and promotes cell proliferation in glioma. Cell Death Dis. 2025 Apr 7;16(1):261.
[9] Wang B, et al. Dexpramipexole Attenuates White Matter Injury to Facilitate Locomotion and Motor Coordination Recovery via Reducing Ferroptosis after Intracerebral Hemorrhage. Oxid Med Cell Longev. 2022 Aug 4;2022:6160701.
[10] Santana-Garrido Á, et al. Retinoprotective Effect of Wild Olive (Acebuche) Oil-Enriched Diet against Ocular Oxidative Stress Induced by Arterial Hypertension. Antioxidants (Basel). 2020 Sep 18;9(9):885.
[11] Gomes A, et al. Fluorescence probes used for detection of reactive oxygen species. J Biochem Biophys Methods. 2005 Dec 31;65(2-3):45-80.
[12] Lyublinskaya OG, et al. Redox environment in stem and differentiated cells: A quantitative approach. Redox Biol. 2017 Aug;12:758-769.
[13] Han W, et al. Self-Sustained Biophotocatalytic Nano-Organelle Reactors with Programmable DNA Switches for Combating Tumor Metastasis. Adv Mater. 2025 Mar;37(9):e2415030.
[14] Feeney CJ, et al. Vulnerability of glial cells to hydrogen peroxide in cultured hippocampal slices. Brain Res. 2008 Mar 10;1198:1-15.
[15] Price OT, et al. Quantitative fluorescence of 5-FU-treated fetal rat limbs using confocal laser scanning microscopy and Lysotracker Red. Cytometry A. 2003;53(1):9-21.
[16] Fengjiao Jin, et al. The PI3K/Akt/GSK-3β/ROS/eIF2B pathway promotes breast cancer growth and metastasis via suppression of NK cell cytotoxicity and tumor cell susceptibility. Cancer Biol Med. 2019 Feb;16(1):38-54.
[17] Yu T, et al. SIRT7-mediated NRF2 deacetylation promotes antioxidant response and protects against chemodrug-induced liver injury. Cell Death Dis. 2025 Apr 1;16(1):232.






















 2224
2224

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








