背景介绍
CNV检测已成为全基因组分析的常规内容,并显著提升了阳性诊断率。然而,由于实验室水平和所使用软件的差异,目前仍难以全面、准确地覆盖CNV的检测与细节分析。尤其在数据分析环节,目前尚无开源软件能够在性能优越的同时,全面解决这一问题。以流行的CNVnator为例,这是一款基于Read Depth(RD)原理的拷贝数变异检测软件,主要用于全基因组数据分析。CNVnator不仅能在人群中进行拷贝数变异检测和基因分型,还能根据需求鉴定一些非典型CNV。
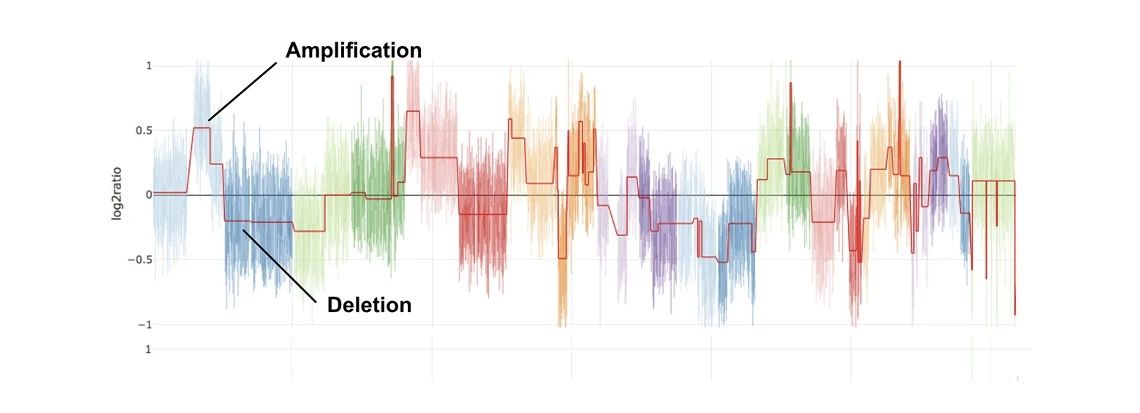
总体而言,CNVnator具备较高的灵敏度、较低的错误发现率、并且其断点检测分辨率较高。然而,作为一款经典软件,CNVnator在应对现今多平台测序数据和新一代参考基因组等最新数据类型时,已表现出一定的局限性。
1.适用场景
CNVscope是Sentieon推出的一款基于机器学习的全基因组CNV分析检测模块。该模块主要用于检测大于5kb的拷贝数增加或缺失,方法是通过分析reads的深度信息,并结合断点检测等其他特征进行拷贝数判断。
2.环境必备
- 软件授权:License须开通CNV模块权限
- 软件下载:https://insvast-download.oss-cn-shanghai.aliyuncs.com/Sen





 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 2253
2253

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








