视频来自:8_StatisticalAnalysis_哔哩哔哩_bilibili
pdf:The R-fMRI Course | The R-fMRI Network
目录
2.2. 多重比较矫正(Multiple Comparison Correction)
2.3. Different Multiple Comparison Correction Strategies
1. 质量控制(Quality Control)
1.1. 软件操作
(1)dpabi→Quality Control(⭐这些应该是进行了数据处理之后才有的文件,不是原始数据集)
(2)QC:Raw T1

①打开的要是如下.tsv文件,可以修改tsv文件中的数值

(3)QC:Raw Fun:会打开功能影像
(4)QC:Normalization
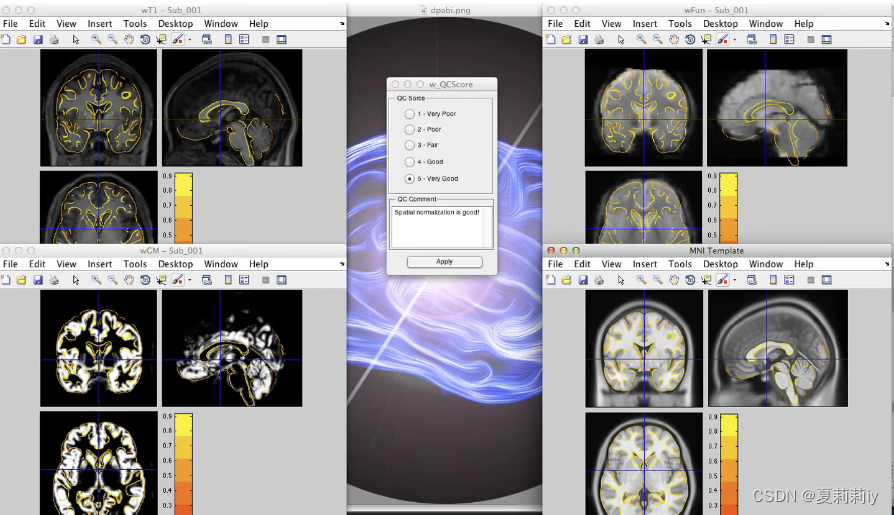
①黄色的线条是白质灰质的边界
②在中间QC Comment可以给对于白质灰质分界判断好坏的分数
③第二次点QC:Normalization的时候会生成分数框
(5)Threshold QC Score

①这个设定得到是分数的下界,所得数据要大于这分数才能进入被统计分析数据
②设定完后左边框中小于这些分数的被试都会被排除掉
(6)Generate Group Masks

①wAutoMask是MI空间,且根据它可以生成GroupMask

②可以在这个文件中输入值,此例为要求90%的被试都cover这个voxel才进入GroupMask(即进入统计分析)
(7)Threshold Coverage
①先选择GroupMask
②再次对比阈值,如果达到了就保留,否则舍去
(8)Motion Report
①显示MeanFD
②再点击view显示被试头动大小

(9)Threshold Motion
①被试的平均值和被排除的阈值设置

(10)最后会生成一个SubjectList把所有不合格的被试都排掉
2. 统计分析(Statistical Analysis)
2.1. 软件操作
(1)dpabi→Statistical Analysis
(2)可以做的统计

(3)One-Sample T-Test
①可以看看患者和普通人ALFF的图像差异

②在左边栏加入被试,右边是协变量,可以不添加

③生成的结果示例

(4)Two-Sample T-Test
①One Sample是没有实际意义的,一般要使用Two-Sample来进行数据分析
②在左方添加两个文件夹(值第一组减去第二组的值,所以顺序很有关系)

③上图中Covariate Images添加的灰质密度图像,Covariate Images为图像协变量
④功能上一般是3*3*3的voxel size,但结构上一般是1*1*1或1.5*1.5*1.5。此时用Utilities→Image Reslicer可以将结构像变成功能像的大小,否则在这不一样大会报错
⑤上图中Text Covariates添加了头动,Text Covariates是文本协变量
(5)Paired T-Test

①对同一个被试的重复测量,此图是同一被试睁眼闭眼的图像
②被试内设计具有很高的说服力,因为排除了组间差异。因此在小样本分析时,尽量用被试内分析
③个人一般不需要加协变量,除非加头动
(6)ANOVA or ANCOVA

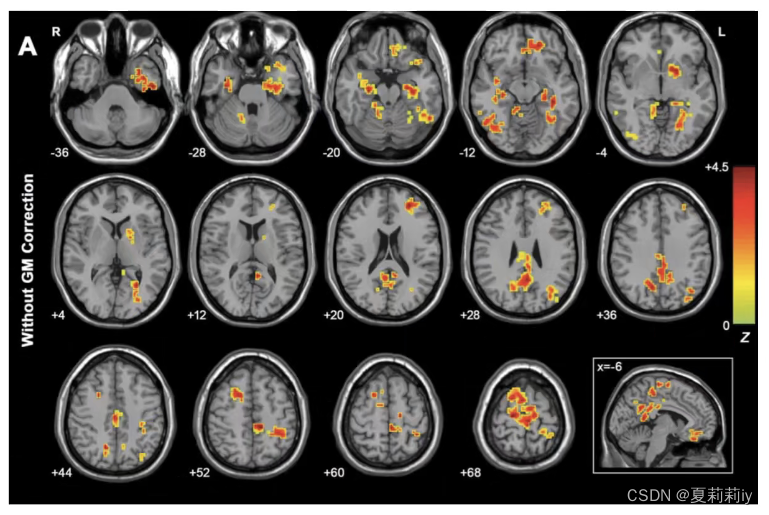
①有三组的时候就需要ANOVA,即推断三组之间有两组具有显著差异,避免多重比较
②可以直接做ANOVA,也可以在做的时候先做多重比较矫正。看结果可以用Z值


(7)Correlation Analysis
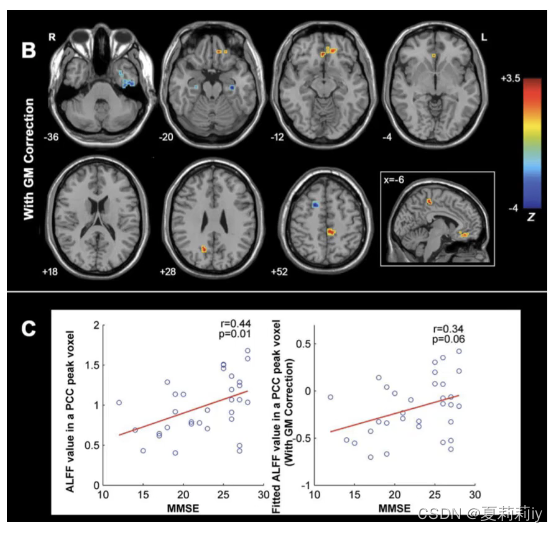
①用于有更多组的时候
②Correlation Seed Series:和什么做相关
(8)Mixed Effect Analysis

①做混合设计,既有被试内因素,也有被试间因素
②得到的文件,最重要的是中间那个

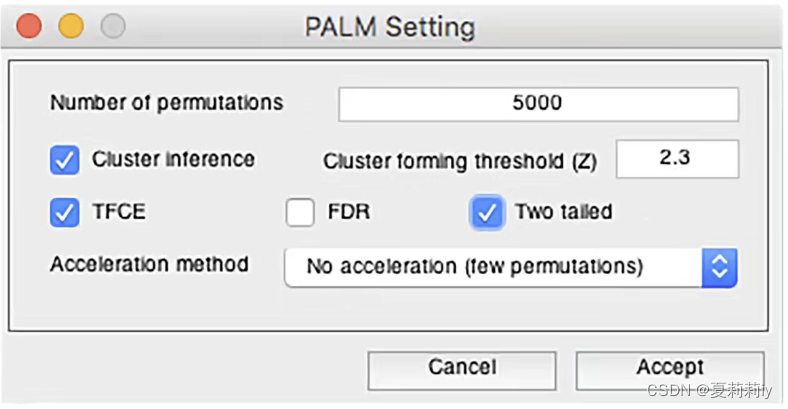
(9)最后在Run左边有Permutation test(PALM)可以勾画上,会跳出来选参框,一般选默认参数就好(但是5000可能太高了)
2.2. 多重比较矫正(Multiple Comparison Correction)
(1)概念:尽管一个被试的正确率可能高达0.95,但要保证在极大量下全部被试的全部正确率却会因为0.95^n(n极大)而显著降低
(2)优化
①Bonferroni:将p值(一般是0.05或0.01)缩小为p/n(n为检验次数)。但这样p值太小了,即算出来的significant值要<p/n才算显著。这样很难有显著的差异的样本
②False Discovery Rate(FDR):有很多种方法(太多且复杂就不一一例举,仅给出严老师PPT里给的方法),比Bonferroni更为温和实用。在DPABI_VIEW里Cluster可选FDR矫正


③Family-Wise Error(FWE):也有很多种办法,如下

④Gaussian Random Field Theory Correction(GRF Correction)

⑤AlphaSim Correction:不是很推荐
⑥Threshold-Free Cluster Enhancement(TFCE):

2.3. Different Multiple Comparison Correction Strategies

2.4. Test-retest Reliability

3. 结果查看(Results Viewing)
3.1. Viewer
(1)Overlay

(2)Overlay Configure:是阈值设置,可以滑动也可以输入
(3)Position上面XYZ是坐标,下面IJK是对应代码矩阵的位置
(4)Time Course:显示时间序列

(5)Atlas:看点击的区域是在大脑的什么位置

(6)Structure






 该文详细介绍了fMRI数据的质量控制流程,包括dpabi软件的操作,如QC步骤、阈值设定等。接着,文章阐述了统计分析的各种方法,如One-SampleT-Test、Two-SampleT-Test、ANOVA等,并提到了多重比较矫正的重要性和不同策略。最后,讨论了结果查看和可靠性测试。
该文详细介绍了fMRI数据的质量控制流程,包括dpabi软件的操作,如QC步骤、阈值设定等。接着,文章阐述了统计分析的各种方法,如One-SampleT-Test、Two-SampleT-Test、ANOVA等,并提到了多重比较矫正的重要性和不同策略。最后,讨论了结果查看和可靠性测试。
















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








