目录
模型简介
neurolib 是一个用python编写的全脑建模计算框架,也是可扩展的、允许轻松实现自定义神经质量模型,能够在介观尺度上表示脑区平均活动。通过加载结构和功能数据集,建立全脑模型,管理其参数,进行模拟,并组织其输出以供后续分析。neurolib为计算神经科学家提供了一个多功能平台,用于原型设计模型、管理大型数值实验、研究脑网络的结构-功能关系,以及对全脑模型进行体外优化。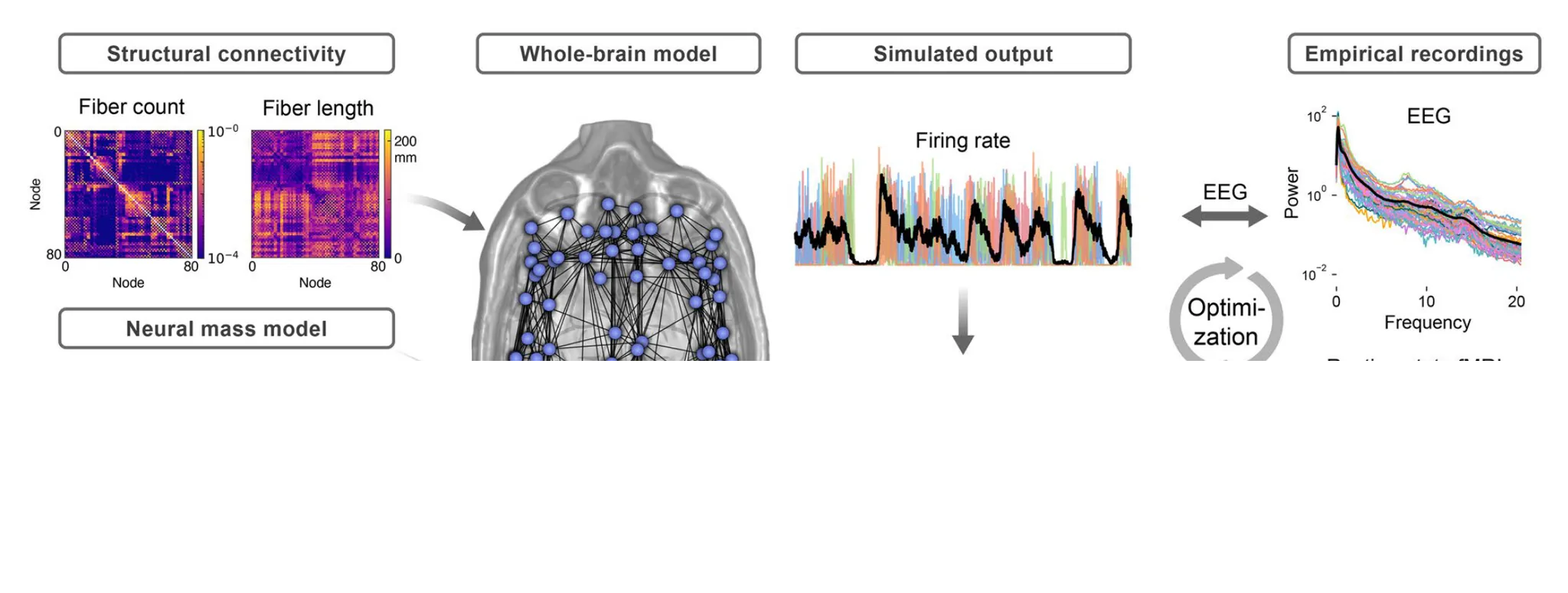
全脑模型:通过DTI纤维追踪技术获得的结构连接信息与神经质量模型相结合得到,之后模拟输出每个区域例如兴奋性放电率等,通过血流动力学气球-风箱模型转换为BOLD信号。并将模型的输出与经验数据进行比较,进行优化。
研发意义
如今神经系统模拟已经有很多软件框架,如 NEST、Brian、NetPyNE、NEURON等,但这些框架专注于微观神经元模型,很少用于宏观全脑建模,而 TVB 是一个较为成熟的脑网络模拟运行平台,虽然有 GUI 界面,但是拓展受限,neurolib 没有图形用户界面,但鼓励具有编程经验的用户修改框架本身的代码以适应其特定用例,实现自己的模型,并使用自己的数据集运行大规模数值实验。
模型部署
1. 全过程加速
neurolib使用了即时编译器numba,尽管TVB也使用了加速的numbacode,但仅加速了模型导数的计算,而非积分本身。在neurolib中,整个数值积分过程(包括脑网络中所有节点的循环计算)均得到加速,与TVB相比,单线程模拟的速度提升了8-24倍。
2. 并行化运行
为了在多核架构上加速参数探索和优化,neurolib利用了pypet 的并行化能力,可在单个CPU上同时运行多个模拟。
3. 可在分布式系统上部署
为了在分布式系统(如大型计算集群)上部署模拟,pypet可通过Python模块SCOOP 在多台机器上运行任务。一个轻量级的替代方案是mopet Python包,它可以利用分布式计算框架Ray 在多台机器上同时运行参数探索。
框架架构
1. 数据集类:
处理结构和功能数据集。
2. 模型核心:
Model基类,该基类负责初始化并运行模型、管理参数以及处理模拟输出。
3. 模型输出:
可通过血氧水平依赖(BOLD)信号模型转换为BOLD信号,从而实现模拟输出与经验性fMRI数据的对比。
4. 后处理函数和信号类:
用于计算功能连接(FC)矩阵、对模型输出应用时间滤波器、计算功率谱等。其中BoxSearch类提供参数探索功能,Evolution类启用模型优化功能。
5. 下载与使用:
在python中可直接 pip install neurolib 下载,源代码 https://github.com/neurolib-dev/neurolib 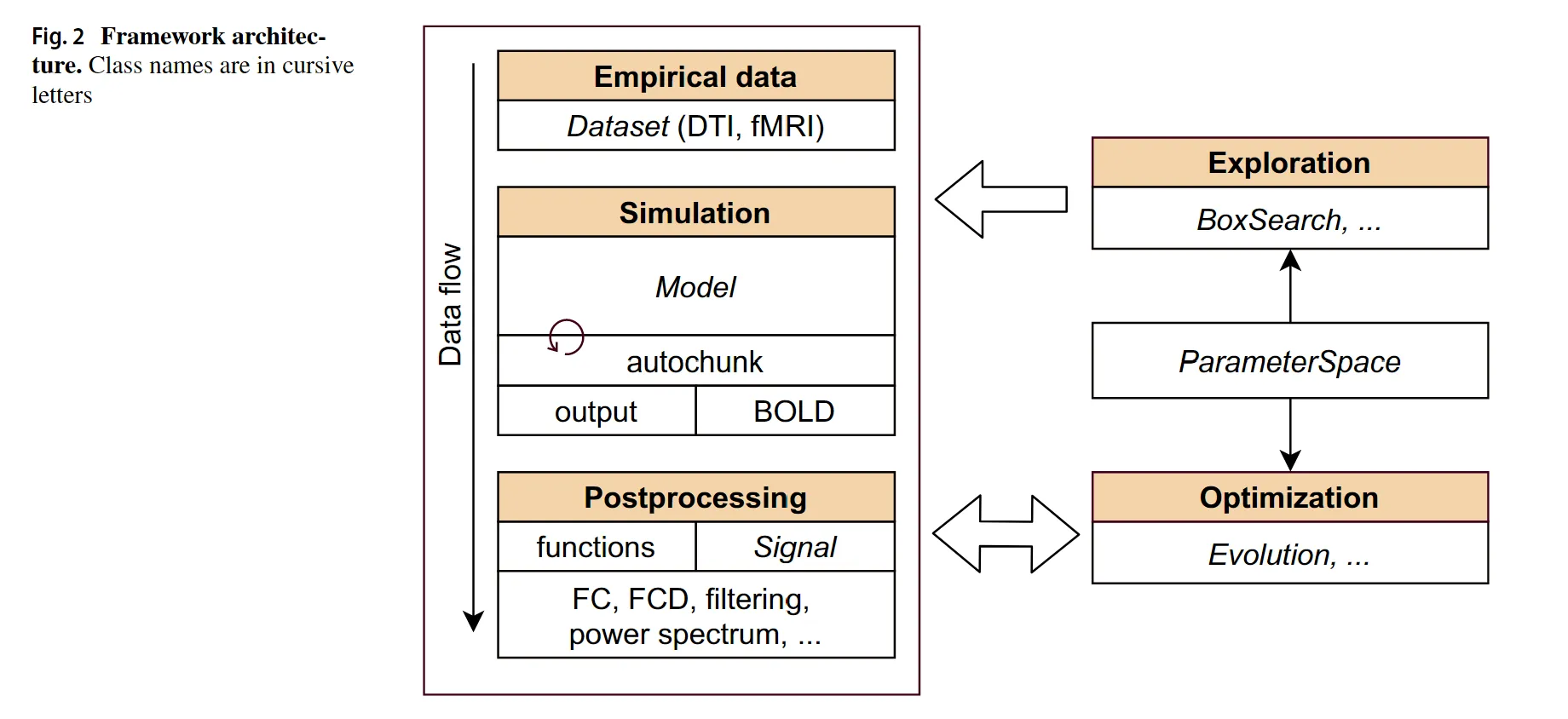
框架图如上所示,先输入数据集,然后进行模拟建模,输出模型并转成BOLD信号,之后进行后处理并于经验数据进行比较。
6. 模型方程: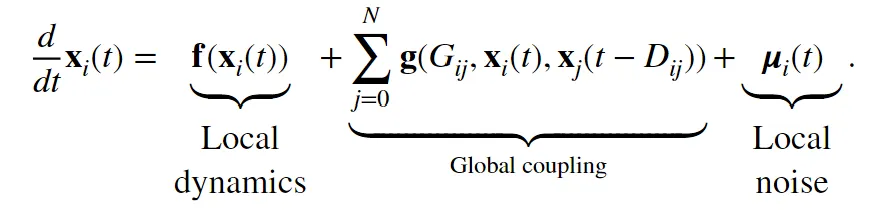
第一项是动力学f,也是模型之间的主要区别;第二项描述第i个与第j个脑区之间的耦合关系,该耦合项通常由 N × N 邻接矩阵 Gij ,第 i 个脑区的当前状态向量,以及第 j 个脑区的时延状态向量构成;第三项表示每个节点的噪声输入。
数据集
使用人类连接组计划(HCP)的ConnectomeDB中预处理过的数据。结构连接性数据以 N × N 矩阵形式存储,功能时间序列则以 N × t 矩阵形式存储,其中 N 表示脑区数量,t 表示时间步数。
其中,结构性DTI数据对于将大脑划分为N个脑区的给定分区,N×N邻接矩阵Cmat(即结构连接矩阵)确定了脑区之间的连接强度。纤维长度矩阵Dmat确定了信号传输延迟,即信号从一个脑区传播到另一个脑区所需的时间。
按脑区划分





 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章
















 3966
3966

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








