目录
2.1读取LiDAR数据使用readLAS(Lidar Scanning Data)
3.1渐进形态滤波器( Progressive Morphological Filters)
3.2 布料模拟功能(Cloth Simulation Filter)
3.3 多尺度曲率分类(Multi-Scale Curvature Classification)
数据类型计算
1简介
lidR是一个 R 包,用于操作和可视化机载激光扫描 (ALS) 数据(Airborne Laser Scanning)
主要功能:
- 读写、
.las(LiDAR Aerial Survey,las)文件.laz(LASzip)和渲染定制点云显示 - 处理点云,包括点分类(第3节)、数字地形模型(第4节)、归一化(第5节)和数字表面模型(第6节)
- 执行单独的树分割(第7节)
2阅读、绘图、查询和验证
2.1读取LiDAR数据使用readLAS
ALS 传感器记录属性数据:
- 三个维度(X、Y、Z)的位置数据
- 每个点的强度
- 返回序列中每个点的位置
- 每个点的光束入射角
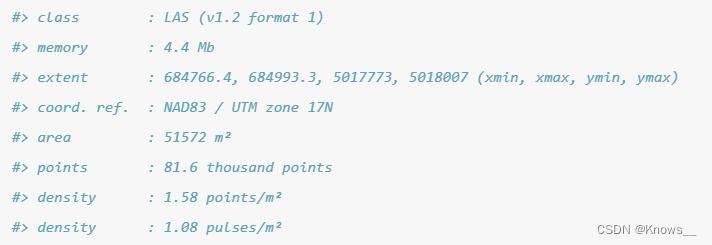 las <- readLAS("files.las")
las <- readLAS("files.las")
print(las)#显示其内容的摘要
#要更深入地打印数据,请使用函数summary()而不是print().
参数select ,节省内存,仅加载X Y Z属性
las <- readLAS("file.las", select = "xyz") # load XYZ only
las <- readLAS("file.las", select = "xyzi") # load XYZ and intensity only

参数filter 在读取时选择“行”(或点) 林业中的常见做法是使用首次返回进行处理。
las <- readLAS("file.las", filter = "-keep_first") # Read only first returns
只读第一个返回 5 到 50 m 之间(命令的完整列表readLAS(filter = "-help"))
las <- readLAS("file.las", filter = "-keep_first -drop_z_below 5 -drop_z_above 50")
#读取所有点,然后过滤 R 中的第一个返回值
las2 <- readLAS("file.las")
las2 <- filter_poi(las2, ReturnNumber == 1L)
2.2验证激光雷达数据
在深入挖掘数据之前,请始终确保运行该函数。
(检查诸如树被检测到两次、指标无效或 DTM 生成错误等问题)
las_check(las)
2.3绘图
默认情况下在黑色背景上使用 Z 坐标着色的点。
2.3.1基本 3D 渲染
plot(las)

对点进行着色的属性名称来更改用于着色的属性
# Plot las object by scan angle,
# make the background white,
# display XYZ axis and scale colors
plot(las, color = "ScanAngleRank", bg = "white", axis = TRUE, legend = TRUE)
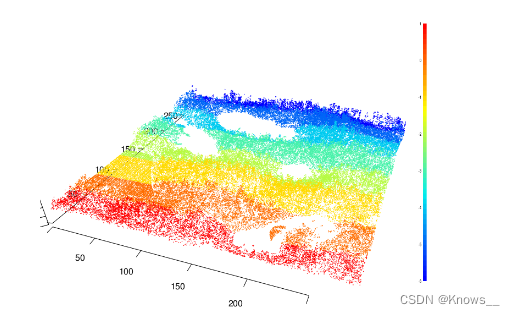
# breaks: 颜色分段方式,这里使用分位数(quantile)进行分段
plot(las, color = "Intensity", breaks = "quantile", bg = "white")

2.3.2叠加
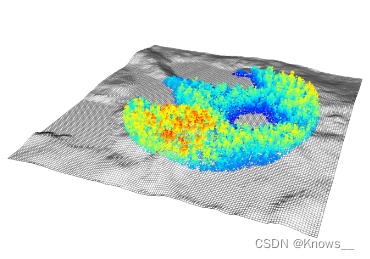
add_dtm3d(),添加数字地形模型(第4节)
add_treetops3d()可视化单个树木检测的输出(第7.1节)
x <- plot(las, bg = "white", size = 3)#点的大小为3
add_dtm3d(x, dtm)
x <- plot(las, bg = "white", size = 3)
add_treetops3d(x, ttops)
将两个具有不同调色板的点云组合起来
# 根据激光扫描数据中的分类(Classification)属性过滤非植被数据集
nonveg <- filter_poi(las, Classification != LASHIGHVEGETATION)
# 根据激光扫描数据中的分类(Classification)属性过滤高植被数据集
veg <- filter_poi(las, Classification == LASHIGHVEGETATION)
# 绘制非植被数据集的点云图像
x <- plot(nonveg, color = "Classification", bg = "white", size = 3)
# 在同一个图像上绘制高植被数据集的点云图像
plot(veg, add = x)
2.3.3高级3D渲染
# 从lidR包中读取LAS文件路径
LASfile <- system.file("extdata", "MixedConifer.laz", package = "lidR")
# 读取LAS文件并选择"xyzc"属性 #点的三维坐标和c分类属性
las <- readLAS(LASfile, select = "xyzc")
# 获取树木的位置
#lmf局部最大高度法(Local Maxima Filtering)
#ws 表示局部地形窗口(window size)
#ttops 是一个包含树木位置信息的数据结构,其中包括树木的 X、Y 和 Z 坐标
ttops <- locate_trees(las, lmf(ws = 5))
# 绘制点云图像
offsets <- plot(las, bg = "white", size = 3)
add_treetops3d(offsets, ttops)
# 提取树木的坐标并将其在渲染坐标系中进行平移
#sf::st_coordinates() 函数提取了树木位置数据 ttops 中的坐标信息
#索引 sf::st_coordinates(ttops)[, 1],我们提取了这些坐标中的第一列,即 X 坐标
#offsets[1] 表示 X 方向上的平移量
x <- sf::st_coordinates(ttops)[, 1] - offsets[1]
y <- sf::st_coordinates(ttops)[, 2] - offsets[2]
#z 变量直接从 ttops$Z 中获取了树木的 Z 坐标
z <- ttops$Z
# 构建GL_LINES矩阵以进行快速渲染
x <- rep(x, each = 2)
y <- rep(y, each = 2) # 将向量 y 中的每个元素复制两次,生成新的向量
tmp <- numeric(2 * length(z)) # 创建一个长度为 2 * length(z) 的空 numeric 向量 tmp
tmp[2 * 1:length(z)] <- z # 将向量 z 中的元素按照一定规律复制到 tmp 中,
假设 length(z) 的值为 4,那么 1:length(z) 将生成序列 [1, 2, 3, 4]。而 2*1:length(z) 将生成序列 [2, 4, 6, 8]
举例:
z <- c(1, 2, 3)
tmp <- numeric(2 * length(z)) # 创建一个长度为 2 * length(z) 的空 numeric 向量 tmp
tmp[2 * 1:length(z)] <- z # 将向量 z 中的元素按照一定规律复制到 tmp 中
z <- tmp # 将 tmp 赋值给变量 z,确保 z 的长度与 x 和 y 相等
M <- cbind(x, y, z) # 将 x、y 和 z 合并成一个矩阵 M,用于表示线段的坐标信息
# 显示连线
rgl::segments3d(M, col = "black", lwd = 2)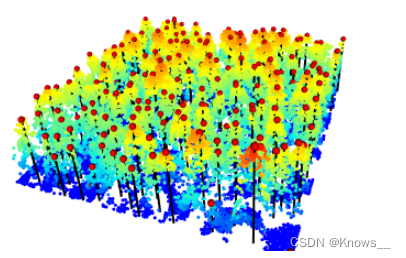
2.3.4体素渲染
vox <- voxelize_points(las, 6) #6 是指定的体素大小
plot(vox, voxel = TRUE, bg = "white")

2.3.5横截面二维渲染
可视化点云的垂直结构,先需要确定横截面的位置(即定义起点和终点)并指定它的宽度,裁剪点云,并使用X和Z坐标创建绘图
# 定义点 p1 和 p2 的坐标
p1 <- c(273357, 5274357)
p2 <- c(273542, 5274542)
# 对点云数据进行横截面剪裁
width = 4指定了剪裁的宽度为 4 个单位长度。xz = TRUE表示剪裁时考虑点云数据的 x-z 平面,即剪裁的区域是二维平面而非三维空间。
las_tr <- clip_transect(las, p1, p2, width = 4, xz = TRUE)
library(ggplot2)
# 使用 ggplot2 创建散点图
ggplot(las_tr@data, aes(X, Z, color = Z)) +
通过 las_tr@data,我们可以访问 las_tr 对象中的数据部分,即点云数据的属性数据。这些属性数据通常以数据框(data.frame)的形式存储,其中每列代表一个属性,每行代表一个点的属性值。
geom_point(size = 0.5) + # 添加散点图层,设置点的大小为 0.5
coord_equal() + # 设置坐标轴比例尺相等
theme_minimal() + # 设置简洁的主题样式
scale_color_gradientn(colours = height.colors(50)) # 设置颜色渐变范围为 50 种颜色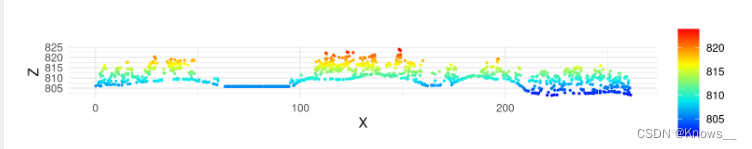
合并创建横截面
plot_crossection <- function(las,
p1 = c(min(las@data$X), mean(las@data$Y)),
p2 = c(max(las@data$X), mean(las@data$Y)),
width = 4, colour_by = NULL) {
colour_by <- rlang::enquo(colour_by) # 将传入的 colour_by 参数进行引用保护
data_clip <- clip_transect(las, p1, p2, width) # 对点云数据进行剪裁,得到剪裁后的数据
p <- ggplot(data_clip@data, aes(X, Z)) + geom_point(size = 0.5) + coord_equal() + theme_minimal() # 创建 ggplot2 的基础图形对象
if (!is.null(colour_by)) # 如果 colour_by 参数不为空
p <- p + aes(color = !!colour_by) + labs(color = "") # 添加颜色映射,并设置颜色的标签为空字符串
return(p) # 返回生成的 ggplot2 图形对象
}
3地面分类
地面点的分类——创建地形高程的连续模型
三种算法:渐进形态滤波器(PMF)、布料模拟函数(CSF)和可与该函数一起使用的多尺度曲率分类(MCC)
3.1渐进形态滤波器( Progressive Morphological Filters)
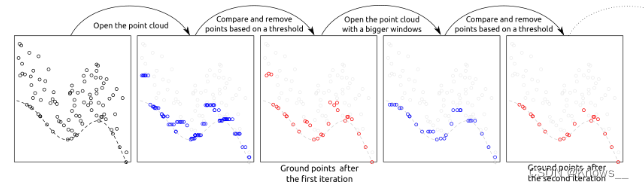
LASfile <- system.file("extdata", "Topography.laz", package="lidR")
las <- readLAS(LASfile, select = "xyzrn")
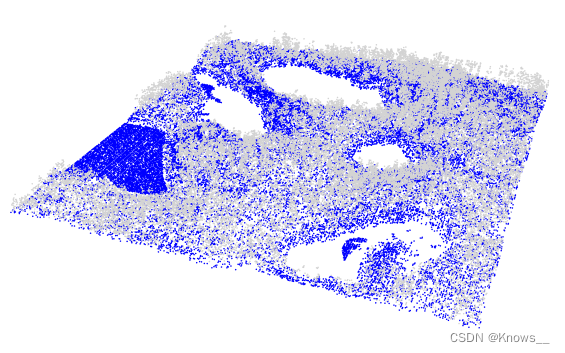
las <- classify_ground(las, algorithm = pmf(ws = 5, th = 3))#窗口大小为5,阈值为3
plot(las, color = "Classification", size = 3, bg = "white")
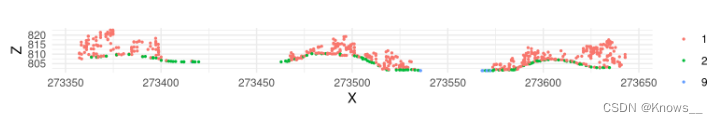
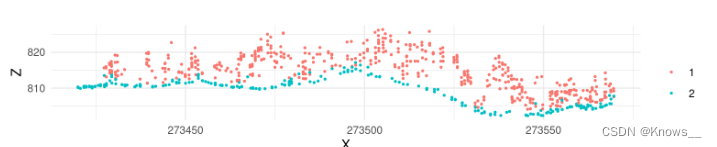
#生成并绘制点云的横截面
p1 <- c(273420, 5274455)
p2 <- c(273570, 5274460)
plot_crossection(las, p1 , p2, colour_by = factor(Classification))
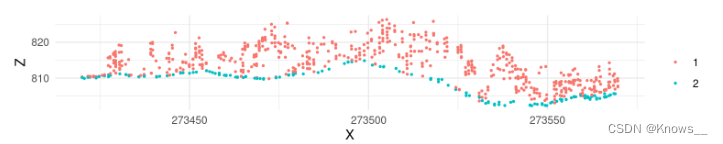
# 设置窗口大小的序列,从3到12,步长为3 ws <- seq(3, 12, 3) # 根据窗口大小序列长度生成阈值序列,从0.1到1.5,使得阈值序列与窗口大小序列长度一致 th <- seq(0.1, 1.5, length.out = length(ws))
ws <- seq(3, 12, 3)
th <- seq(0.1, 1.5, length.out = length(ws))
las <- classify_ground(las, algorithm = pmf(ws = ws, th = th))
plot_crossection(las, p1 = p1, p2 = p2, colour_by = factor(Classification))
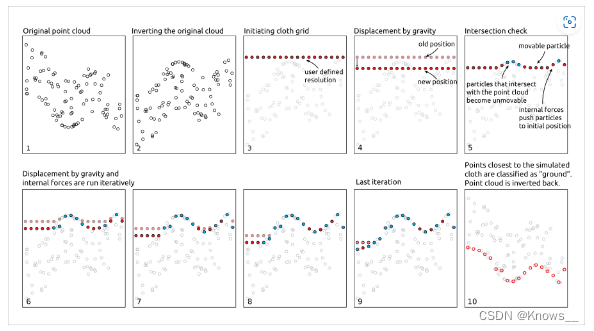
3.2 布料模拟功能(Cloth Simulation Filter)
包括模拟覆盖在反向点云上的一块布。在这种方法中,将点云倒置,然后将一块布掉在倒置的表面上。通过分析布料节点与倒置表面之间的相互作用来确定接地点。布料模拟本身基于一个网格,该网格由具有质量和互连的粒子组成,这些粒子共同决定了布料的三维位置和形状。
las <- classify_ground(las, algorithm = csf())
plot_crossection(las, p1 = p1, p2 = p2, colour_by = factor(Classification))
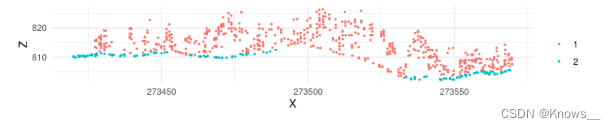
该算法在我们的示例中无法正常工作
mycsf <- csf(sloop_smooth = TRUE, class_threshold = 1, cloth_resolution = 1, time_step = 1)
las <- classify_ground(las, mycsf)
plot_crossection(las, p1 = p1, p2 = p2, colour_by = factor(Classification))
gnd <- filter_ground(las)
plot(gnd, size = 3, bg = "white")
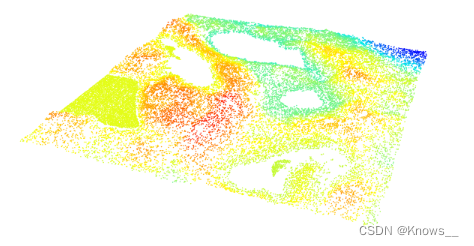
3.3 多尺度曲率分类(Multi-Scale Curvature Classification)
las <- classify_ground(las, mcc(1.5,0.3))
plot_crossection(las, p1 = p1, p2 = p2, colour_by = factor(Classification))
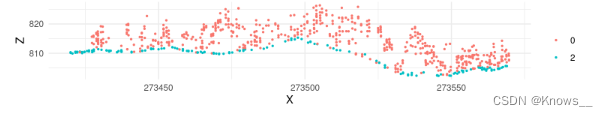
3.4 边缘工件
3.5 如何选择方法及其参数?
3.6 其他方法
地面分割不限于上述 3 种方法
las <- classify_ground(las, algorithm = my_new_method(param1 = 0.05))4数字地形模型
生成数字地形模型 (DTM) 通常是地面点分类之后的处理的第二步,使用户能够标准化点云,即从点的高程中减去局部地形,以允许对点云进行操作,就好像它们是在平坦的表面上获取的一样
LASfile <- system.file("extdata", "Topography.laz", package="lidR")
las <- readLAS(LASfile, select = "xyzc")
plot(las, size = 3, bg = "white")
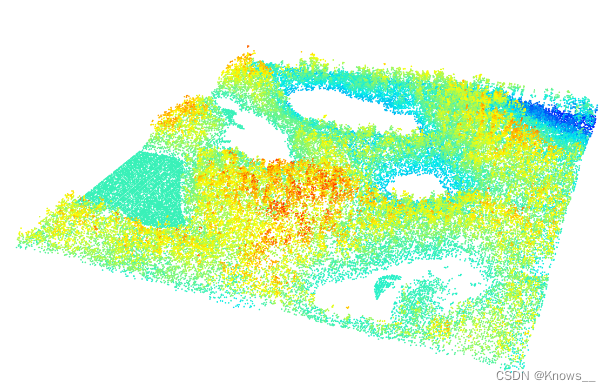
4.1 三角不规则网络
要使用 TIN 算法生成 DTM 模型,我们使用其中 .rasterize_terrain()algorithm = tin()
dtm_tin <- rasterize_terrain(las, res = 1, algorithm = tin())
plot_dtm3d(dtm_tin, bg = "white")

4.2 反转距离加权
要使用 IDW 算法生成 DTM 模型,我们使用 其中 .rasterize_terrain()algorithm = knnidw()
dtm_idw <- rasterize_terrain(las, algorithm = knnidw(k = 10L, p = 2))
plot_dtm3d(dtm_idw, bg = "white")

4.3 克里金法
要使用克里金算法生成 DTM 模型,我们使用 其中 .rasterize_terrain()algorithm = kriging()
请注意,该算法在插值缺少点的区域(如湖泊)时存在问题。
dtm_kriging <- rasterize_terrain(las, algorithm = kriging(k = 40))
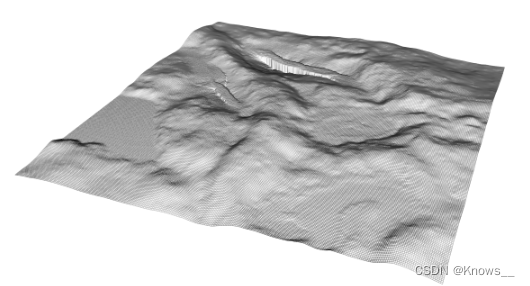
plot_dtm3d(dtm_kriging, bg = "white")
4.4 优缺点
三角测量是一种非常快速有效的方法,可以生成非常好的 DTM,并且对点云内的空白区域具有鲁棒性。然而,它在边缘很弱。
反转距离加权速度很快,但大约是 TIN 的两倍。
-
克里金法非常慢,因为它对计算要求很高。不建议在大中型区域使用。它可用于没有缓冲区的小图,以获得没有强边伪影的漂亮 DTM。
4.5 其他方法
MBA 包创建基于多级 B 样条近似 (MBA) 的插件算法。rasterize_terrain()
dtm_mba <- rasterize_terrain(las, algorithm = mba())
plot_dtm3d(dtm_mba, bg = "white")
4.6 渲染着色 DTM
在 R 中生成山体阴影层相对简单,并且使用包中的函数完成。可以组合 and 函数以将 DTM 栅格图层作为输入并返回山体阴影栅格:terraterrain()hillShade()
library(terra)
#> terra 1.5.34
# 使用lidR包中的LAS文件创建一个数字地面模型(DTM)
dtm <- rasterize_terrain(las, algorithm = tin(), pkg = "terra")
# 基于DTM计算坡度(slope)和坡向(aspect)
dtm_prod <- terrain(dtm, v = c("slope", "aspect"), unit = "radians")
# 根据坡度和坡向生成山体阴影
dtm_hillshade <- shade(slope = dtm_prod$slope, aspect = dtm_prod$aspect)
# 绘制山体阴影图像,使用灰度颜色映射,并隐藏图例
plot(dtm_hillshade, col = gray(0:30/30), legend = FALSE)
光线着色器包还提供了有趣的工具来生成着色 DTM。dtm 必须是RasterLayer
library(rayshader)
# 将dtm转换为raster对象
dtm <- raster::raster(dtm)
# 将raster对象转换为矩阵
elmat <- raster_to_matrix(dtm)
# 创建地形表面,并应用纹理和水体效果
map <- elmat %>%
sphere_shade(texture = "imhof1", progbar = FALSE) %>%
add_water(detect_water(elmat), color = "imhof1")
# 添加阴影效果
map <- map %>%
add_shadow(ray_shade(elmat, progbar = FALSE), 0.5) %>%
add_shadow(ambient_shade(elmat, progbar = FALSE), 0)
plot_map(map)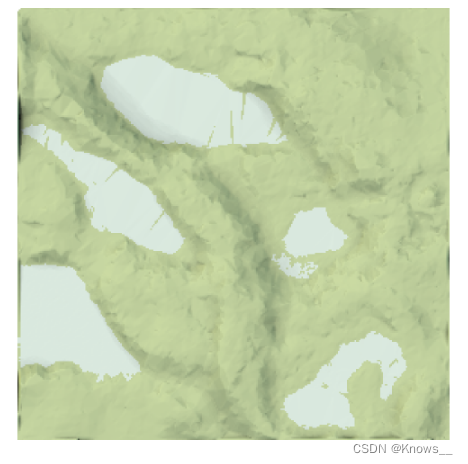
plot_3d(map, elmat, zscale = 1, windowsize = c(800, 800))
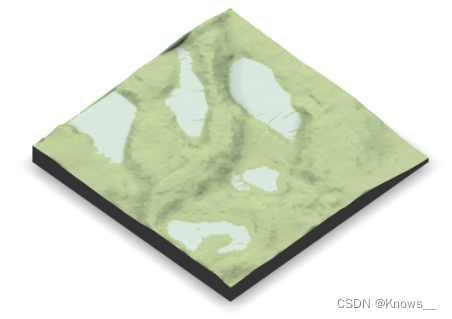
5 高程归一化
点云归一化消除了地形对地上测量的影响。这使得比较地上植被高度成为可能
LASfile <- system.file("extdata", "Topography.laz", package="lidR")
las <- readLAS(LASfile)
plot(las, size = 3, bg = "white")
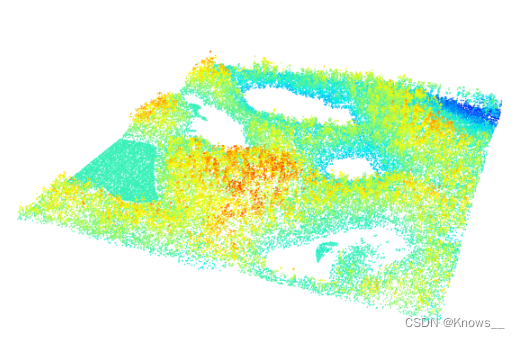
gnd <- filter_ground(las)
plot(gnd, size = 3, bg = "white", color = "Classification")

5.1 DTM 规范化
# 使用lidR包中的LAS文件创建数字地面模型(DTM)
dtm <- rasterize_terrain(las, 1, knnidw())
# 绘制DTM,使用灰度颜色映射
plot(dtm, col = gray(1:50/50))1:50 生成一个从 1 到 50 的整数向量,表示灰度级别。然后,/50 将该整数向量除以 50,将其范围缩放到 0 到 1 之间
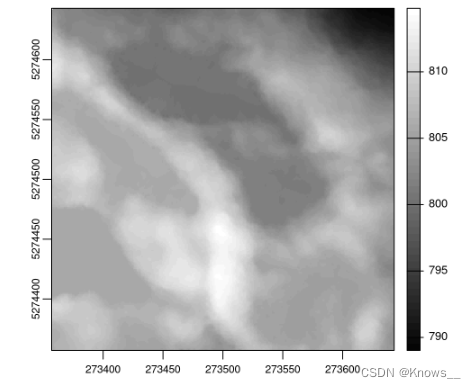
nlas <- las - dtm
plot(nlas, size = 4, bg = "white")

hist(filter_ground(nlas)$Z, breaks = seq(-0.6, 0.6, 0.01), main = "", xlab = "Elevation")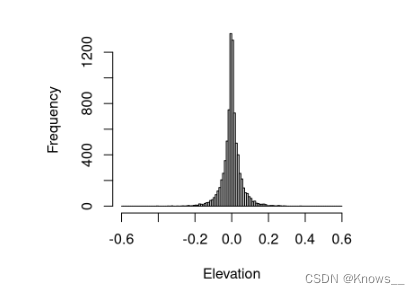
5.2 点云归一化
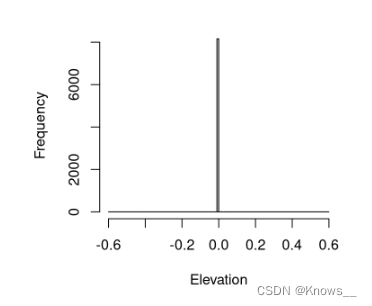
nlas <- normalize_height(las, knnidw())
hist(filter_ground(nlas)$Z, breaks = seq(-0.6, 0.6, 0.01), main = "", xlab = "Elevation")
5.3 混合法
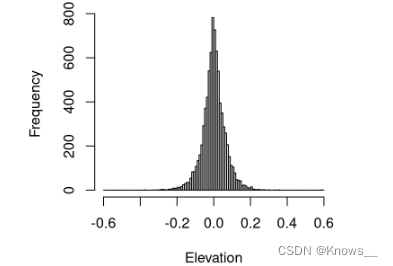
nlas <- las - dtm进行非常简单的减法,无需任何插值。 动态计算地面点的插值,并估计每个点的确切高程。混合方法包括对已计算的 DTM 的像素进行插值。normalize_height(las, knnidw())
nlas <- normalize_height(las, tin(), dtm = dtm)
hist(filter_ground(nlas)$Z, breaks = seq(-0.6, 0.6, 0.01), main = "", xlab = "Elevation")#main = "" 是 hist() 函数的参数,用于设置直方图的主标题。
5.4 优点和缺点
5.5 反转归一化
lidR还具有使用该函数反向规范化的能力。这会将归一化点云恢复到其归一化前的状态。unnormalize_height
<span style="color:#333333"><span style="background-color:#ffffff"><span style="background-color:#f7f7f7"><span style="color:inherit"><code><span style="color:inherit">las <span style="color:#007020"><-</span> <span style="color:#06287e">unnormalize_height</span>(nlas)</span></code></span></span></span></span>6 数字表面模型和冠层高度模型
数字表面模型 (DSM) 和冠层高度模型 (CHM) 是栅格图层
在归一化点云的情况下,派生表面表示冠层高度(对于植被区域),称为CHM
存在不同的方法来创建 DSM 和 CHM。最简单的--点到栅格
LASfile <- system.file("extdata", "MixedConifer.laz", package ="lidR")
las <- readLAS(LASfile)
plot(las, size = 3, bg = "white")

6.1 点到栅格
点云数据(三维坐标点)转换为栅格数据(二维网格)
点云转栅格:常见的算法包括 Point-to-Raster (P2R)、Pit-free、Kriging、TIN
-
确定栅格的分辨率:首先需要确定生成的栅格的分辨率,即每个栅格单元格的大小。分辨率的选择取决于所需的精度和应用场景。
-
创建空的栅格:根据给定的栅格分辨率,创建一个空的栅格数据结构,其中包含指定分辨率下的栅格单元格。
-
将点云数据投影到栅格上:对于每个点云数据点,将其投影到对应的栅格单元格中。可以使用插值方法(如最近邻、三角插值等)来确定点在栅格单元格内的值。
-
处理重叠点:如果多个点投影到同一个栅格单元格中,需要进行处理。常见的方法包括计算平均值、最大值或使用优先级规则来选择最终的值。
-
栅格化结果:将经过投影和处理的点云数据赋值给栅格单元格,形成最终的栅格数据。每个栅格单元格可以表示某种属性,如高度、强度等。
# 使用lidR包中的LAS文件创建冠层高度模型(CHM),res = 1 指定了栅格分辨率为1,意味着每个像素在实际空间中代表一个单位的距离
chm <- rasterize_canopy(las, res = 1, algorithm = p2r())
# 定义颜色映射,其中25代表颜色级别的数量。
col <- height.colors(25)
# 绘制冠层高度模型,,颜色映射(Color map)是一种将数据值映射到特定颜色的技术。
plot(chm, col = col)
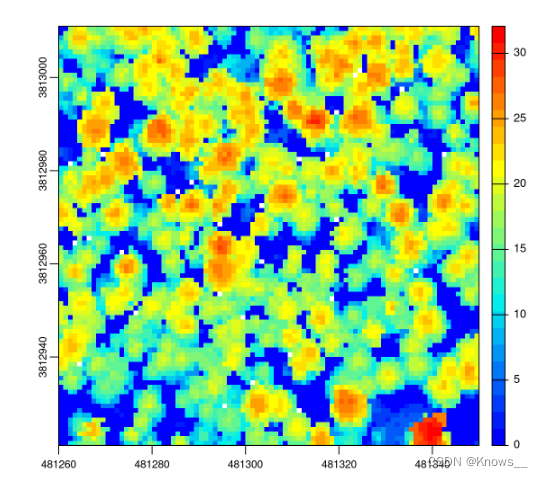
点到栅格方法的一个缺点;格网分辨率对于可用点密度来说太细,则某些像素可能为空
chm <- rasterize_canopy(las, res = 0.5, algorithm = p2r())
plot(chm, col = col)
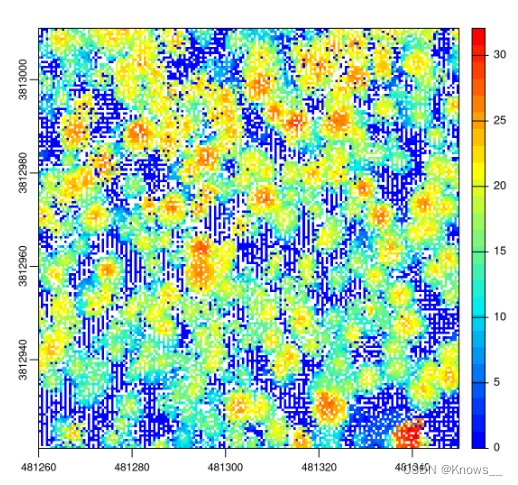
#点云中的每个点替换为已知半径(例如 15 厘米)的圆盘
chm <- rasterize_canopy(las, res = 0.5, algorithm = p2r(subcircle = 0.15))
plot(chm, col = col)
放大以查看替换每个点的小圆盘
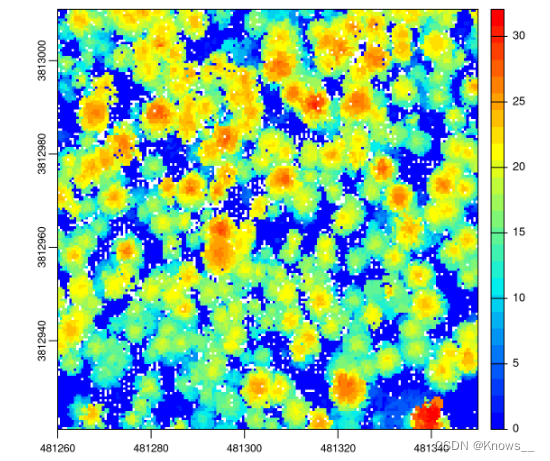
p2r(0.2, na.fill = tin()) 是栅格化算法的参数,其中 0.2 是 p2r 算法中子圆的半径大小,相对于栅格分辨率;na.fill = tin() 表示使用TIN(三角形不规则网格)算法来填充栅格中未插值的空值(NA)。
chm <- rasterize_canopy(las, res = 0.5, p2r(0.2, na.fill = tin()))
plot(chm, col = col)
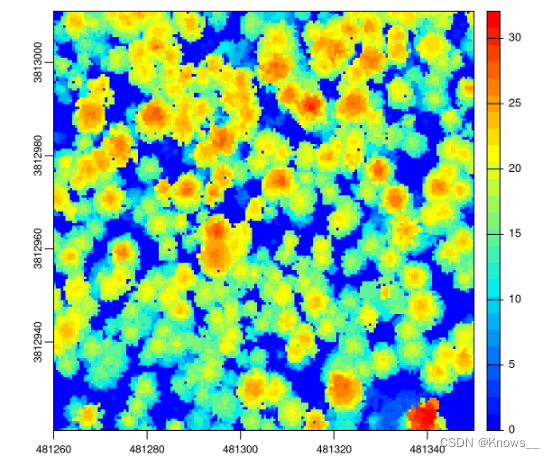
6.2 三角测量
要使用三角测量创建曲面模型,我们使用 .algorithm = dsmtin()
chm <- rasterize_canopy(las, res = 0.5, algorithm = dsmtin())
plot(chm, col = col)
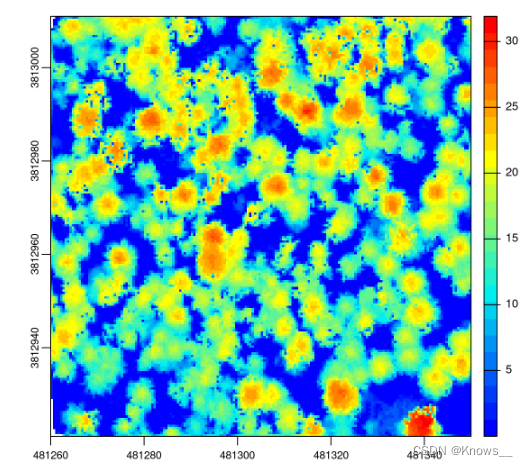
缺少很多点时,三角测量方法也可能很弱
LASfile <- system.file("extdata", "Topography.laz", package = "lidR")
las2 <- readLAS(LASfile)
las2 <- normalize_height(las2, algorithm = tin())
plot(las2, size = 3, bg = "white")
chm <- rasterize_canopy(las2, res = 0.5, algorithm = dsmtin())
plot(chm, col = col)
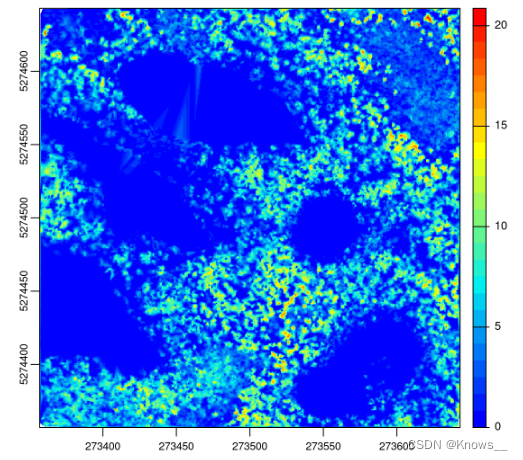
参数 Delaunay 如果设置为例如 8,则意味着每个边长于 8 的三角形都将从三角测量中丢弃
chm <- rasterize_canopy(las2, res = 0.5, algorithm = dsmtin(max_edge = 8))
plot(chm, col = col)
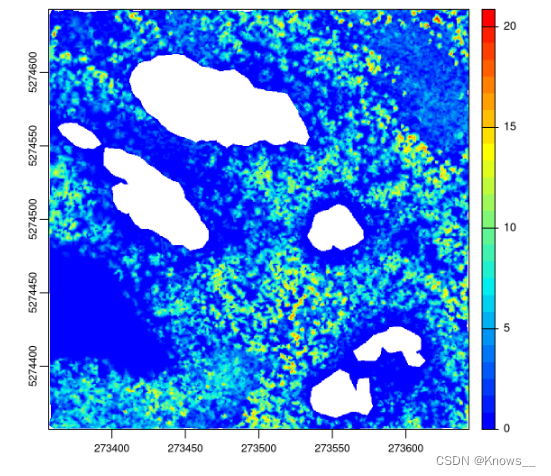
6.3 无坑算法
该算法由一系列连续的高度阈值组成,其中Delaunay三角测量应用于首次返回。对于每个阈值,三角测量中都会清除太大的三角形。
pitfree()
chm <- rasterize_canopy(las, res = 0.5, pitfree(thresholds = c(0, 10, 20), max_edge = c(0, 1.5)))
plot(chm, col = col)
# 第一层是常规三角化
layer0 <- rasterize_canopy(las, res = 0.5, algorithm = dsmtin())
# 高于10米的第一次返回点云的三角化
above10 <- filter_poi(las, Z >= 10)
layer10 <- rasterize_canopy(above10, res = 0.5, algorithm = dsmtin(max_edge = 1.5))
# 高于20米的第一次返回点云的三角化
above20 <- filter_poi(above10, Z >= 20)
layer20 <- rasterize_canopy(above20, res = 0.5, algorithm = dsmtin(max_edge = 1.5))
# 最终的表面是部分栅格的堆叠
dsm <- layer0
dsm[] <- pmax(as.numeric(layer0[]), as.numeric(layer10[]), as.numeric(layer20[]), na.rm = TRUE)
layers <- c(layer0, layer10, layer20, dsm)
names(layers) <- c("Base", "Layer10m", "Layer20m", "pitfree")
plot(layers, col = col)
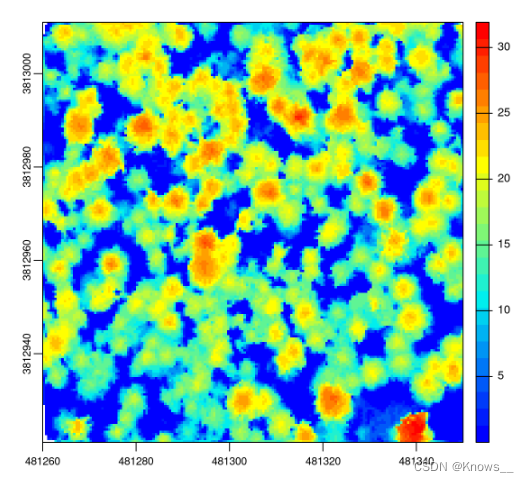
chm <- rasterize_canopy(las, res = 0.5, pitfree(max_edge = c(0, 2.5)))
plot(chm, col = col)
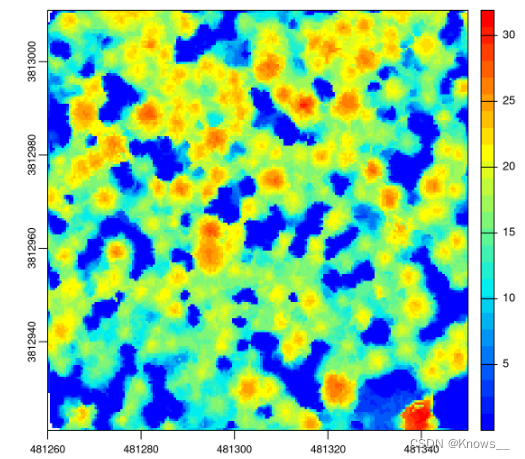
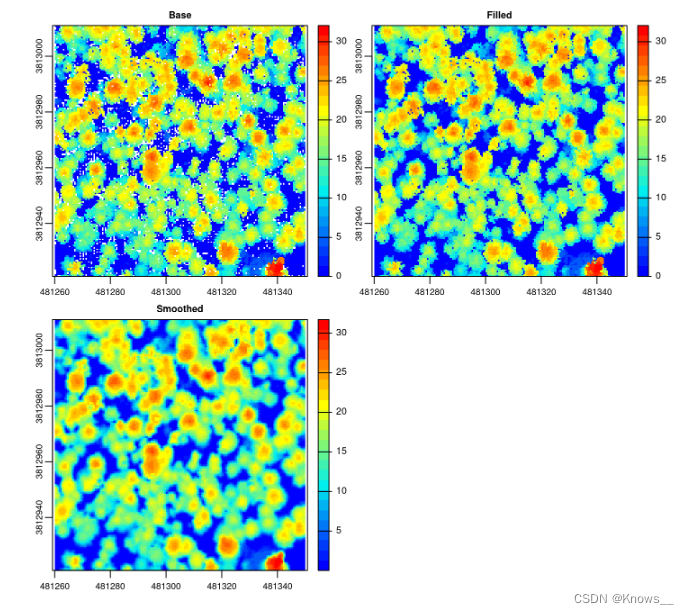
6.4 信息交换所机制的后处理
填充 NA 并使用包平滑 CHM
fill.na <- function(x, i=5) { if (is.na(x)[i]) { return(mean(x, na.rm = TRUE)) } else { return(x[i]) }}
w <- matrix(1, 3, 3)
chm <- rasterize_canopy(las, res = 0.5, algorithm = p2r(subcircle = 0.15), pkg = "terra")
filled <- terra::focal(chm, w, fun = fill.na)
smoothed <- terra::focal(chm, w, fun = mean, na.rm = TRUE)
chms <- c(chm, filled, smoothed)
names(chms) <- c("Base", "Filled", "Smoothed")
plot(chms, col = col)
7 单树的划分和分割
单个树木检测 (ITD) 是在空间上定位树木和提取高度信息的过程。
lidRlocate_trees()segment_trees()MixedConifer.lazlidR
LASfile <- system.file("extdata", "MixedConifer.laz", package = "lidR") # LAS文件路径"r"通常代表点的反射强度。
las <- readLAS(LASfile, select = "xyzr", filter = "-drop_z_below 0") # 读取LAS文件并选择"xyzr"字段,排除高程值小于0的点()
chm <- rasterize_canopy(las, 0.5, pitfree(subcircle = 0.2)) # 将点云数据转换为栅格数据,使用0.5的分辨率和pitfree算法进行处理,子圆大小为0.2
plot(las, bg = "white", size = 4) # 绘制原始的点云数据,背景颜色为白色,点的大小为4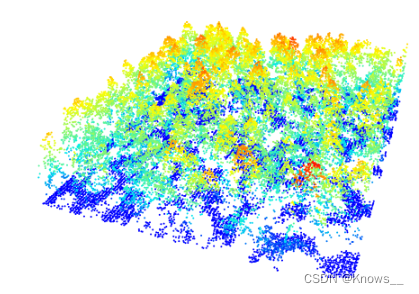
7.1 单个树木检测
在进行单木分割时,通常会使用一些算法和技术来识别和分割树木。以下是一些常见的单木分割方法:
-
基于高度阈值:通过设定一个高度阈值,将点云中高度超过该阈值的点划分为一个独立的树木对象。这种方法适用于树木的高度差异较大的情况。
-
基于聚类:使用聚类算法,如DBSCAN、MeanShift等,将点云数据中的点按照其在空间上的密度分布进行分割。密集的点被认为是同一个树木的一部分。
-
基于形态学分析:利用形态学操作,如开运算、闭运算等,对点云数据进行处理,提取树木的形状特征,从而实现树木的分割。
-
基于区域生长:从点云数据中的种子点开始,通过生长算法将相邻的点逐步加入到同一个树木对象中,直到无法再生长为止。
开运算和闭运算是形态学图像处理中常用的两种基本操作。
开运算(Opening)是由一个腐蚀(Erosion)操作后接一个膨胀(Dilation)操作组成的操作序列。它的作用是去除图像中的小型噪声、填充小的空洞和细小的断裂。开运算先对图像进行腐蚀操作,将物体边缘向内缩小,然后再进行膨胀操作,使边缘恢复到原始位置。这样可以平滑物体的轮廓并保持其整体形状,同时去除了细小的细节。
闭运算(Closing)与开运算相反,是由一个膨胀操作后接一个腐蚀操作组成的操作序列。它的作用是填充图像中的小型空洞、连接断裂的物体边缘。闭运算先对图像进行膨胀操作,将物体边缘向外扩展,然后再进行腐蚀操作,使边缘恢复到原始位置。这样可以填充空洞、连接断裂,并保持物体的整体形状。
开运算和闭运算常用于去除图像中的噪声、平滑图像边缘、填充空洞、连接断裂等图像处理任务。在点云数据的处理中,开运算和闭运算可以应用于树木分割、地形分析、对象提取等方面,以提升数据质量和准确性。
腐蚀是一种图像处理操作,用于减小图像中物体的边界或厚度。在腐蚀操作中,图像中的每个像素都会与其周围的像素进行比较,然后根据定义的结构元素(也称为内核)进行处理。
腐蚀操作的基本思想是将结构元素与图像进行卷积运算,如果结构元素覆盖的所有像素都是前景像素(白色像素),那么中心像素将保持不变,否则,中心像素将被置为背景像素(黑色像素)。
腐蚀操作可以用于图像处理的多个应用,包括但不限于:
- 去除图像中的小噪点或细小的物体。
- 缩小物体的边界或厚度。
- 分离粘连在一起的物体。
在点云数据处理中,腐蚀操作也可以应用于类似的场景,例如去除点云数据中的噪声点、减小点云数据中物体的边界或厚度等。
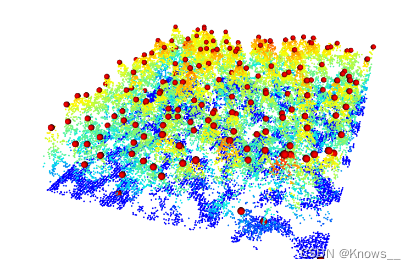
本地最大过滤器 (LMF) 来检测树顶(Local Maximum Filter)
(对于给定点,该算法分析邻域点,检查处理后的点是否最高)
7.1.1 固定窗口大小的局部最大过滤器
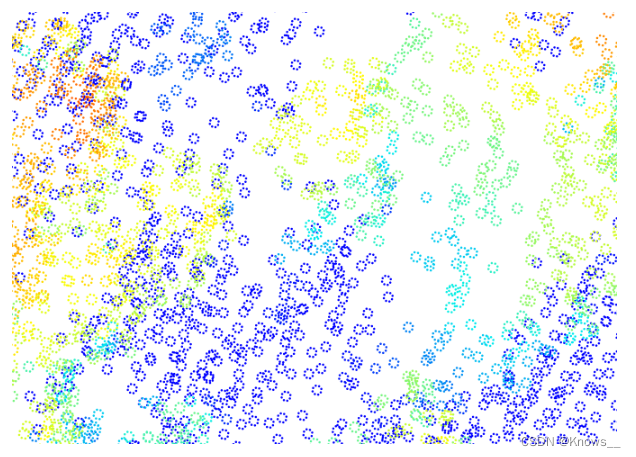
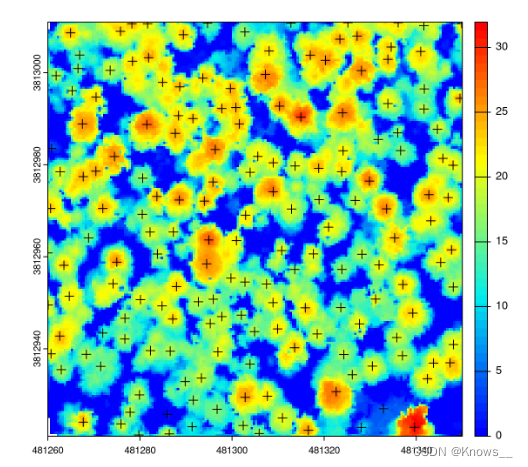
ttops <- locate_trees(las, lmf(ws = 5))
plot(chm, col = height.colors(50))
#st_geometry 是 sf 包中的一个函数,用于提取空间对象(spatial object)的几何信息
plot(sf::st_geometry(ttops), add = TRUE, pch = 3)#使用三角形标记(pch = 3)表示树木的位置。#ttops 是一个数据对象,它包含了点云数据中被识别为树木的位置信息。
#add_treetops3d 函数会将树木位置的几何信息与先前绘制的点云数据相结合,以可视化方式展示树木位置。
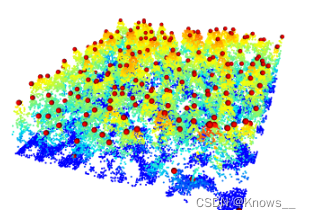
x <- plot(las, bg = "white", size = 4)
add_treetops3d(x, ttops)
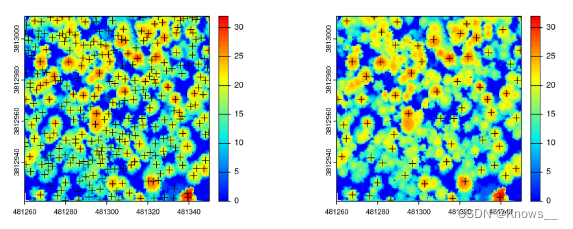
ttops_3m <- locate_trees(las, lmf(ws = 3))
ttops_11m <- locate_trees(las, lmf(ws = 11))
par(mfrow=c(1,2))#mfrow=c(1,2),将图形设备划分为 1 行和 2 列的子图网格。
plot(chm, col = height.colors(50))
plot(sf::st_geometry(ttops_3m), add = TRUE, pch = 3)
plot(chm, col = height.colors(50))
plot(sf::st_geometry(ttops_11m), add = TRUE, pch = 3)
7.1.2 具有可变窗口大小的局部最大过滤器
可变窗口尺寸特别适用于结构更复杂的林分,或者在较大区域进行树木检测时,覆盖异质林分。
最小和最大窗口大小应该与最小和最大树高度相关
f <- function(x) {x * 0.1 + 3}
heights <- seq(0,30,5)
ws <- f(heights)
plot(heights, ws, type = "l", ylim = c(0,6))
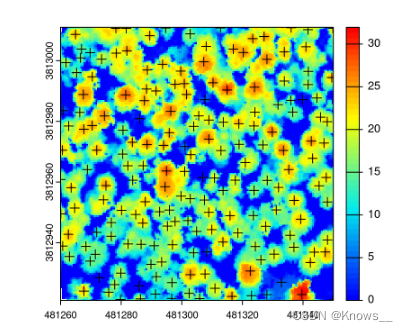
ttops <- locate_trees(las, lmf(f))
plot(chm, col = height.colors(50))
plot(sf::st_geometry(ttops), add = TRUE, pch = 3)
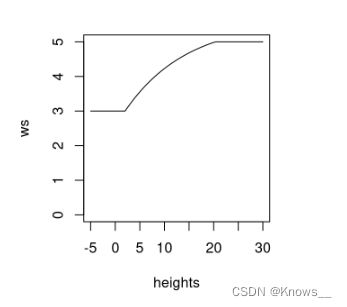
内置阈值(2 到 20 米之间的任何内容都将具有非线性关系)
f <- function(x) {
y <- 2.6 * (-(exp(-0.08*(x-2)) - 1)) + 3
y[x < 2] <- 3
y[x > 20] <- 5
return(y)
}
heights <- seq(-5,30,0.5)
ws <- f(heights)
plot(heights, ws, type = "l", ylim = c(0,5))
7.1.3 CHM上的局部最大滤波器
#pkg 是一个参数,用于指定使用的软件包或包名称
#pkg = "terra",代码将使用 Terra 软件包中的函数和功能来执行栅格化操作
首先生成不同的 CHM:
# Point-to-raster 2 resolutions
chm_p2r_05 <- rasterize_canopy(las, 0.5, p2r(subcircle = 0.2), pkg = "terra")
chm_p2r_1 <- rasterize_canopy(las, 1, p2r(subcircle = 0.2), pkg = "terra")
# Pitfree with and without subcircle tweak
chm_pitfree_05_1 <- rasterize_canopy(las, 0.5, pitfree(), pkg = "terra")
chm_pitfree_05_2 <- rasterize_canopy(las, 0.5, pitfree(subcircle = 0.2), pkg = "terra")
# Post-processing median filter
kernel <- matrix(1,3,3)
chm_p2r_05_smoothed <- terra::focal(chm_p2r_05, w = kernel, fun = median, na.rm = TRUE)
chm_p2r_1_smoothed <- terra::focal(chm_p2r_1, w = kernel, fun = median, na.rm = TRUE)
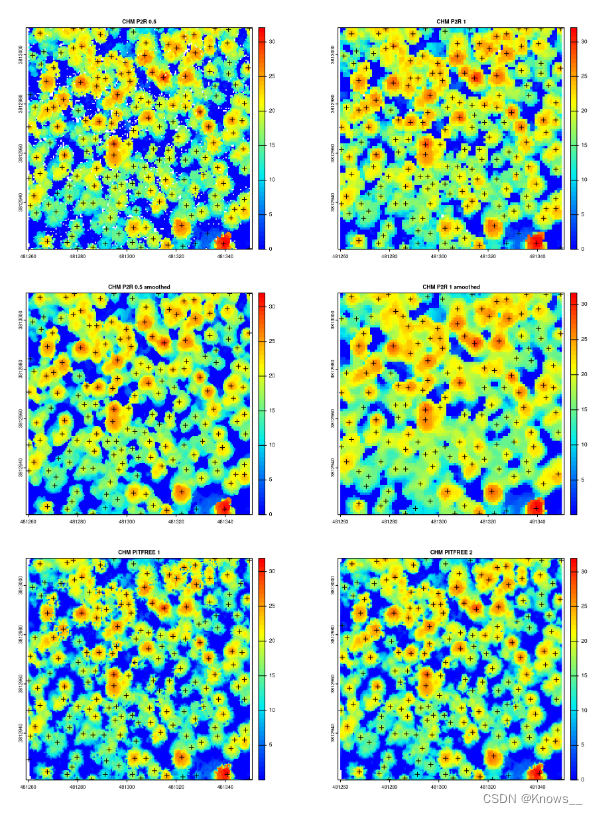
然后将具有 5 m 恒定窗口大小的相同树检测例程应用于每个 CHM:
ttops_chm_p2r_05 <- locate_trees(chm_p2r_05, lmf(5))
ttops_chm_p2r_1 <- locate_trees(chm_p2r_1, lmf(5))
ttops_chm_pitfree_05_1 <- locate_trees(chm_pitfree_05_1, lmf(5))
ttops_chm_pitfree_05_2 <- locate_trees(chm_pitfree_05_2, lmf(5))
ttops_chm_p2r_05_smoothed <- locate_trees(chm_p2r_05_smoothed, lmf(5))
ttops_chm_p2r_1_smoothed <- locate_trees(chm_p2r_1_smoothed, lmf(5))
最后,将检测结果可视化,以查看作为CHM建筑选择函数的各种输出:
par(mfrow=c(3,2))
col <- height.colors(50)
plot(chm_p2r_05, main = "CHM P2R 0.5", col = col); plot(sf::st_geometry(ttops_chm_p2r_05), add = T, pch =3)
plot(chm_p2r_1, main = "CHM P2R 1", col = col); plot(sf::st_geometry(ttops_chm_p2r_1), add = T, pch = 3)
plot(chm_p2r_05_smoothed, main = "CHM P2R 0.5 smoothed", col = col); plot(sf::st_geometry(ttops_chm_p2r_05_smoothed), add = T, pch =3)
plot(chm_p2r_1_smoothed, main = "CHM P2R 1 smoothed", col = col); plot(sf::st_geometry(ttops_chm_p2r_1_smoothed), add = T, pch =3)
plot(chm_pitfree_05_1, main = "CHM PITFREE 1", col = col); plot(sf::st_geometry(ttops_chm_pitfree_05_1), add = T, pch =3)
plot(chm_pitfree_05_2, main = "CHM PITFREE 2", col = col); plot(sf::st_geometry(ttops_chm_pitfree_05_2), add = T, pch =3)
7.2 单个树分割(ITS)
进一步细分和提取每棵树
两个系列:
- 基于云的点,无需 CHM 即可执行。
- 使用 CHM 执行的基于光栅的。
每个家庭可以分为两个子家庭
- 分两步工作的算法 - 单个树检测,然后进行分割。
- 多合一的算法。
7.2.1 点云的分割
# 使用 dalponte2016 算法处理平滑后的 chm 数据
algo <- dalponte2016(chm_p2r_05_smoothed, ttops_chm_p2r_05_smoothed)
# 对点云数据进行树木分割,使用前面得到的 algo 变量
las <- segment_trees(las, algo)
# 可视化树木分割后的点云数据
plot(las, bg = "white", size = 4, color = "treeID")
每个点分配一个 ID
tree110 <- filter_poi(las, treeID == 110)
plot(tree110, size = 8, bg = "white")
segment_trees()crown_metrics()
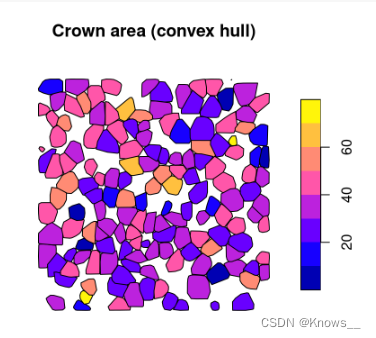
此函数计算每棵树的船体(凸或凹)
凸包是一种凸多边形,它是将树冠周边的边界点连接而成的最小凸多边形
# 计算树冠指标
crowns <- crown_metrics(las, func = .stdtreemetrics, geom = "convex")
# 绘制树冠面积(凸包面积)
plot(crowns["convhull_area"], main = "Crown area (convex hull)")
7.2.2 信息交换所机制的细分
无论是使用点云还是栅格,分割结果都将完全相同。区别在于分割结果的数据格式。
algo <- dalponte2016(chm_p2r_05_smoothed, ttops_chm_p2r_05_smoothed)
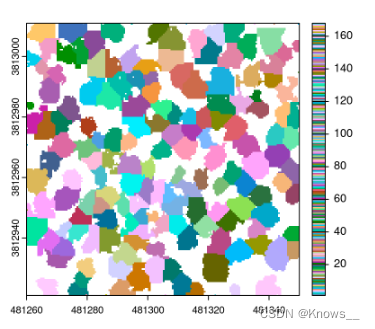
crowns <- algo()
plot(crowns, col = pastel.colors(200))
7.2.3 树形分割的比较
dalponte2016 算法是一种用于分割点云数据中的树木的算法,主要用于提取树冠的几何信息。它基于多分辨率分割方法,通过将点云数据分割成不同尺度的子集,并结合形态学处理和规则过滤来识别和分割树冠。
该算法的主要步骤包括:
-
创建高程模型:将点云数据转换为栅格数据,生成树冠高程模型。
-
初始化树冠:通过对高程模型进行开运算和闭运算,去除噪点并平滑树冠的形状。
-
多尺度分割:将初始化的树冠从粗到细进行多次分割,逐渐细化树冠的边界。
-
树冠形状修正:对分割后的树冠形状进行修正,以更准确地表示树冠的几何形状。
-
树冠合并:合并相邻的树冠,去除重叠部分,得到最终的树冠分割结果。
# 使用dalponte2016算法进行单木分割
algo1 <- dalponte2016(chm_p2r_05_smoothed, ttops_chm_p2r_05_smoothed)
las <- segment_trees(las, algo1, attribute = "IDdalponte")
# 使用li2012算法进行单木分割
algo2 <- li2012()
las <- segment_trees(las, algo2, attribute = "IDli")
# 绘制使用dalponte2016算法进行单木分割的结果
x <- plot(las, bg = "white", size = 4, color = "IDdalponte", colorPalette = pastel.colors(200))
# 绘制使用li2012算法进行单木分割的结果,并在同一图上添加到dalponte2016结果的右侧
plot(las, add = x + c(100,0), bg = "white", size = 4, color = "IDli", colorPalette = pastel.colors(200))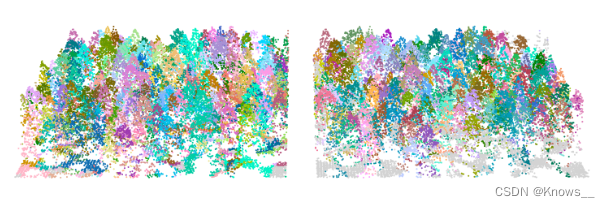
# 使用凹多边形几何形状计算dalponte2016算法的树冠指标
crowns_dalponte <- crown_metrics(las, func = NULL, attribute = "IDdalponte", geom = "concave")
# 使用凹多边形几何形状计算li2012算法的树冠指标
crowns_li <- crown_metrics(las, func = NULL, attribute = "IDli", geom = "concave")
# 设置绘图参数,创建一个一行两列的绘图布局,同时将边距的上、下、左、右四个参数都设置为0
par(mfrow=c(1,2), mar=rep(0,4))
# 绘制dalponte2016算法的树冠结果,不重置绘图参数
plot(sf::st_geometry(crowns_dalponte), reset = FALSE)
# 绘制li2012算法的树冠结果,不重置绘图参数
plot(sf::st_geometry(crowns_li), reset = FALSE)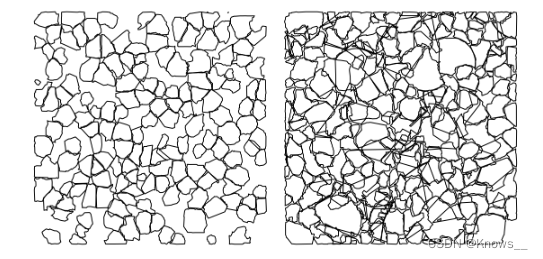




 文章介绍了lidR包在处理机载激光扫描数据(ALS)时的功能,包括读取、验证、绘图、地面分类、数字地形模型(DTM)和冠层高度模型(CHM)生成、单树分割等。重点讨论了不同算法如渐进形态滤波器、布料模拟和多尺度曲率分类在地面分类中的应用,以及点云归一化方法。此外,还涉及了点云数据的可视化和单个树木的检测与分割策略。
文章介绍了lidR包在处理机载激光扫描数据(ALS)时的功能,包括读取、验证、绘图、地面分类、数字地形模型(DTM)和冠层高度模型(CHM)生成、单树分割等。重点讨论了不同算法如渐进形态滤波器、布料模拟和多尺度曲率分类在地面分类中的应用,以及点云归一化方法。此外,还涉及了点云数据的可视化和单个树木的检测与分割策略。

















 509
509

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








