邻近标记技术(Proximity Labeling,PL)是一种用于研究蛋白质-蛋白质相互作用或蛋白质-近端分子网络的高通量技术。相较于传统的Co-IP、GST-pull down等技术,PL技术因其可以在自然条件下或细胞内进行,并能有效捕获瞬时或弱蛋白相互作用而备受关注。该技术的核心原理是通过基因工程或化学手段将目标蛋白与具有邻近标记功能的酶(一般为生物素连接酶或过氧化物酶)融合表达。在活细胞或特定条件下激活酶的活性,使其催化标记底物(如生物素)共价结合到邻近的内源蛋白或核酸分子上(标记半径为10-20 nm),最后通过亲和素磁珠富集带有标记的蛋白并进行质谱分析,从而实现对目标蛋白周围相互作用分子或邻近分子的捕获与鉴定。随PL技术的不断发展,科学家们开发出多种不同的PL酶。本次讨论聚焦于APEX2、TurboID、miniTurboID、AirID介导的PL技术,深入剖析其原理及优缺点,提供全方位的使用指南。
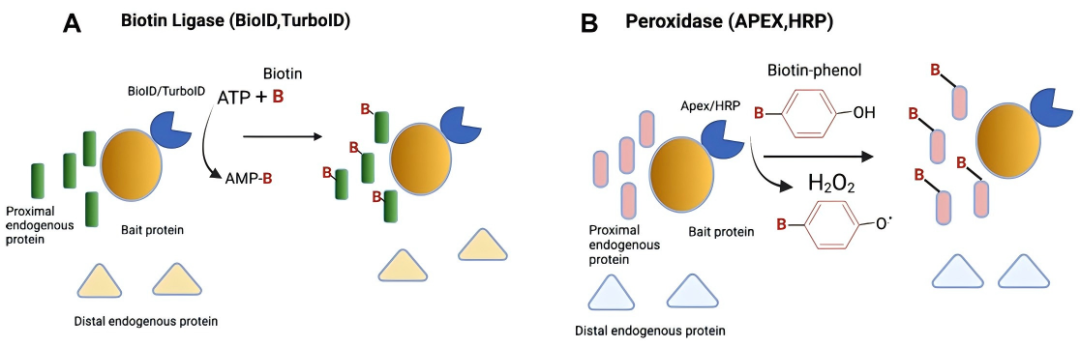
图1 邻近标记技术原理示意图(Boby et al., 2022)
01 工程化抗坏血酸过氧化物酶APEX2
1、原理:将诱饵蛋白与APEX2融合表达,APEX2经H2O2激活将底物中的生物素-苯酚(Biotin-phenol,BP)转化为生物素-苯氧基自由基,与邻近蛋白中富含电子的氨基酸残基(如酪氨酸、色氨酸、组氨酸和半胱氨酸)侧链共价结合,迅速标记邻近的蛋白质分子,最后通过亲和纯化技术和质谱分析以鉴定蛋白互作。
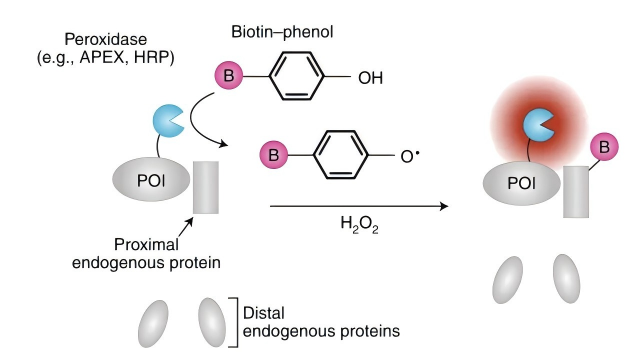
图2 APEX2邻近标记技术原理示意图(Qin W et al., 2021)。
2、优点:能够在极短的时间(<1min)内对邻近蛋白进行标记。
3、缺点:H2O2带有细胞毒性,需严格控制实验条件。
4、案例:G-四链体(G4)结构作为一种非典型核酸二级结构在调控基因表达、DNA复制和端粒维持等细胞生物学过程中起重要作用。为解决传统的实验技术在空间分辨率上不足的问题,研究人员通过设计基于APEX2的PL策略实现在活细胞亚细胞结构中捕获线粒体DNA(mtDNA)G4相互作用蛋白(G4BPs)。将APEX2酶与G4特异性纳米抗体(SG4)相融合,在H2O2激活条件下与邻近的G4BPs氨基酸共价结合。Western Blot结果有效评估了SG4-APEX2和mSG4-APEX2融合蛋白的表达情况。使用线粒体标志物TOMM70的免疫荧光共定位实验确认了融合蛋白定位于HEK-293T细胞线粒体中。
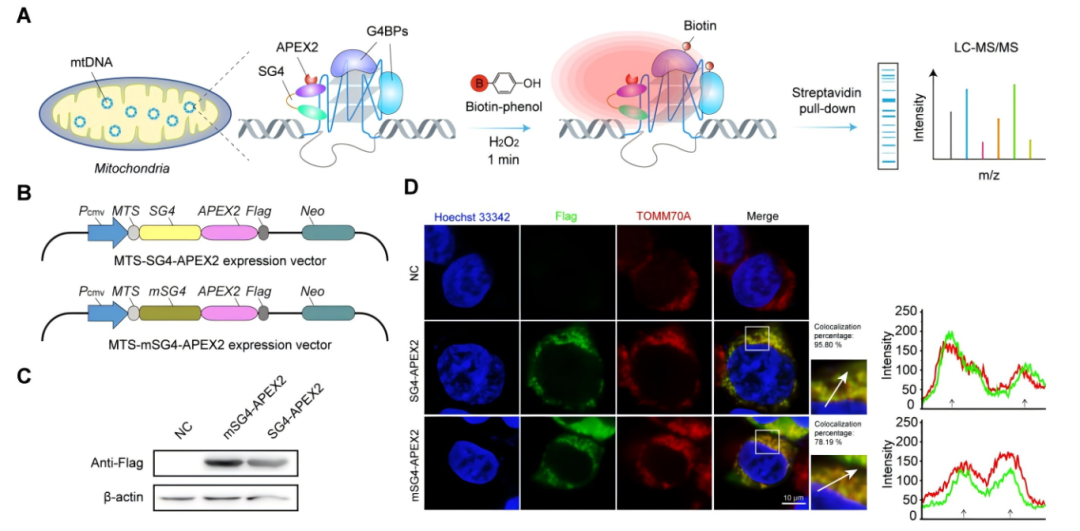
图3 APEX2邻近标记策略靶向线粒体DNA G4(Wang X et al., 2025)。
02 高效生物素连接酶TurboID
1、原理:TurboID是BioID的改进版本。诱饵蛋白与TurboID融合表达,TurboID利用ATP依赖活性,将外源生物素(Biotin)转化为高反应性的Biotin-AMP中间体,Biotin-AMP共价结合到邻近目标蛋白的赖氨酸上实现Biotin标记。进而通过链霉亲和素磁珠富集带有Biotin标记的蛋白,并进行质谱鉴定以揭示蛋白的互作网络。
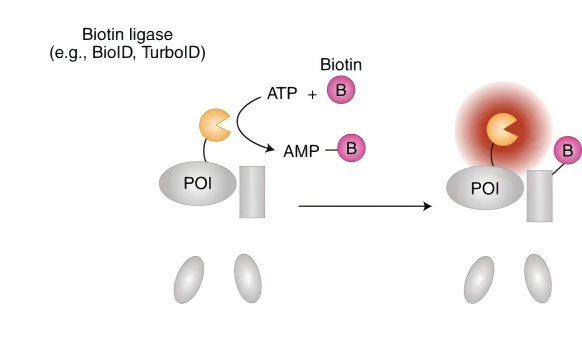
图4 TuroboID邻近标记技术原理示意图(Qin W et al., 2021.)。
2、优点:① 无细胞毒性。② 具有较高的催化效率,标记时间短(10min-1h)。③ TurboID的小分子量(大约16 kDa)使其更容易融入各种类型融合蛋白而不干扰其正常的功能。
3、缺点:标记范围可能由于非特异性标记导致假阳性结果。
4、案例:2023年,Wu Y等人通过TurboID邻近标记技术确定了肿瘤细胞中组蛋白乙酰转移酶KAT8通过与干扰调节因子1(IRF1)相互作用形成KAT8-IRF1复合体。在黑色素瘤细胞系A375细胞中表达KAT8-TurboID融合蛋白,加入生物素短暂孵育后,TurboID迅速标记KAT8的邻近蛋白,经质谱分析鉴定发现IRF1是KAT8的潜在互作蛋白,IP实验检测确定了二者的相互作用。该团队经后续实验检测发现KAT8-IRF1复合体通过相分离形成核内凝聚体,高效招募转录机器,显著上调免疫检查点蛋白PD-L1的表达,从而帮助肿瘤细胞逃脱免疫杀伤。
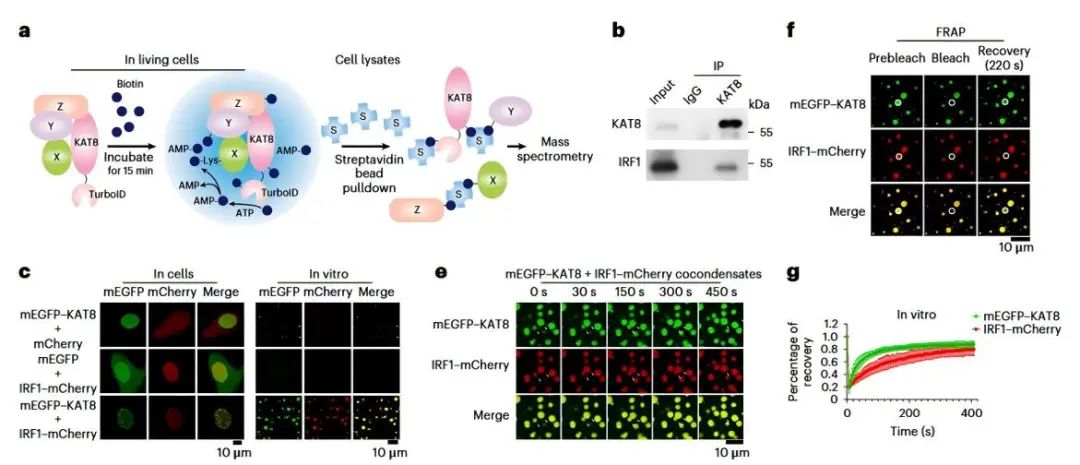
图5 TurboID邻近标记确定KAT8-IRF1复合体的相分离(Wu Y et al., 2023)。
03 高效生物素连接酶miniTurboID
1、原理:华人科学家Alice Ting团队定向改造开发出TurboID后,通过后续研究又进一步开发出TurboID的截断体miniTuboID,其删除了TurboID的非必须区域(N端的结构域)以缩小体积。其实验原理与TurboID基本一致,但同时也表现出其他的性能优势。
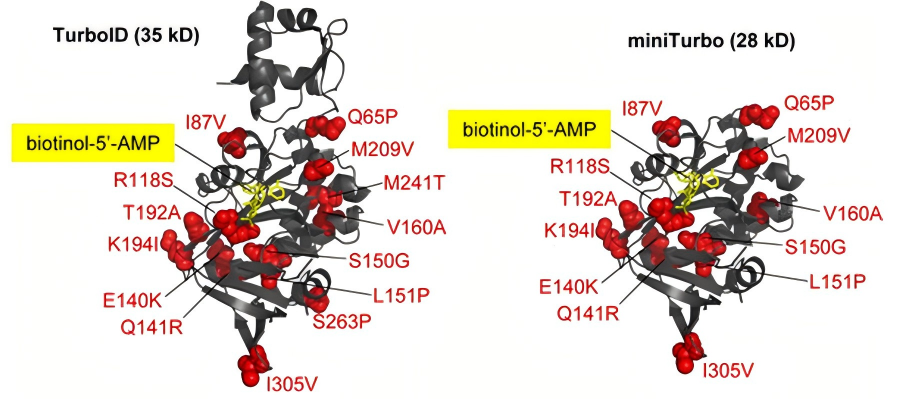
图6 TurboID与miniTurbo的工程化改造示意图(Branon T C et al., 2018)。
2、优点:① 平衡了标记速度与特异性,有效降低背景影响,适合长期标记和低丰度蛋白研究。② 可以解决大蛋白表达量难检测的问题。
3、缺点:催化效率低于TurboID。
4、案例:当线粒体受损(如膜电位去极化)时,细胞会通过选择性自噬途径清除这些线粒体以维持稳态。RAB7A是一种调控膜运输的GTP酶,有研究发现,线粒体去极化后,RAB7A会转位至线粒体表面,参与线粒体的自噬。该课题组将miniTurbo与RAB7A融合表达,构建3xFlag-miniTurbo-RAB7A融合蛋白表达载体,包括野生型RAB7A(WT)和突变体RAB7A-GTP(CA)、RAB7A-GDP(DN),Co-IP等实验验证结果表明C5orf51蛋白和DN突变体的相互作用增强,确定了C5orf51蛋白是RAB7A在线粒体自噬中的新调控因子。
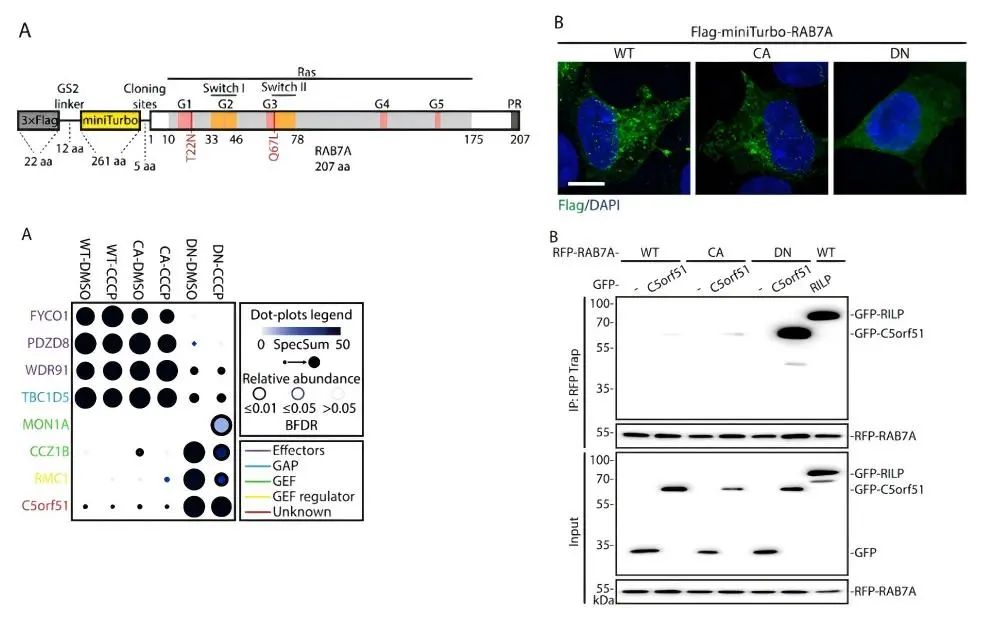
图7 miniTurbo技术研究线粒体自噬中的RAB7A蛋白互作网络(Yan B R, et al., 2022)。
04 新一代高效生物素连接酶AirID
1、原理:为解决TurboID在高浓度生物素条件下容易引起非特异性标记和细胞毒性的问题,2020年,Kido等人基于祖先酶重建算法设计,在TurboID基础上进一步优化开发出了新型生物素邻近标记酶AirID。
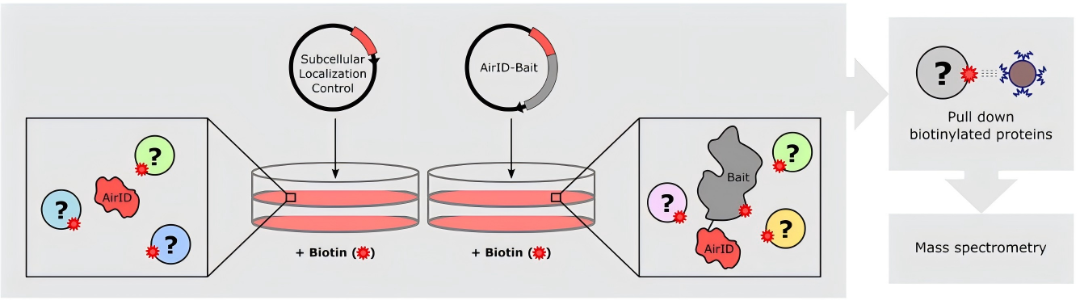
图8 AirID邻近标记技术原理示意图(Schaack G A et al., 2023.)。
2、优点:① 在生物素浓度较低情况下也能实验生物素标记,减少了非特异性标记,降低假阳性结果的影响。② 低细胞毒性,不影响细胞活性。③ 排除细胞自生物素化对实验结果的影响。
3、缺点:标记速度低于TurboID。
4、案例:研究人员通过AirID标记技术验证了小分子抑制剂Nutlin-3对p53-MDM2相互作用的抑制效果。已有研究表明,抑癌蛋白p53与E3-泛素连接酶MDM2的结合可以促进p53的降解,而小分子抑制剂Nutlin-3可以特异性阻断p53和MDM2的相互作用。免疫印迹结果显示,AirID的标记作用减弱表明Nutlin-3显著降低了FG-MDM2的生物素化水平,证明药物有效阻断了p53-MDM2互作。AlphaScreen技术定量蛋白互作水平结果也显示随Nutlin-3药物浓度的增加,p53-MDM2的互作信号也明显降低。
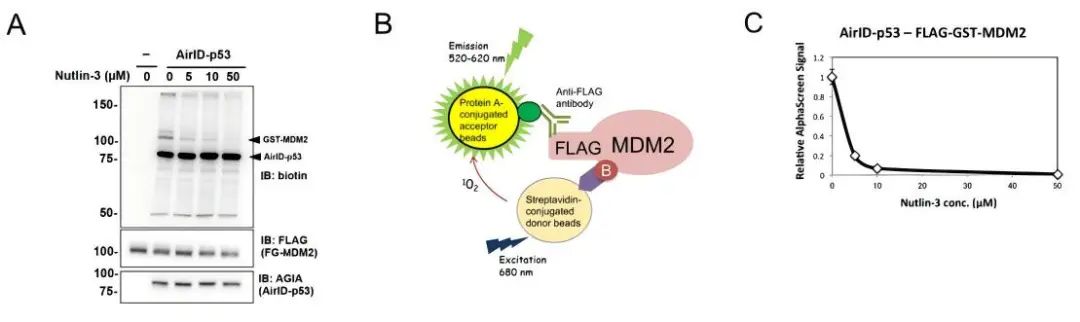
图9 AirID标记技术检测Nutlin-3对p53-MDM2相互作用的抑制(Kido K et al., 2020)。
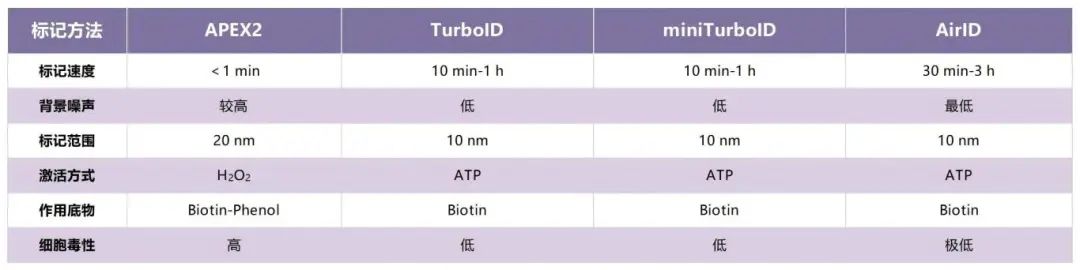
05 APEX2、TurboID、miniTurboID、AirID特性比对
参考文献
[1] Mathew B, Bathla S, Williams K R, et al. Deciphering spatial protein–protein interactions in brain using proximity labeling[J]. Molecular & Cellular Proteomics, 2022, 21(11).
[2] Qin W, Cho K F, Cavanagh P E, et al. Deciphering molecular interactions by proximity labeling[J]. Nature Methods, 2021, 18(2): 133-143.
[3] Wang X, Qin G, Yang J, et al. A subcellular selective APEX2-based proximity labeling used for identifying mitochondrial G-quadruplex DNA binding proteins[J]. Nucleic Acids Research, 2025, 53(1): gkae1259.
[4] Wu Y, Zhou L, Zou Y, et al. Disrupting the phase separation of KAT8-IRF1 diminishes PD-L1 expression and promotes antitumor immunity[J]. Nature Cancer, 2023, 4(3): 382-400.
[5] Branon T C, Bosch J A, Sanchez A D, et al. Efficient proximity labeling in living cells and organisms with TurboID[J]. Nature Biotechnology, 2018, 36(9): 880-887.
[6] Yan B R, Li T, Coyaud E, et al. C5orf51 is a component of the MON1-CCZ1 complex and controls RAB7A localization and stability during mitophagy[J]. Autophagy, 2022, 18(4): 829-840.
[7] Schaack G A, Sullivan O M, Mehle A. Identifying Protein‐Protein Interactions by Proximity Biotinylation with AirID and splitAirID[J]. Current protocols, 2023, 3(3): e702.
[8] Kido K, Yamanaka S, Nakano S, et al. AirID, a novel proximity biotinylation enzyme, for analysis of protein-protein interactions[J]. eLife, 2020, 9: e54983.
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








