一、基因组研究的盲区:被忽视的微蛋白世界
在过去的研究中科学家们普遍认为人类基因组中只有约2%的DNA序列编码蛋白质,其余部分被归类为非编码部分。近年来越来越多的研究表明传统认为的"非编码区域"实际上蕴藏着大量被忽视的小型开放阅读框(sORF)及其编码的微蛋白(microproteins)。传统基因组注释流程通常仅关注长度超过100个氨基酸的蛋白质,对于被认为非编码的RNA区域(如UTR区、lncRNA或circRNA上的区域)翻译产生的功能性微蛋白长度一般小于100个氨基酸,其对应的sORF长度一般不足300 nt。这些被忽视的微蛋白在生命活动中扮演着关键调控角色,它们参与RNA代谢、DNA修复、离子运输、转录调控等多种核心生物学过程,更与多种疾病状态密切相关,例如某些微蛋白能够促进或抑制肿瘤发生和发展,成为潜在的治疗靶点(Tong and Martinez., 2025)。面对这一隐藏的蛋白质世界,我们需要一种能够全面捕捉翻译活动的高通量技术。2009年,Ingolia及其团队开发的Ribo-seq技术应运而生,填补了这一技术空白,开启了微蛋白研究的新纪元。
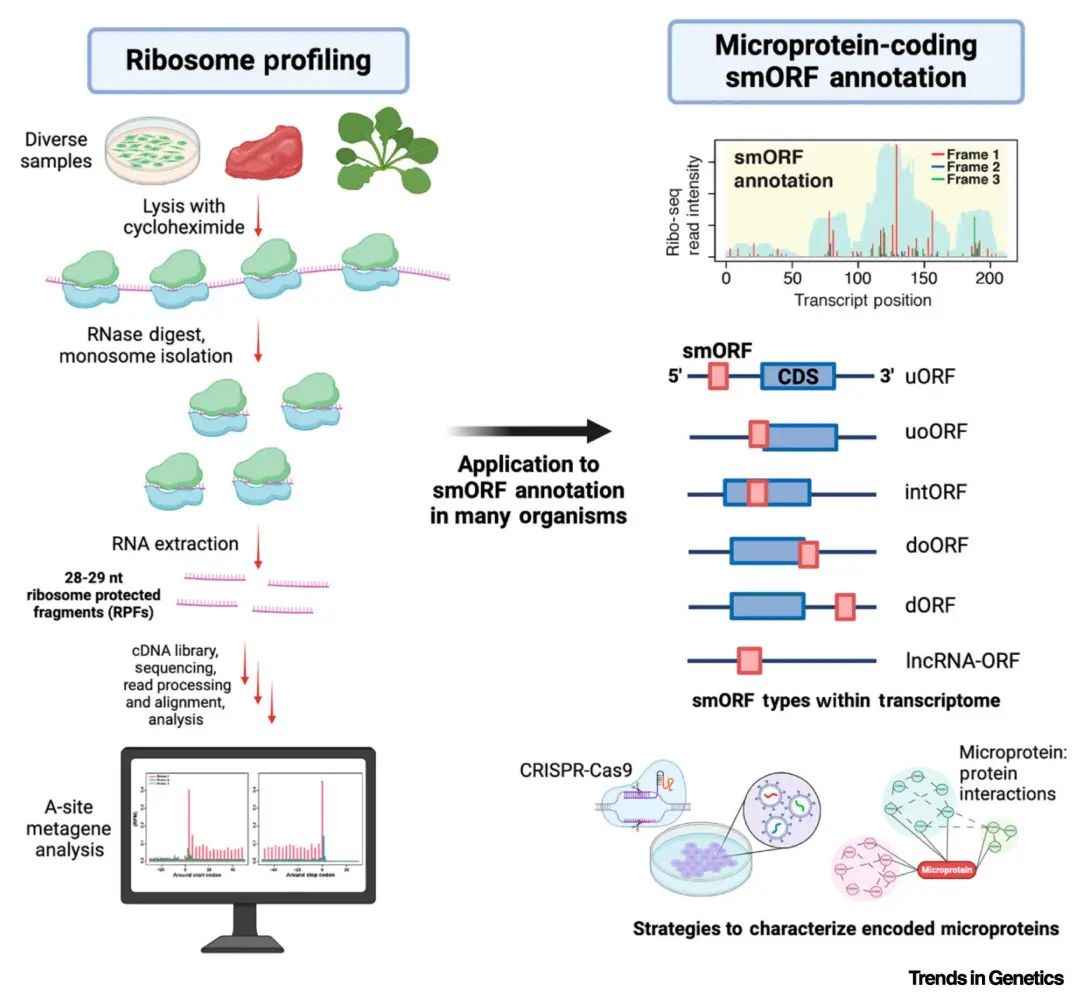
图1 Ribo-seq识别翻译smORFs(Tong and Martinez., 2025)。
二、Ribo-seq技术原理:解码翻译组的革命性工具
Ribo-seq的工作流程主要分为三个步骤。首先,在翻译抑制剂(如环己酰亚胺)存在的情况下裂解细胞,使延伸中的核糖体停滞,随后用RNase降解未受保护的RNA,分离单核糖体并提取核糖体保护的RNA 片段(Ribosome Protected Fragments,RPFs)。这些 RPFs 长度约为30 nt,是核糖体在翻译过程中“足迹”的体现。接着,将RPFs转化为DNA文库进行高通量测序。最后,运用生信分析将测序得到的读段映射回基因组,从而确定正在翻译的 开放阅读框(ORF)并测量其丰度。与传统的多核糖体分析相比,Ribo-seq的最大优势在于能够以单密码子的分辨率监测核糖体定位,精准地判断哪个ORF正在被翻译。活跃翻译的ORF在读段比对中会呈现出三核苷酸周期性,这一特征为生物信息学工具解读ORF翻译提供了关键依据。
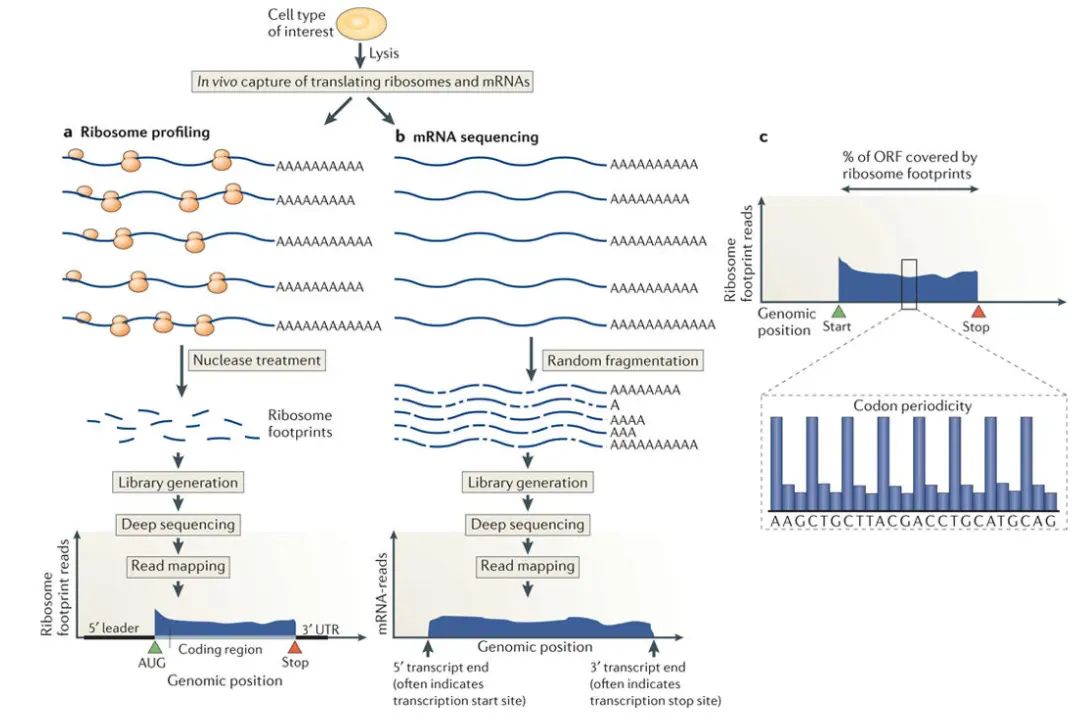
图2 核糖体足迹分析概述(Brar G A and Weissman J S., 2015)。
三、神经发育中sORF翻译的微蛋白案例

发表期刊:Nature neuroscience(IF=21.2)
研究背景:这项研究通过整合Ribo-seq和RNA-seq技术,系统解析了人类大脑发育过程中RNA翻译的动态调控机制,成功构建了人类大脑的翻译全景图谱,不仅精准定位了基因表达调控的关键时间节点,还新发现了数千个以往未被识别的翻译事件,为理解大脑发育的分子机制提供了全新视角。
研究材料:30个产前大脑皮质样本和43个成人大脑样本;人胚胎干细胞(hESC)衍生的神经元细胞。
研究内容:
RNA-seq揭示了大脑中mRNA的表达谱,而Ribo-seq则提供了活跃mRNA翻译的定量评估。研究团队鉴定出172,187个不同的活跃翻译开放阅读框,对应13,305个基因。这些ORFs不仅包括已知的蛋白质编码序列,还发现了大量非经典ORF(如sORFs、uORFs)。研究聚焦于鉴定和分析长度不超过100个氨基酸的sORFs及其编码的微蛋白。通过对8278个基因来源的38,187个活跃sORFs的系统分析,发现大量sORFs源自先前被注释为非编码的转录本,包括lncRNA、假基因和反义转录本等。与典型ORF相比,sORFs表现出显著更低的核糖体密度,这一低翻译特征与既往研究报道一致。值得注意的是,研究揭示了sORFs通过RNA丰度与翻译效率的协同动态调控参与发育过程,这种精细调控机制可能有助于大脑发育成熟过程中微蛋白水平的精确调节。为验证sORFs的翻译活性,研究团队整合了质谱检测与Ribo-seq数据,在蛋白质水平上独立证实了sORFs的翻译事件,从而显著提升了微蛋白检测的可靠性和灵敏度。Ribo-seq与蛋白质组学联用,是发现非经典翻译事件的强有力工具。
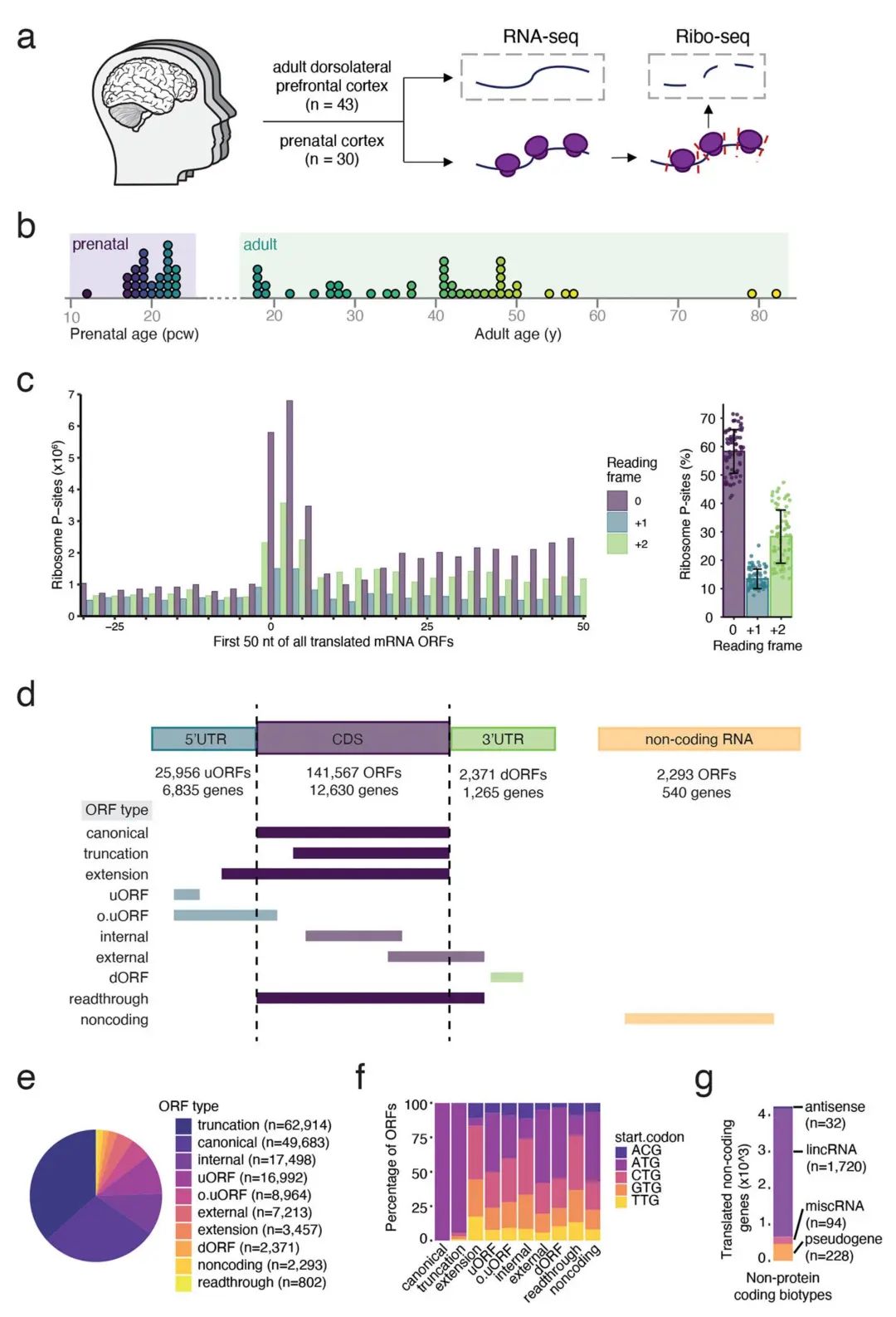
图3 Ribo-seq捕获成人和产前大脑中的活跃翻译。
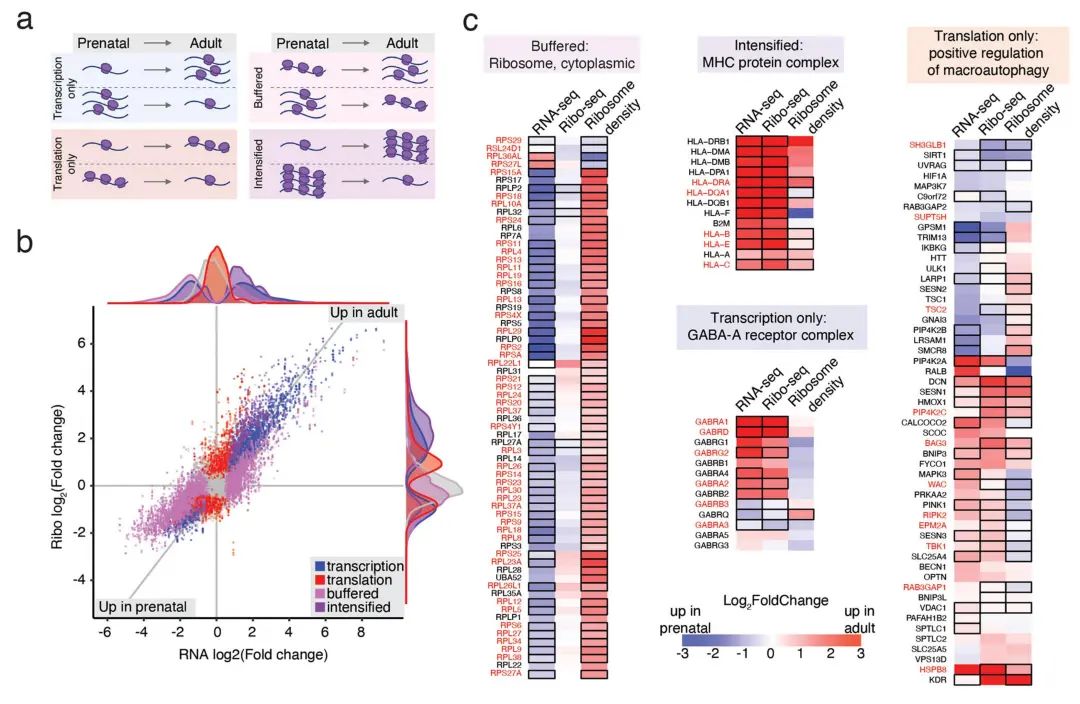
图4 人类大脑发育的转录和翻译调控。
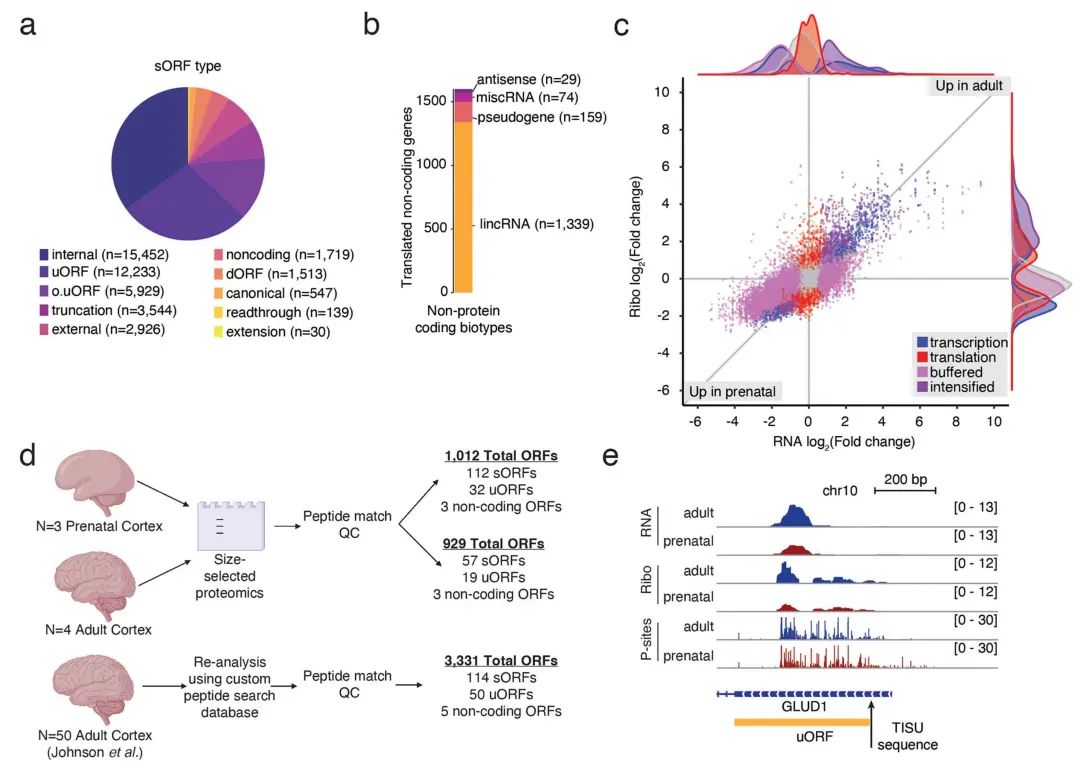
图5 大脑发育过程中的微蛋白表达和验证。
参考文献
[1] Brar G A, Weissman J S. Ribosome profiling reveals the what, when, where and how of protein synthesis[J]. Nature reviews Molecular cell biology, 2015, 16(11): 651-664.
[2] Tong G, Martinez T F. Ribosome profiling reveals hidden world of small proteins[J]. Trends in Genetics, 2025, 41(2): 101-103.
[3] Duffy E E, Finander B, Choi G H, et al. Developmental dynamics of RNA translation in the human brain[J]. Nature neuroscience, 2022, 25(10): 1353-1365.




















 473
473

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








