ftp.ebi.ac.uk 数据集介绍 表型数据 数据库 Harm_sumstats_status.db如何读取
要读取这个 SQLite 数据库文件(Harm_sumstats_status.db),可以按照以下步骤操作:
前提条件
- 先下载该文件:通过 FTP 工具(如 FileZilla)或命令行工具(如
wget或curl)从地址/ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/sumstats_harm_status/下载Harm_sumstats_status.db到本地。- 命令行下载示例(Linux/macOS):
wget ftp://ftp.ebi.ac.uk/pub/databases/gwas/summary_statistics/sumstats_harm_status/Harm_sumstats_status.db
- 命令行下载示例(Linux/macOS):
读取方法
1. 使用 SQLite 命令行工具(最简单)
- 安装 SQLite 命令行工具(多数 Linux 系统预装,Windows/macOS 可从 SQLite 官网 下载)。
- 打开终端/命令提示符,进入文件所在目录,执行:
sqlite3 Harm_sumstats_status.db - 进入交互模式后,可执行 SQL 命令查询数据:
- 查看所有表:
.tables - 查看表结构:
PRAGMA table_info(表名); - 查询数据:
SELECT * FROM 表名 LIMIT 10;(示例:查询前10行) - 退出:
.exit
- 查看所有表:
2. 使用 Python 读取(适合编程处理)
- Python 内置
sqlite3模块,无需额外安装。 - 示例代码:
import sqlite3 # 连接数据库(文件路径替换为你的本地路径) conn = sqlite3.connect("Harm_sumstats_status.db") # 创建游标 cursor = conn.cursor() # 查看所有表 cursor.execute("SELECT name FROM sqlite_master WHERE type='table';") tables = cursor.fetchall() print("所有表:", tables) # 假设表名为 "harm_status",查询前10行 if tables: table_name = tables[0][0] # 取第一个表名 cursor.execute(f"SELECT * FROM {table_name} LIMIT 10;") rows = cursor.fetchall() print(f"\n{table_name} 表数据(前10行):") for row in rows: print(row) # 关闭连接 cursor.close() conn.close()
3. 使用可视化工具(适合非编程用户)
- 推荐工具:
- DB Browser for SQLite(免费开源,支持 Windows/macOS/Linux,下载地址):
- 打开软件 → 点击 “Open Database” → 选择下载的
.db文件。 - 在左侧导航栏查看表,右键表名选择 “Browse Data” 即可可视化查看数据。
- 打开软件 → 点击 “Open Database” → 选择下载的
- 其他工具:Navicat、DBeaver 等(支持 SQLite 的数据库客户端)。
- DB Browser for SQLite(免费开源,支持 Windows/macOS/Linux,下载地址):
注意事项
- 若文件较大,查询时建议使用
LIMIT限制返回行数,避免卡顿。 - 若不清楚表名或字段含义,可先通过
.schema命令(SQLite 命令行)查看表结构和注释。
通过以上方法,你可以方便地读取和处理该 SQLite 数据库文件中的数据。
如何把 里面的表 导出为 tsv,
要将 SQLite 数据库中的表导出为 TSV(制表符分隔值)文件,可以通过 SQLite 命令行工具 或 Python 脚本 实现,以下是具体方法:
方法1:使用 SQLite 命令行工具(高效直接)
SQLite 命令行支持通过 .mode 和 .output 命令设置输出格式和文件,步骤如下:
-
打开数据库:
在终端/命令提示符中进入文件所在目录,执行:sqlite3 Harm_sumstats_status.db -
查看表名:
先确认需要导出的表名(假设表名为harm_status,替换为实际表名):.tables # 列出所有表 -
导出为 TSV:
执行以下命令(替换表名和输出文件名.tsv):.mode tabs # 设置输出格式为制表符分隔(TSV) .output 输出文件名.tsv # 指定输出文件路径(如 harm_status.tsv) SELECT * FROM 表名; # 导出表中所有数据 .output stdout # 恢复输出到终端(可选) .exit # 退出- 示例(导出表
harm_status到harm_status.tsv):
- 示例(导出表
.mode tabs
.output harm_status.tsv
SELECT * FROM studies;
.output stdout
.exit
```
方法2:使用 Python 脚本(灵活可控)
如果需要对导出数据做额外处理(如筛选、重命名列),可用 Python 实现:
import sqlite3
import csv
# 数据库文件路径
db_path = "Harm_sumstats_status.db"
# 导出的TSV文件路径
tsv_path = "harm_status.tsv"
# 要导出的表名(替换为实际表名)
table_name = "harm_status"
# 连接数据库
conn = sqlite3.connect(db_path)
cursor = conn.cursor()
# 获取表的列名(用于TSV表头)
cursor.execute(f"PRAGMA table_info({table_name});")
columns = [col[1] for col in cursor.fetchall()] # 提取列名
# 读取表中所有数据
cursor.execute(f"SELECT * FROM {table_name};")
rows = cursor.fetchall()
# 写入TSV文件(用制表符分隔,编码设为utf-8)
with open(tsv_path, "w", newline="", encoding="utf-8") as f:
writer = csv.writer(f, delimiter="\t") # 制表符分隔
writer.writerow(columns) # 写入表头
writer.writerows(rows) # 写入数据
# 关闭连接
cursor.close()
conn.close()
print(f"表 {table_name} 已导出到 {tsv_path}")
关键说明
-
表名确认:
先用.tables(命令行)或SELECT name FROM sqlite_master WHERE type='table';(SQL)确认表名,避免写错。 -
大表处理:
若表数据量很大,命令行方法更高效;Python 可通过fetchmany()分批读取,避免内存占用过高:# 分批读取示例(每次1000行) batch_size = 1000 while True: batch = cursor.fetchmany(batch_size) if not batch: break writer.writerows(batch) -
TSV 格式:
TSV 以制表符\t分隔字段,换行符分隔行,适合存储结构化数据,可直接用 Excel、R、Python 等工具打开。
通过以上方法,即可将 SQLite 表快速导出为 TSV 文件。
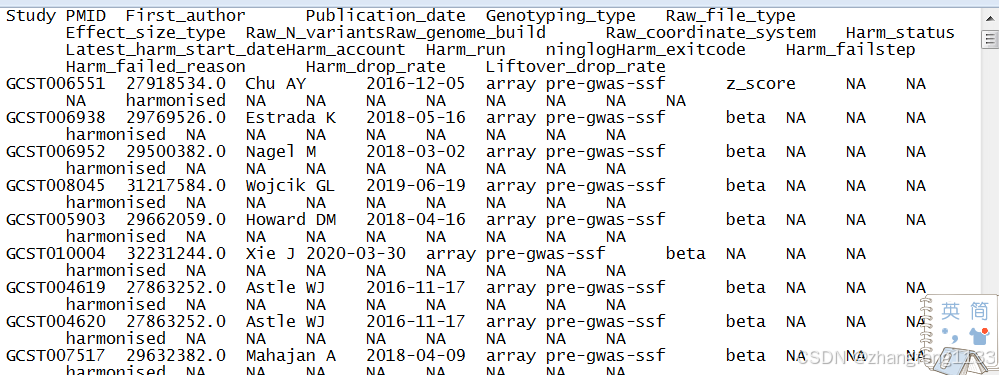
数据这样子的
Study PMID First_author Publication_date Genotyping_type Raw_file_type Effect_size_type Raw_N_variantsRaw_genome_build Raw_coordinate_system Harm_status Latest_harm_start_dateHarm_account Harm_run ninglogHarm_exitcode Harm_failstep Harm_failed_reason Harm_drop_rate Liftover_drop_rate
GCST90693191 NA NA NA NA gwas-ssf beta 13001605 GRCh38 1-based harmonised 2025-10-15 gwas_lsf yes NA NA NA 0.021 0.0
在这里插入图片描述





















 384
384

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








