文章目录
背景介绍:Affymetrix的探针(probe)一般是长为25碱基的寡聚核苷酸;探针总是以perfect match 和mismatch成对出现,其信号值称为PM和MM,成对的perfect match 和mismatch有一个共同的affyID。
CEL文件:信号值和定位信息。
CDF文件:探针对在芯片上的定位信息
affy包是R语言的bioconductor系列包的一个,就一个功能,读取affymetix的基因表达芯片数据-CEL格式数据,处理成表达矩阵!!!(转)
一、从GEO查询GSE65496,并下载GSE65496_family.soft和原始数据(.cel格式)
我直接进入NCBI官网->GEO Database -> 搜索GSE65496后下拉至页面底部进行下载
但是 不推荐!!!
建议直接安装GEOquery包进行下载
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("GEOquery")
library("GEOquery")
getGEOSuppFiles("GSE65496")
##解压
untar("GSE46106_RAW.tar")
二、仔细研读soft格式文件
#是对平台的描述
数据从ID那一行开始
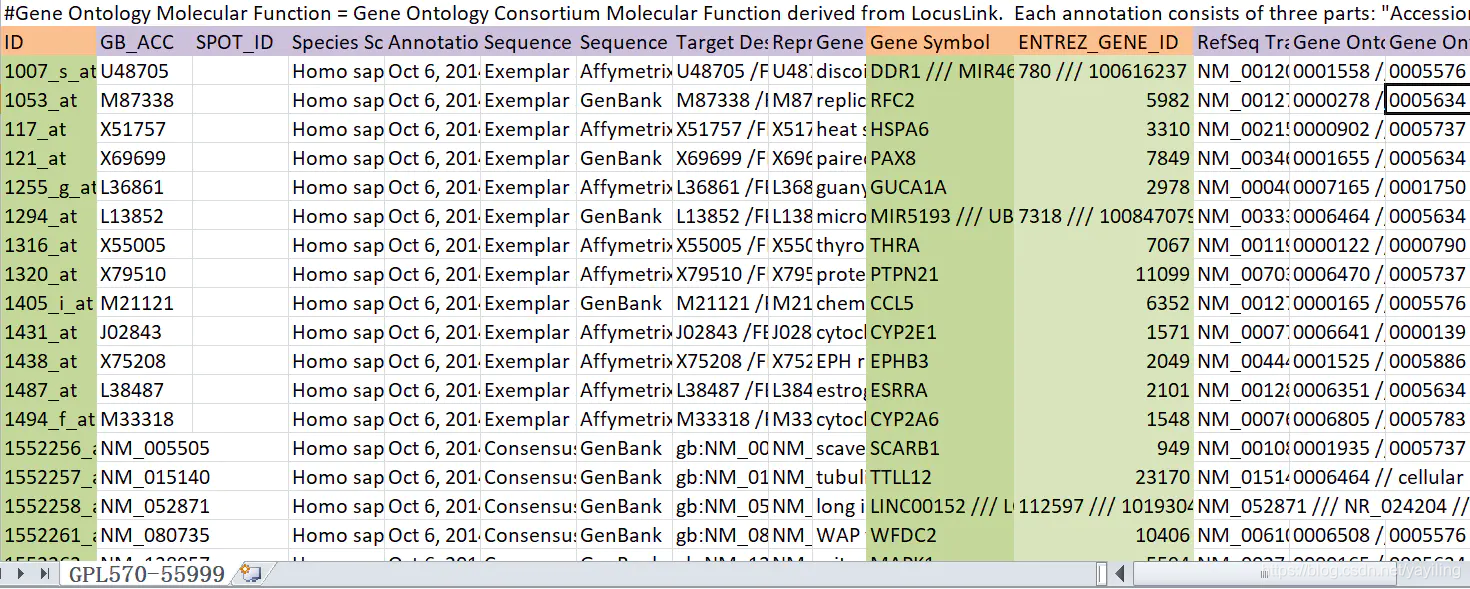
我们要用到标绿的那三列(ID,GeneSymbol,ENTREZ_GENE_ID)
三、用Bionconductor下的affy包下函数处理.cel文件,得到探针表达值
1.用Bioconductor中的相应包对表达谱数据做质量分析
首先,安装affy包,读取.cel文件
if (!requireNamesp




 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 6360
6360

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








