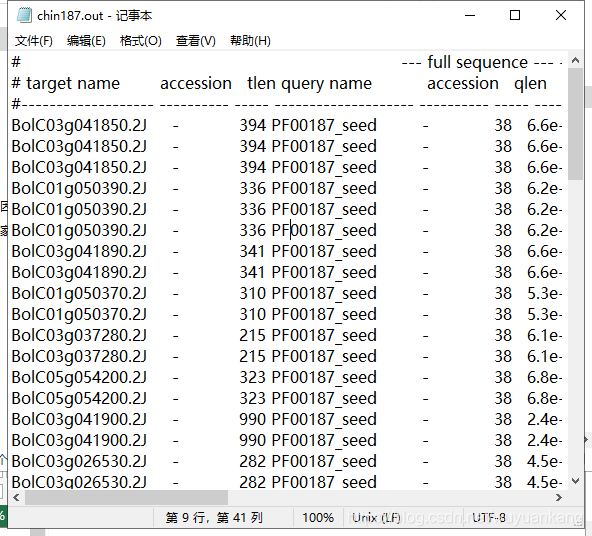
将linux下使用hmmsearch得到的out文件,用excel打开,得到其蛋白序列:
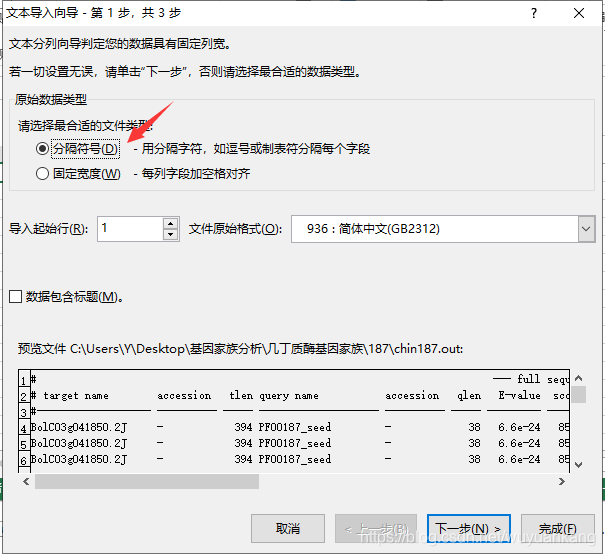
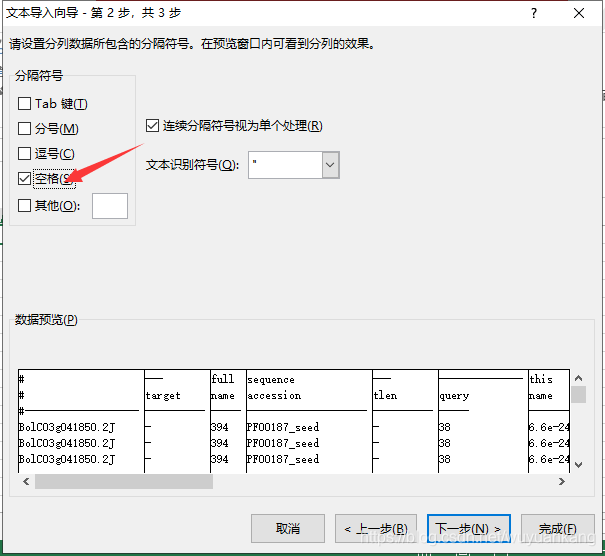
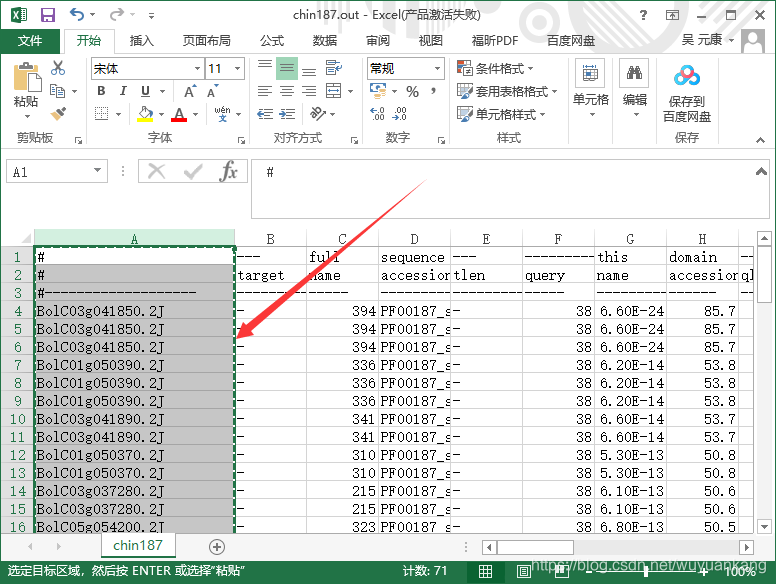
用TBTOOLS,根据序列ID,提取这些序列:
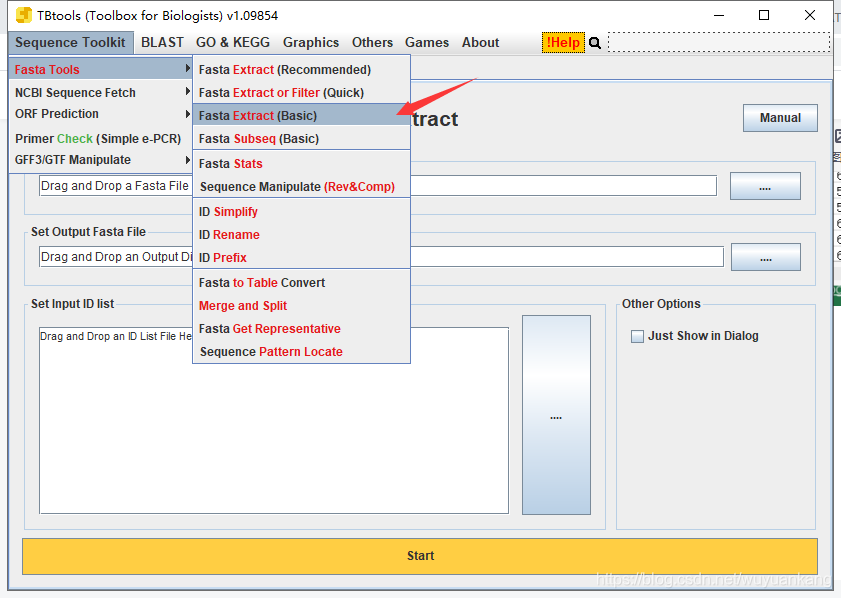
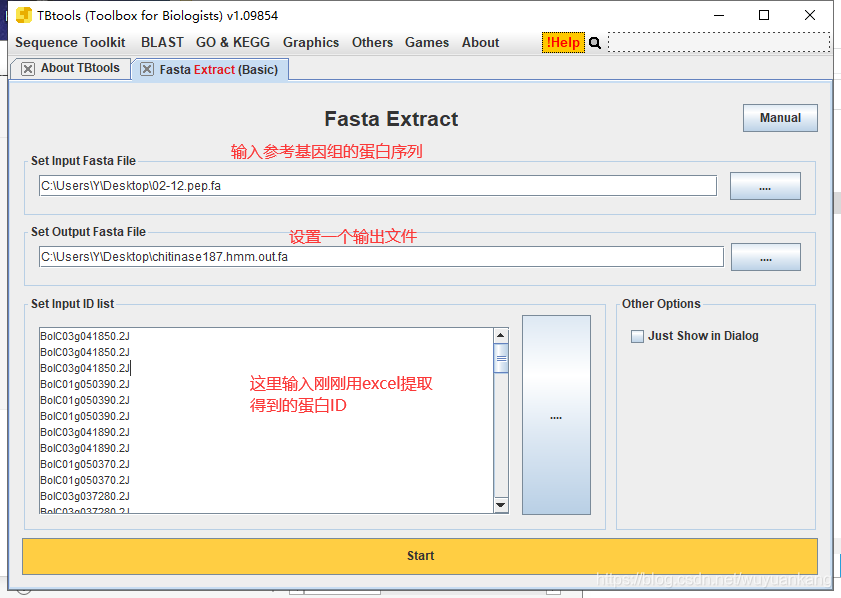
如此,我们便能初步得到目的基因家族的序列
接下来进行结构域分析
打开NCBI——CDD——batch—CD—search
输入刚刚提取得到的蛋白序列,点击Browse result,全选序列:
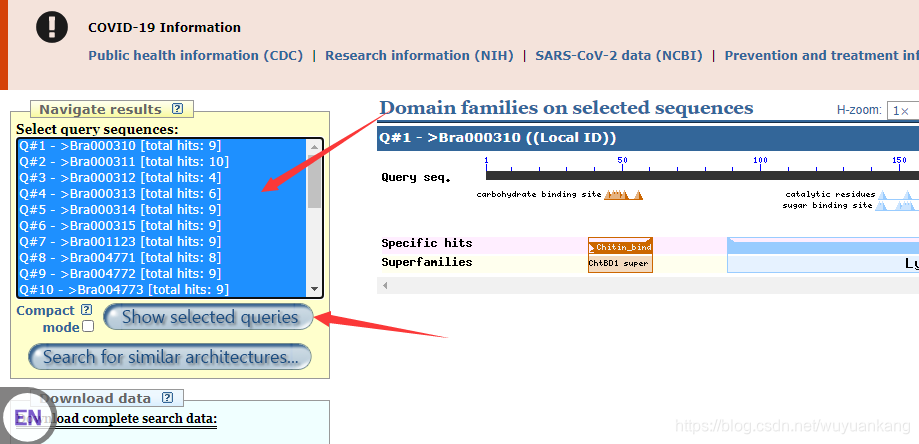
再点击show selected queries,查看蛋白结构域
将不含有目的基因家族结构域的蛋白删除,最后得到的蛋白序列即为该基因家族的序列。
 本文介绍了如何在Linux环境下利用hmmsearch获取蛋白序列,并通过TBTOOLS提取特定ID的序列。接着,通过NCBI的CDD批量CD搜索分析蛋白结构域,筛选出包含目的基因家族结构域的序列。最终,删除不符合条件的序列,获得目标基因家族的精确序列集合。
本文介绍了如何在Linux环境下利用hmmsearch获取蛋白序列,并通过TBTOOLS提取特定ID的序列。接着,通过NCBI的CDD批量CD搜索分析蛋白结构域,筛选出包含目的基因家族结构域的序列。最终,删除不符合条件的序列,获得目标基因家族的精确序列集合。
将linux下使用hmmsearch得到的out文件,用excel打开,得到其蛋白序列:




用TBTOOLS,根据序列ID,提取这些序列:


如此,我们便能初步得到目的基因家族的序列
接下来进行结构域分析
打开NCBI——CDD——batch—CD—search
输入刚刚提取得到的蛋白序列,点击Browse result,全选序列:

再点击show selected queries,查看蛋白结构域
将不含有目的基因家族结构域的蛋白删除,最后得到的蛋白序列即为该基因家族的序列。
 1万+
1万+