注:本篇博客不局限于深度学习层次化组织的项目结构,更倾向于一般的、稍微大型的python分析项目,无论项目是否涉及到深度学习等,也无论项目大小。
背景:每个项目之间都有一些共通的element在,
比如说代码脚本,可以是script中的.sh、.r或.py,或者是notebook中的.ipynb,
如果有实行步骤step严格需求,可以在脚本前后定义数字number,比如说01_xxxx.ipynb等;
在执行过程中,如果有配置文件需求,可以有config文件夹;还可以有相应的log输出。
如果是更一般的脚本,比如说是在各种数据分析中都通用的模块,相比于前面特化的script/notebook文件夹,我们还可以设置utils文件夹,用于在后续文件中导入各种模块。
又比如说数据,data中的各种层次数据,有自己的raw data,有公共测试的test data,还有各种中间处理获取的feature等数据。
总之,一个项目,尽管处于研究目的有不同的文件整合以及运行方式,
但在架构组织上,还是能够找到一点共通之处,因此采用规范的项目结构,是一个程序员在执行项目分析时的基本功,我认为也是一个项目开始之前最应该花功夫打基础的部分,只有项目结构组织好了,才方便后期的各种维护、升级,也才便于项目打包、编译、在社区共享。
原则上,良好的项目架构通常包含以下几个基本要素:
- 可读性:代码和目录结构清晰明了,便于理解.
- 可维护性:逻辑清楚,便于调试和更新.
- 可扩展性:方便新功能的添加,能轻松适应项目的变化.
一,基本项目结构
下面是我分析常用的文件夹结构组织,偏向于机器学习应用项目,仅代表个人意见
my_project/ # 项目根目录,最好就是最后的github项目名
├── data/ # 数据管理(版本控制用.gitignore或DVC)
│ ├── raw/ # 原始数据(禁止修改),如果数据清洗并非主要数据处理步骤,可以在此处放置部分清洗数据文件
│ ├── processed/ # 清洗后的结构化数据,比如说是特征化之后的数据等
│ └── external/ # 第三方数据集,第3方公共数据库、文献中的的数据等
│
├── notebooks/ # Jupyter实验笔记,实验性探索,个人建议一般情况下以notebook作为项目主线运行的逻辑日志
│ │ # 意思就是将notebook作为运行日志来记录整个项目的运行逻辑,重点在于日志记录,方便即时复盘自己哪一步做了什么
│ │ # 所以对于notebook文件要标注好step,也就是01_、02等数字运行前缀
│ │
│ ├── 01-eda.ipynb # 探索性分析,每一步最好都有对应的目的/目标任务,方便给notebook命名
│ ├── 02-feature-prototype.ipynb
│ └── 03-model-experiment.ipynb
│
├── src/ # 核心工程化代码
│ ├── data/ # 数据层
│ │ ├── loaders.py # 数据加载
│ │ └── preprocess.py # 数据清洗
│ │
│ ├── features/ # 特征层
│ │ ├── transformers.py # 特征转换
│ │ └── selectors.py # 特征选择
│ │
│ ├── models/ # 模型层
│ │ ├── train.py # 训练逻辑
│ │ ├── predict.py # 推理逻辑
│ │ └── architectures/ # 模型架构(如Mamba/Transformer等)
│ │
│ ├── evaluation/ # 评估层
│ │ └── metrics.py # 自定义指标
│ │
│ └── utils/ # 共享工具,一般是数据分析中常用的模块化代码,可以跨项目复用,看个人项目代码积累
│ ├── __init__.py # 将该文件夹转换为包(package),方便导入各种模块(module)
│ ├── logging.py # 日志配置
│ └── file_io.py # 文件操作
│
├── scripts/ # 执行入口,放置临时脚本文件,最后可以统一整理到src/utils/中
│ ├── train_model.py # 训练管道
│ ├── predict.py # 批量预测
│ └── deploy_api.py # 模型部署
│
├── models/ # 模型存储
│ ├── experimental/ # 实验模型
│ └── production/ # 生产模型
│
├── config/ # 配置管理:所有路径和超参数集中管理,避免硬编码
│ ├── params.yaml # 超参数
│ └── paths.yaml # 路径配置
│
├── tests/ # 单元测试
│ ├── test_data.py
│ └── test_models.py
│
├── reports # 输出成果,可以命名为reports,或者是results
│ ├── figures/ # 可视化图表
│ ├── results/ # 输出中间数据表格等,一般是中间分析过程中用于figures可视化的前置文件
│ ├── logs/ # 日志文件夹,主要用于存放notebook所使用脚本运行时的监控日志文件
│ └── final_report.md # 分析报告
│
├── requirements.txt # Python依赖
├── environment.yml # Conda环境
├── setup.py # 安装、部署、打包的脚本
├── Dockerfile # 容器配置(可选)
└── README.md # 项目文档
二,基本介绍:
主要是对照一,一一介绍我这么组织的基本逻辑,还是那句话,仅代表我个人项目组织的习惯,仅供参考。
1, 数据管理 (data/)
raw/:原始数据(CSV/JSON等),只读不修改,只要有了这些文件,后续可以再次执行业务流程逻辑;processed/:预处理后的数据(特征工程输出),或者是各种必要的中间输出数据,注意!是必要的中间数据,如果是不重要的临时数据,只需要在notebook中执行业务流程时说明一下如何获得如何处理即可,占据空间的无用数据要即时清理!external/:外部数据集(如公开数据集),在计算生物学背景中比较常见的,比如说是文献中的数据,或者是第三方公共数据库中的数据等;- 最佳实践:大文件用
.gitignore排除,使用DVC进行版本控制
2,实验开发 (notebooks/)
与其叫做实验开发,其实我更倾向于称呼其为项目执行日志,即Proj_log。
我这么组织的原委很简单,
我们的业务执行逻辑,也就是主力脚本其实是在scripts/以及src/中,
所以notebook其实本质上是记录我们调用这些主力脚本的日志,也就是一个记录,只不过我们脚本有很多,一步一步调用需要注意逻辑,所以我们需要在日志中表明我们先怎么做调用了脚本A,得到了中间数据B,我们想看一下特征C,所以再怎么做执行了脚本D,得到了E,然后有了灵感F,再怎么怎么规划脚本G,一步一步记录清楚。
- 按数字编号的Jupyter笔记(01-xxx, 02-xxx)
- 包含EDA、特征实验、模型原型等,总之就是一个任务一个notebook,尽量不要1个notebook什么业务探索逻辑都写,那样维护起来也麻烦,尽量一个notebook大小控制在10M以内,因为有些图是探索性的,不一定都要保存下来,但是图片、表格等中间输出都很占空间,一般在10~20M左右,太大了notebook打开都麻烦,而且后续稍微改动一下,光是save差一点的电脑都要vscode转圈半天,太浪费时间了。
- 注意:最终逻辑需迁移到
src/中,也就是探索过程中想到的脚本、要写的脚本,最终都要按照逻辑迁移到src/文件夹中(我一般是临时脚本都先写在scripts/中,后续第1轮小复盘的时候,再放到src/中)
其实对于scripts/和src/,我的意见是这样的:
初学者在构建脚本的时候,并不一定能够按照逻辑流程,严格区分哪一个逻辑步骤step中要写什么对应的脚本,所以一般分析脚本会写得很多、很杂,有些也会很长。
我一般是快速上手,在notebooksi阶段有灵感或者是有需求了,先临时写一个脚本,开始写的时候就放在scripts/中,然后后面边写边想、或者是在后续复盘的时候,我就基本上对这个脚本有了一个清晰的认知了,知道它是放在项目的哪一个逻辑步骤中,即对应于notebook中的哪一个,或者是用于整体model分析的哪一块内容中,总之这个时候我对于这个脚本有了清晰的认知,我就会重新整理,将这个脚本的代码逻辑移植到src/中;
3,核心代码 (src/)
- 模块化组织(比平面脚本更易维护):
data/:数据加载/清洗features/:特征转换器models/:模型训练/预测流程visualization/:定制化绘图- 我上面这么列,只不过是为了举例说明,核心代码这一块的组织是按照模块化来组织,其实可以隐约对应notebook,与其说是对应,其实应该是类比,也就是说任务逻辑如果分成很多个下游子任务,那么src/也分成几个模块化来管理核心脚本。
- 关键原则:每个文件<1000行,函数单一职责;我一般写一个单一的模块py,就算涉及到各种class类的处理,也一般确保一个代码尽量不要超过1000行,最好是在几百行之内,很少一个业务逻辑细分下来还要写那么多的,除非是算法,实在逻辑衔接上不好分离。
重点在于其中的utils/,主要是放置一些可以复用的关键函数模块,
比如说是机器学习中的常见降维、聚类函数模块,或者是一些常见的数据处理经典算法的实现等,在script/中的实现最后可以归类、整理到这里。
至于__init__.py文件,这个也不赘述了,可以参考我前面的博客https://blog.youkuaiyun.com/weixin_62528784/article/details/149749704?spm=1001.2014.3001.5502
4,配置管理 (config/)
所有路径和超参数集中管理的地方,避免硬编码。
我一般是使用YAML来管理超参数:

用的多的,主要就是pyyaml库
pip install pyyaml
# 然后后续导入,只需要
import yaml
YAML 库的用法
(1)解析 YAML 文件
将 YAML 文件或字符串解析为 Python 对象:
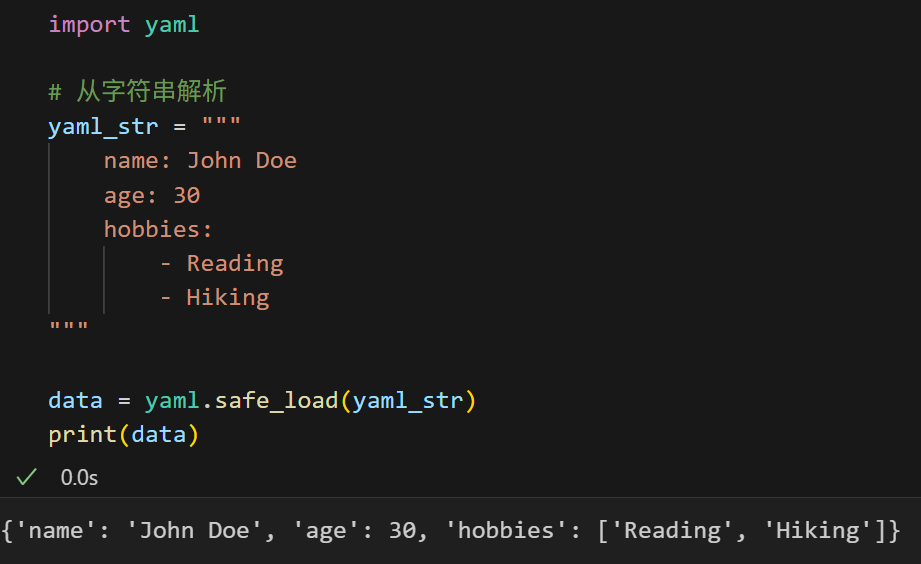
import yaml
# 从字符串解析
yaml_str = """
name: John Doe
age: 30
hobbies:
- Reading
- Hiking
"""
data = yaml.safe_load(yaml_str)
print(data)
当然,用的最多的就是从配置文件中解析:
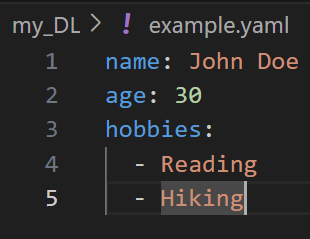
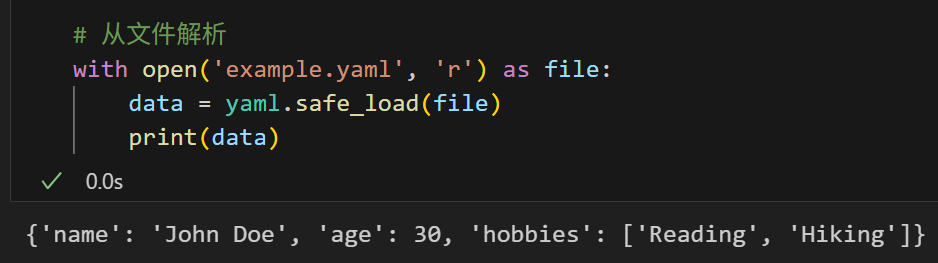
# 从文件解析
with open('example.yaml', 'r') as file:
data = yaml.safe_load(file)
print(data)
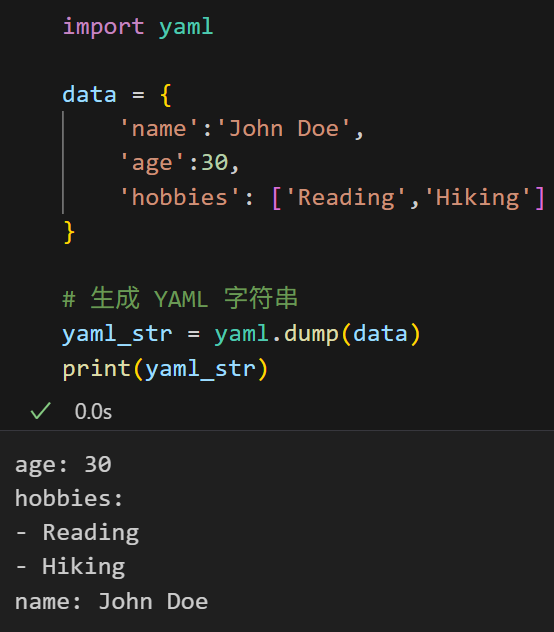
(2)生成 YAML 文件
将 Python 对象序列化为 YAML 格式的字符串或文件:
import yaml
data = {
'name':'John Doe',
'age':30,
'hobbies': ['Reading','Hiking']
}
# 生成 YAML 字符串
yaml_str = yaml.dump(data)
print(yaml_str)
是不是很眼熟,dump和load类,
参考我之前的博客:https://blog.youkuaiyun.com/weixin_62528784/article/details/148962157?spm=1001.2014.3001.5502
类比json和pkl格式。
5,模型存储 (models/)
- 按格式保存训练好的模型:
- Scikit-learn:
.pkl/.joblib - TensorFlow:
.h5/SavedModel - PyTorch:
.pt
- Scikit-learn:
- 命名包含实验信息,做好关键信息的备份溯源(如
model_acc85.pkl)
6,图表结果文件输出(reports/)
主要是项目运行结果(主要是一些可视化的文件,比如说是图表之类)的存储,
主要设置为figures/,或figs/文件夹存储;
以及results文件夹,存储一些中间输出的结果文件,用于后续制表等流程需要;
简单来说figs文件用于可视化输出,呈现中间(阶段性结果)或者是最后结果;
results/文件夹用于存储各种可用于可视化的中间文件或结果文件。
然后就是logs/文件夹,因为有很多脚本运行的时间比较长,需要挂在后台,就需要随时监控后台运行情况如何,也就是查看日志文件;
另外我本人其实习惯于在程序中随时打印一些提示信息、异常捕捉信息,这些信息对于程序运行以及debug都很重要,所以也需要log文件来最后复盘。
我见过很多学生,一开始写脚本的时候,对于日志文件的命名以及存放都很随意,基本上刚开始都习惯于在脚本执行文件夹中放置日志文件,比如说前面的scripts/文件夹,或者是notebook/文件夹。然后命名的时候也很随意,比如说随便使用数字,1.log之类,到了后面项目一大起来,要看的日志文件越来越多,到时候debug或者是复盘,都是很麻烦的,所以个人建议是一开始就养成一个好的习惯,这样在后续的复盘阶段都便于自己项目的修改。
至于日志文件的命名,除了使用运行脚本的项目命名便于查找之外,还可以使用一些时间相关的mark来进行标记,比如说是使用python中与时间相关的库,类似于date、datetime之类,可以实时获取时间,
建议命名习惯是脚本来源_目的或功能_时间,除了命名日志文件使用这种逻辑之外,其他的文件保存,比如说图片保存,或者是一些经常使用的、重复性的结果输出文件,都可以使用这种逻辑来保存,便于后续复盘时进行区分,以及便于查找。
然后就是一个pdf文件或者是md文件(或者是一系列pdf、md文件),对这个项目进行的一个复盘,
其实就是项目报告,或者是项目总结,就是记录整体的分析流程,以及一些图表以及可解读的结果文件等。
7,其余基本配置文件
- requirements.txt 或 enrionments.yml:包含项目依赖的包列表。
- README.md:包含项目的简要说明、安装指南、使用方法等。
- Dockerfile
- setup.py:安装、部署、打包的脚本
三,代码实现
参考:https://github.com/MaybeBio/bioinfor_script_modules/blob/main/32_Create_project_structure.py
import os
import argparse
def create_project_structure(project_name):
"""
Args:
project_name (str): 新项目的名称
Fun:
为新项目创建一个结构化的目录,注意该项目是建立在当前文件夹下,也就是pwd下
"""
# Define the directory structure
structure = {
'data': {
'raw': None,
'processed': None,
'external': None
},
'notebooks': None, # 改为空目录,不预设文件
'src': {
'data': None,
'features': None,
'models': None,
'evaluation': None,
'utils': {
'__init__.py': None
}
},
'scripts': None,
'models': None,
'config': None,
'tests': None,
'reports': {
'figures': None,
'results': None,
'logs': None,
'final_report.md': None
},
'requirements.txt': None,
'environment.yml': None,
'setup.py': None,
'Dockerfile': None,
'README.md': None
}
# 创建项目根目录
project_path = os.path.abspath(project_name)
os.makedirs(project_path, exist_ok=True)
print(f"Created project directory: {project_path}")
def create_structure(base_path, structure):
"""
Args:
base_path (str): 基础路径
structure (dict): 目录结构字典
Fun:
根据给定的结构在指定路径下创建目录和文件(递归创建目录结构)
"""
for name, content in structure.items():
path = os.path.join(base_path, name)
# 如果内容为None且名称不包含'.',则创建目录
if content is None:
if '.' in name:
# 包含点的视为文件
open(path, 'a').close()
print(f"Created file: {path}")
else:
# 不包含点的视为目录
os.makedirs(path, exist_ok=True)
print(f"Created directory: {path}")
else:
# content为字典,创建目录并递归
os.makedirs(path, exist_ok=True)
print(f"Created directory: {path}")
if isinstance(content, dict):
create_structure(path, content)
# 创建目录结构
create_structure(project_path, structure)
if __name__ == "__main__":
parser = argparse.ArgumentParser(description='Create a structured project directory.')
parser.add_argument('project_name', help='Name of the project directory to create')
args = parser.parse_args()
create_project_structure(args.project_name)
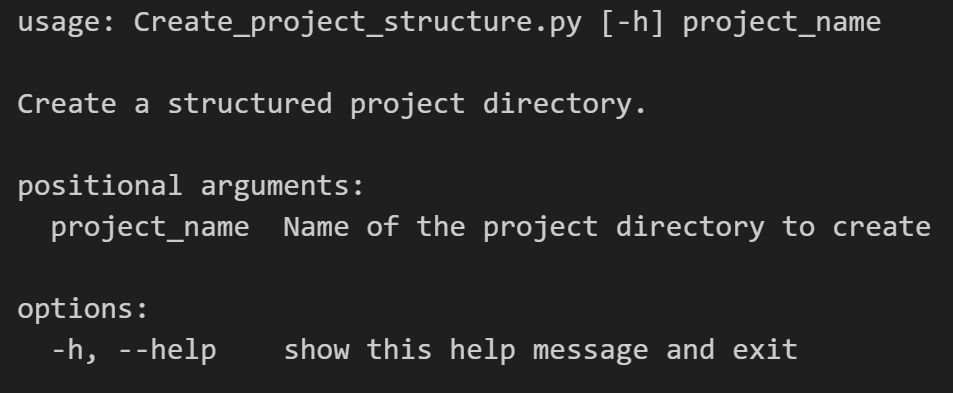
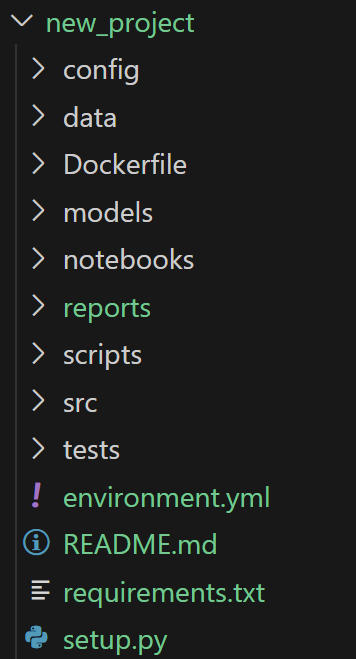
然后可以用于新建一个新项目,示例如下:
python Create_project_structure.py "new_project"
























 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








