这是GATK Best Practice系列学习文章中的一篇,本文尝试使用Gatk Germline spns-indels Pipeline来分析遗传病(耳聋)
数据
这次没有拿到遗传病的室间质评的数据,直接从NCBI上找一些数据来分析。NCBI上搜索deaf,点击第一条搜索结果,最后几经跳转找到数据下载页面:https://trace.ncbi.nlm.nih.gov/Traces/sra/?study=SRP218677
可以看到:Targeted next generation sequencing of 139 known deafness-related genes in 44 deaf patients with mono-allelic GJB2 mutations
这里一共44个样本,有兴趣的可以全部下载来分析。
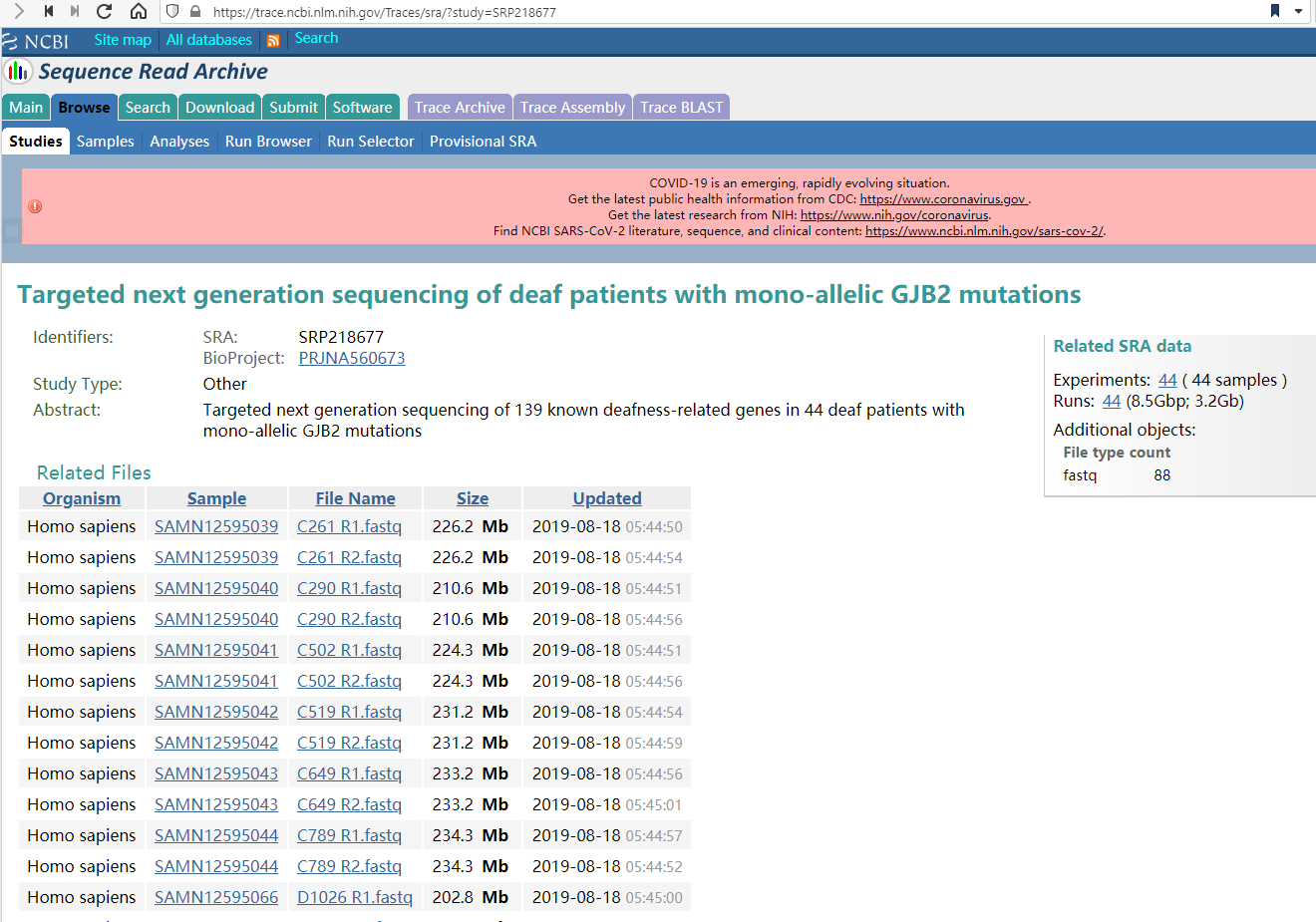
奇怪的是,这里不是sra下载,是直接下载fastq文件,不管了。使用下载工具,最后得到的文件是C261 R1.fastq.1这种文件名,这里首先改名为C261_R1.fastq,然后使用gzip,bgzip压缩得到 C261_R1.fastq.gz;C261_R2.fastq.gz也是同样.
也可以直接从一下链接下载:
C261_R1.fastq.gz bc72a0b02876273e2457c753a87241aa 54M
C261_R2.fastq.gz a5f6fcac16cbbe2b2a0c98982243a949 54M
概览:

用到的分析系统及分析流程文件
| 名称 (点击下载) | 备注 |
|---|---|








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 8278
8278

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








