生存分析(survival analysis)是生物医学研究中常用的分析方法。在队列随访研究中,我们会事先定义一些观察终点,比如肿瘤复发、患者死亡、血压达标等,这些终点称为事件(event)。从研究开始到发生事件的时间间隔称为生存时间(survival time),某些场景下也称为失效时间(failure time)。由于生存时间数据具有以下两个特点,所以提出生存分析这一特殊的分析方法。
一、文献解读
实操数据是从seer公共数据库提取的,包括1101名2004-2018诊断的老年恶性骨肿瘤患者的临床病理数据。暴露因素包括年龄、种族、性别、诊断年份、组织学类型、分级、分期、原发位置、TNM分期、肿瘤大小、是否化疗、是否放疗及手术方式。
这里绘制Kaplan-Meier(K-M)曲线的目的是为了比较不同手术方式下,患者的生存差异。
二、利用R语言绘制
1.安装加载R包并导入数据
绘制K-M曲线主要用到"survival"与"survminer"两个包,"readxl"包仅在导入xlsx形式数据集时用到。
install.packages("survival")install.packages("survminer")library(survival)library(survminer)library(readxl)raw<- read_excel("seer.xlsx")
2.绘制K-M曲线
在survival包中使用surv()函数创建生存对象,生存对象是将事件时间和删失信息合并在一起的数据结构,survfit()函数用来拟合生存曲线。
ggsurvplot()函数用于绘制K-M曲线,详细代码解读在下方进行了标注。
fit_Surgery <- survfit(Surv(months,status) ~ Surgery,data = raw)ggsurvplot(fit_Surgery,data = raw,pval.method=T,#添加计算p值的统计方法pval = TRUE,#添加P值risk.table = TRUE,#添加风险表xlab = "Follow up time(Months)",legend = c(0.8,0.15), #指定风险表图例位置break.x.by = 12) #设置X轴刻度间距
R语言绘制的曲线如下图所示: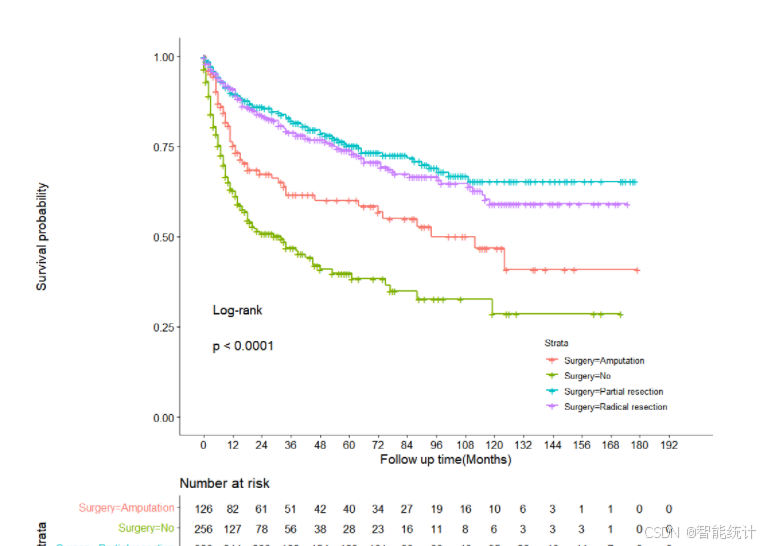
























 829
829

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








