Hello~今天小 M 要和大家聊一聊被称为“隐形杀手”的血脂异常,虽无声无息,却能在日积月累中侵蚀血管健康!我们一起来看看血管异常,都有哪些干预靶点吧~
血脂异常:
一种代谢紊乱,表现为血液中的脂质 (如甘油三酯、胆固醇、磷脂) 或脂蛋白含量异常升高或降低,与多种疾病相关,尤其是低密度脂蛋白胆固醇 (LDL-C) 升高被认为是动脉粥样硬化性心血管疾病关键的危险因素。
血脂异常的一线治疗是抑制 HMG-CoA 还原酶的他汀类药物,如洛伐他汀(Lovastatin) 和辛伐他汀 (Simvastatin)[1]。其次,胆汁酸螯合剂可减少胆汁酸的重吸收,从而增加低密度脂蛋白 (LDL) 的清除率,如考来维仑 (colesevelam)和考来烯胺(cholestyramine)。此外,纤维酸衍生物,如非诺贝特 (Fenofibrate) 是过氧化物酶体增殖物激活受体 (PPAR) 的激动剂,能增加高密度脂蛋白 (HDL) 并降低甘油三酯, LDL 和极低密度脂蛋白 (very low density lipoprotein, VLDL) 水平。尽管通过他汀类药物治疗可以很好地控制 LDL-C 水平,但是仍有高达 40% 的患者被心血管疾病困扰[2]。
近年研究显示前蛋白转化酶枯草杆菌蛋白酶/kexin 9型 (PCSK9)、血管生成素样蛋白 3 (ANGPTL3)、载脂蛋白 C3 (APOC3)、和 Lp (a) 等在降脂方面表现出良好的效果,是降脂治疗领域的新兴靶点[2][3]。
01
PCSK9 抑制剂
PCSK9 是脂质代谢中的关键酶,也是高胆固醇血症及其相关心血管疾病 (CVD)的治疗靶点。PCSK9 充当导航器,引导特定的表面蛋白受体(如 LDLR)在细胞质内体和/或溶酶体中降解。
低密度脂蛋白受体 (LDLR) 在控制血液中 LDL 胆固醇 (LDL-C) 的水平方面至关重要。通常情况下,LDLR 与 LDL-C 结合,转运和清除循环中的多余 LDL-C (图 1a)。
当 PCSK9 从肝细胞分泌并与细胞表面的 LDLR 结合时,LDLR 循环到细胞表面受到阻碍。由于 PCSK9 引起 LDLR 构象变化,LDLR 无法离开内体循环回到细胞表面。相反,PCSK9-LDLR-LDL-C 复合物会运输到溶酶体进行降解。PCSK9 通过促进 LDLR 降解,降低细胞表面的 LDLR 水平,从而增加血清 LDL-C (图 1b)。
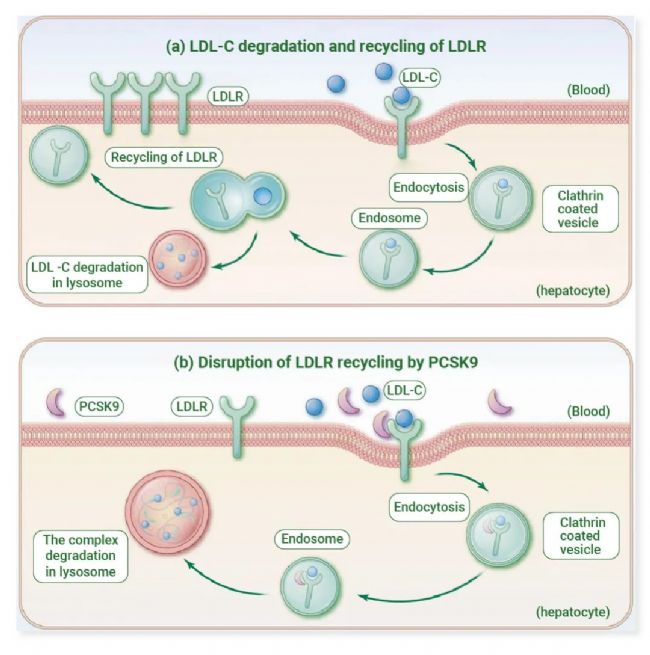
图 1. PCSK9 和 LDLR 在肝细胞中的生物学特性[4]。
目前,已经有四种 PCSK9 抑制剂获批用于临床:单克隆抗体 (Evolocumab, Alirocumab, Tafolecimab)和siRNA (Inclisiran)。其中临床研究已证实 Inclisiran 能降低 LDL-C 达 50%,可持续抑制 LDL-C 至少 6 个月,从而实现年两次的皮下注射,可以大大改善患者的依从性[5]。
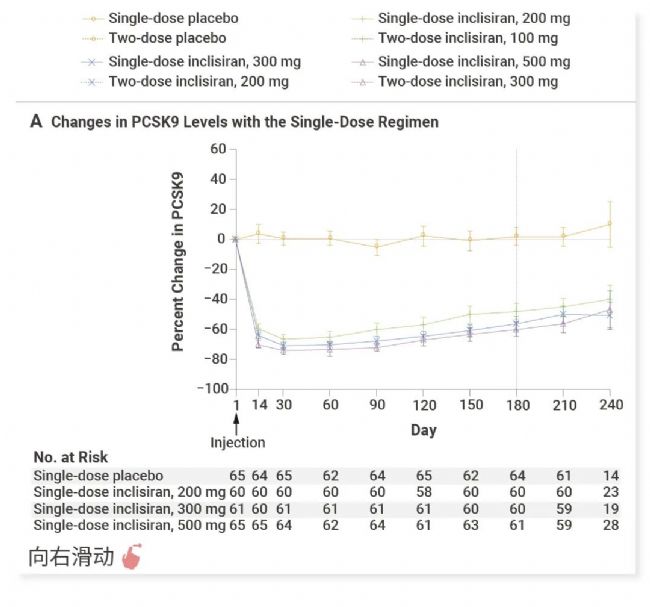
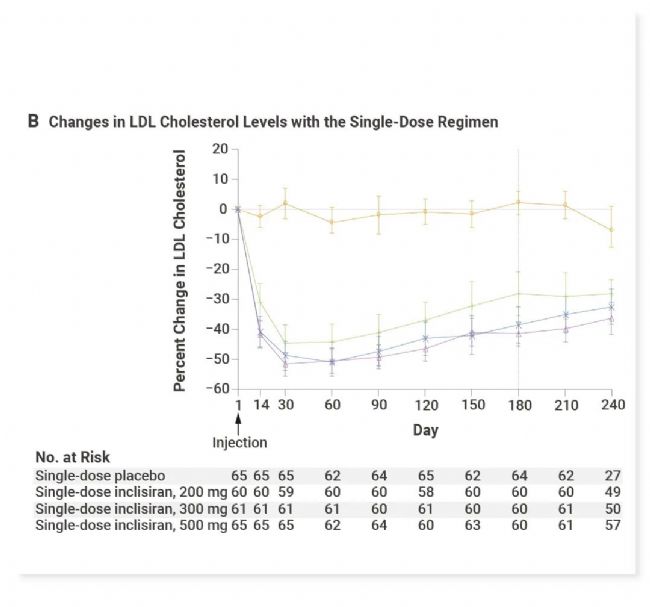

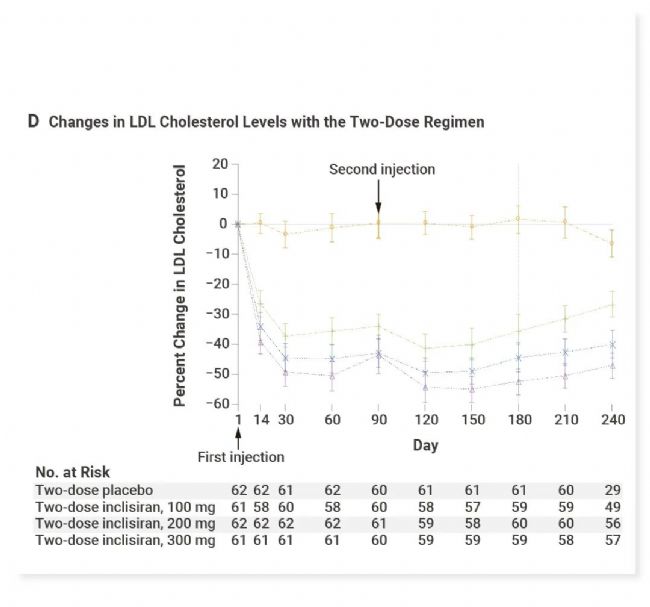
图 2. Inclisiran 对 PCSK9 和 LDL 胆固醇水平的影响[5]。
02
Lipoprotein (a) 抑制剂
脂蛋白 (a) (Lipoprotein (a), Lp(a)) 是一种与载脂蛋白 (a) 结合的低密度脂蛋白样颗粒,通过二硫键与载脂蛋白 (a) 共价结合 (图 3)。Lp (a) 被认为是动脉粥样硬化性心血管疾病的风险因素,高水平的 Lp (a) 会增加心血管风险。

图 3. Lp (a) 结构[8]。
Lp (a) 通过分子孔进入动脉内膜并被氧化,最终导致活性氧的形成,诱导炎症发生。而被氧化的 LDL 核心部分被巨噬细胞大量摄取,产生泡沫细胞,促进动脉粥样硬化斑块形成。Lp (a) 是唯一一种可以运输氧化型磷脂的脂蛋白,当 Lp (a)浓度高时,氧化型磷脂被递送至受损的血管,并引起内皮功能障碍、脂质蓄积、钙化和炎症。
目前临床上还没有药物能显著降低 Lp (a) 水平,PCSK9 抑制剂仅可将 Lp (a) 从基线降低约 15% 至 25%[6],而他汀类药物不能降低反而会轻度升高 Lp (a) 水平。所以人们将目光转向了新型的寡核苷酸药物。目前,已开发有靶向 Lp (a) 的反义寡核苷酸 Pelacarsen 和 siRNA 药物 Olpasiran。Olpasiran 的二期临床数据突出,显示 Olpasiran 可将 Lp (a) 水平降低高达 96%[7]。
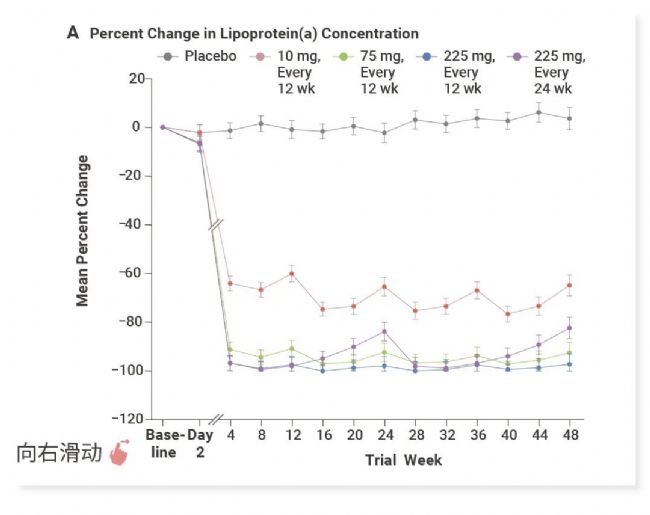
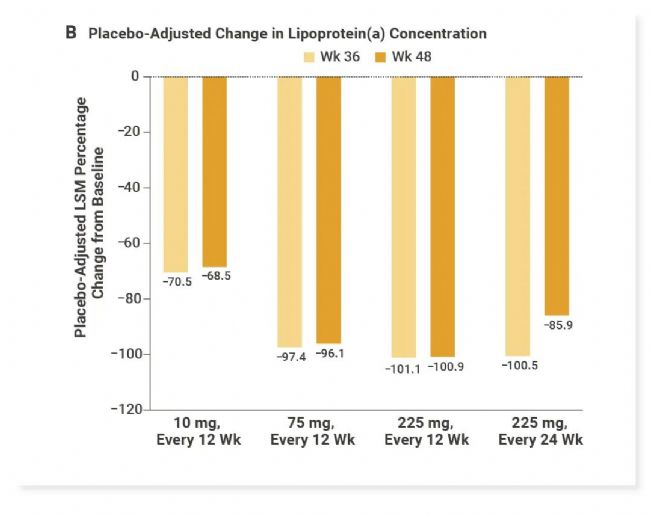
图 4. 服用 Olpasiran 后第 36 周和第 48 周 Lp (a) 浓度的变化百分比以及经安慰剂校正后脂蛋白 (a) 浓度相对于基线的平均变化百分比[7]。
此外,一款口服小分子 Lp (a) 抑制剂 Muvalaplin 通过阻断 apo (a)-apoB100相互作用来抑制 Lp (a) 形成,目前处于处于一期临床试验中。与安慰剂治疗组相比,连续 2 周每天服用剂量 ≥100 mg 的 Muvalaplin 可以使受试者 Lp (a) 水平下降 63%-65%[8]。
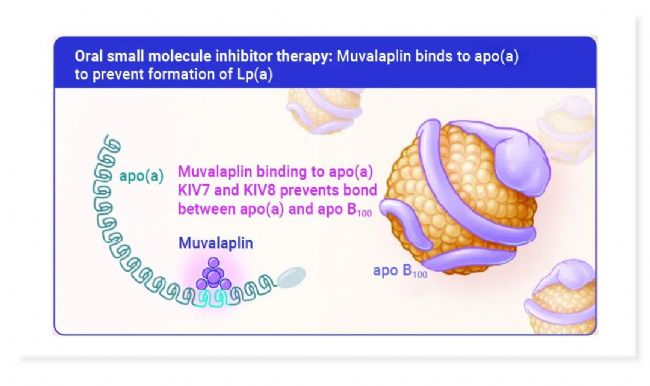
图 5. Muvalaplin 结合载脂蛋白 (a) 阻止 Lp (a) 的形成[8]。
03
APOC3
尽管降低低密度脂蛋白 (LDL) 的药物在减少心血管疾病方面取得了成功,但仍然存在持续性血脂异常,其特征是富含甘油三酯的脂蛋白 (Triglyceride-rich lipoproteins, TRL) 水平升高和 HDL 水平降低。
在血液循环中,载脂蛋白 C3 (Apolipoprotein C3, APOC3) 与 TRL 和 HDL 结合。此外,还有少量 APOC3 与 LDL 结合。APOC3 通过抑制脂蛋白脂酶 (lipoprotein lipase, LPL) 活性来增加血浆甘油三酯水平。LPL 在脂肪和肌肉组织中水解甘油三酯。APOC3 还可以通过 LDLR 和低密度脂蛋白受体相关蛋白 1 (Low-density Lipoprotein Receptor-related Protein 1, LRP1) 对富含甘油三酯的脂蛋白及其残留物的清除来增加甘油三酯和胆固醇水平[9][10]。
目前靶向 APOC3 的反义寡核苷酸 Volanesorsen 已获批上市。家族性乳糜血症综合征患者接受 Volanesorsen 治疗后平均血浆 APOC3 下降了 84%,血浆甘油三酯下降了 77%[11]。此外,还有多个药物处于临床阶段,如反义寡核苷酸 Olezarsen,siRNA 药物 Plozasiran 和 LY-3875383。
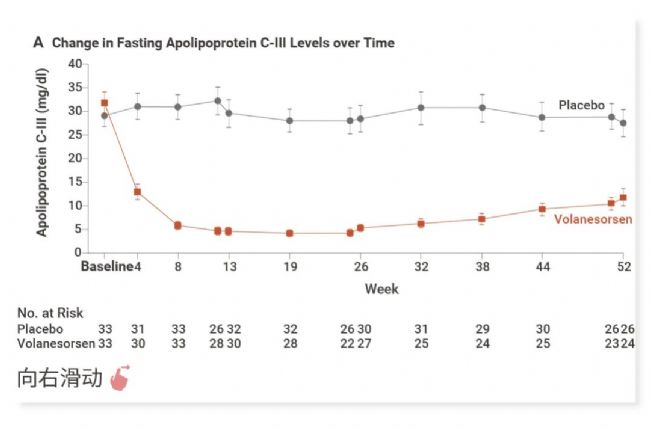
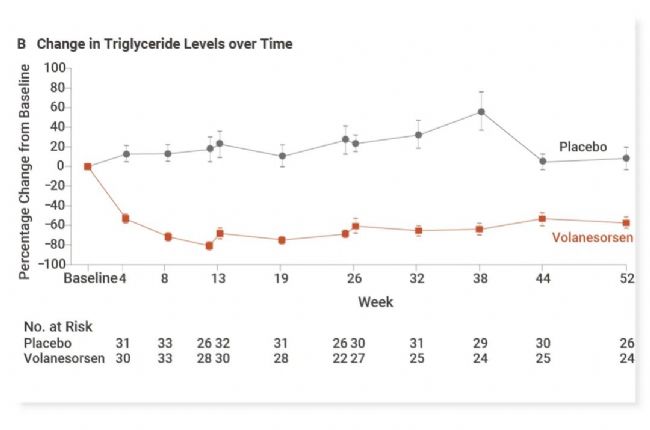
图 6. 空腹载脂蛋白 C-III 和甘油三酯水平的变化[11]。
04
ANGPTL3 抑制剂
血管生成素样 (Angiopoietin-like protein, ANGPTL) 蛋白家族的三个成员ANGPTL3、ANGPTL4 和 ANGPTL8 与 APOC3 作用相似,通过抑制脂蛋白脂酶活性来提高血浆中甘油三酯的含量。
ANGPTL3 只在肝脏中产生,与 ANGPTL8 形成功能复合体,协同抑制脂蛋白脂酶活性。与 ANGPTL3 不同,ANGPTL4 可在脂肪组织、肝脏、骨骼肌、心脏、肠道和巨噬细胞等中合成。与 ANGPTL3 类似,ANGPTL4 可以抑制脂蛋白脂酶活性。但是 ANGPTL4 敲除小鼠以及通过单克隆抗体使 ANGPTL4 失活的猴在肠系膜淋巴结中表现出脂质积累,这一结果阻碍了靶向 ANGPTL4 治疗血脂异常和动脉粥样硬化的努力。在这三种血管生成素样蛋白中,ANGPTL3 是目前最有潜力的治疗血脂异常的靶点。
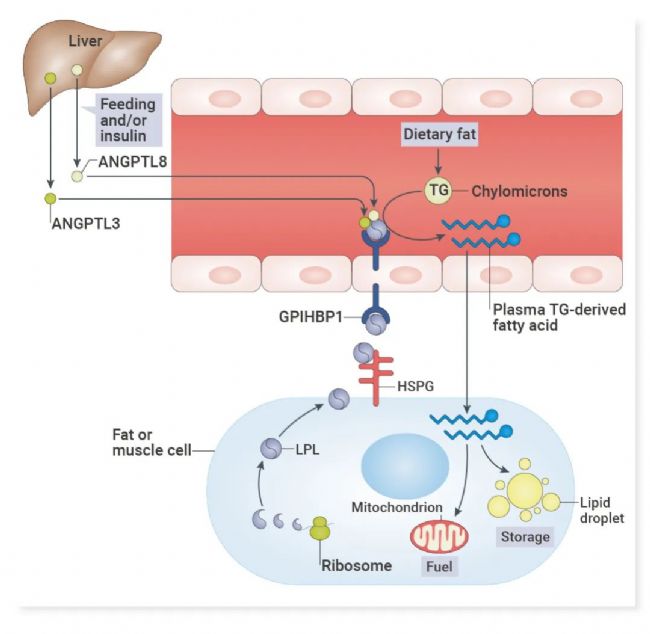
图 7. ANGPTL3 在血浆甘油三酯代谢中的作用概述[12]。
目前 ANGPTL3 抑制物有 3 个,反义寡核苷酸 Vupanorsen,siRNA 药物ARO-ANG3 和 ANGPTL3 单克隆抗体 Evinacumab,其中 Evinacumab 已获批上市。临床研究结果显示在轻中度血脂升高的健康人类志愿者中,与安慰剂组相比,接受 Evinacumab 治疗的受试者空腹甘油三酯水平最高下降 76%,LDL 水平下降 23%,且存在剂量依赖性[13]。而且接受过他汀类药物或 PCSK9 抑制剂的纯合子家族性高胆固醇血症患者在使用 Evinacumab 治疗后 LDL 水平同样显著降低。在第 24 周,Evinacumab 治疗组患者的 LDL 胆固醇水平较基线相对降低了 47.1%[14]。
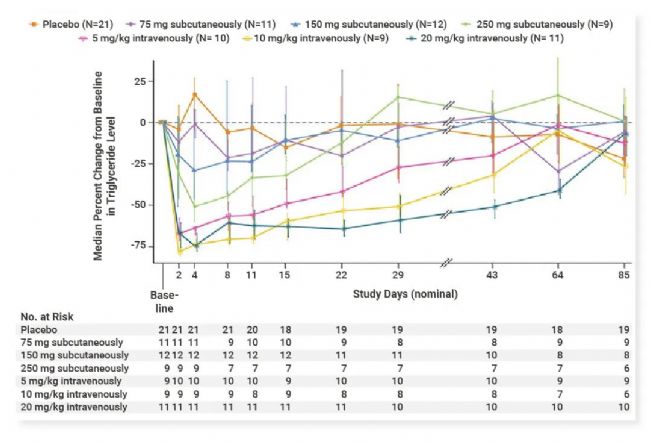
图 8. 使用单克隆抗体抑制 ANGPTL3 对人类志愿者甘油三酯水平的影响[13]。
05
小结
虽然他汀类药物在治疗血脂异常领域拥有重要作用,但其使用时也存在一些局限性。患者迫切需要更安全、强效的降低血脂的新型药物。基于这一预期,开发靶向 PCSK9、APO3、ANGPTL3 和 Lp (a) 等靶点的反义寡核苷酸、siRNA 或抗体药物为患者提供了全新的治疗模式。
| 产品推荐 |
| Inclisiran sodium (HY-132591A) 一种可抑制 PCSK9 转录的 siRNA。 |
| SPC5001 sodium (HY-148687A) 一种靶向 PCSK9 的反义寡核苷酸。 |
| Alirocumab (HY-P9928) 一种抑制 PCSK9 的人源单克隆抗体。 |
| Evolocumab (HY-P9930) 一种抑制 PCSK9 的人源单克隆抗体。 |
| Tafolecimab (HY-P99552) 一种抑制 PCSK9 的人源 lgG2 单克隆抗体。 |
| Olpasiran sodium (HY-153492A) 一种 siRNA,可减少肝脏中脂蛋白 lipoprotein (a) 的合成。 |
参考文献:
[1] Arvanitis M, et al. Dyslipidemia. Ann Intern Med. 2023;176(6):ITC81-ITC96.
[2] Jang AY, Lim S, Jo SH, Han SH, Koh KK. New Trends in Dyslipidemia Treatment. Circ J. 2021;85(6):759-768.
[3] Kim K, ,et al. New, Novel Lipid-Lowering Agents for Reducing Cardiovascular Risk: Beyond Statins [published correction appears in Diabetes Metab J. 2022 Sep;46(5):817-818.
[4] Bao X, et al. Targeting proprotein convertase subtilisin/kexin type 9 (PCSK9): from bench to bedside. Signal Transduct Target Ther. 2024;9(1):13. Published 2024 Jan 8.
[5] Ray KK, et al. Inclisiran in Patients at High Cardiovascular Risk with Elevated LDL Cholesterol. N Engl J Med. 2017;376(15):1430-1440.
[6] Duarte Lau F, et al. Lipoprotein(a) and its Significance in Cardiovascular Disease: A Review [published correction appears in JAMA Cardiol. 2022 Jul 1;7(7):776.
[7] O'Donoghue ML, et al. Small Interfering RNA to Reduce Lipoprotein(a) in Cardiovascular Disease. N Engl J Med. 2022;387(20):1855-1864.
[8] Nicholls SJ, et al. Muvalaplin, an Oral Small Molecule Inhibitor of Lipoprotein(a) Formation: A Randomized Clinical Trial. JAMA. 2023;330(11):1042-1053.
[9] Bornfeldt KE. Apolipoprotein C3: form begets function. J Lipid Res. 2024;65(1):100475.
[10] Tall AR, et al. Addressing dyslipidemic risk beyond LDL-cholesterol. J Clin Invest. 2022;132(1):e148559.
[11] Witztum JL, et al. Volanesorsen and Triglyceride Levels in Familial Chylomicronemia Syndrome. N Engl J Med. 2019;381(6):531-542.
[12] Kersten S. Angiopoietin-like 3 in lipoprotein metabolism. Nat Rev Endocrinol. 2017;13(12):731-739.
[13] Dewey FE, Gusarova V, Dunbar RL, et al. Genetic and Pharmacologic Inactivation of ANGPTL3 and Cardiovascular Disease. N Engl J Med. 2017;377(3):211-221.
[14] Raal FJ, Rosenson RS, Reeskamp LF, et al. Evinacumab for Homozygous Familial Hypercholesterolemia. N Engl J Med. 2020;383(8):711-720.
 血脂异常干预靶点及新型降脂药物
血脂异常干预靶点及新型降脂药物





















 950
950

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








