Alphafold2 开源了!!!这将进一步推动学界预测和设计蛋白。
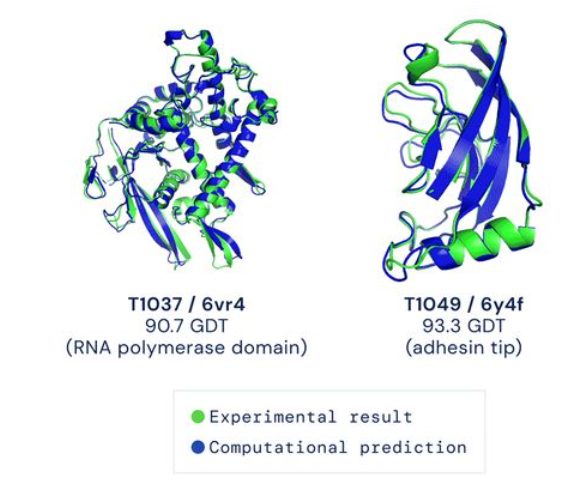
可以看到官网上给出的结果图,结构生物学实验解得的结构与预测的别无二致。
——————————— 最新最省力的方法————————————————
直接点击Colab的Alphafold2预测链接,输入序列,全部运行就可以预测了。
—————————分割线下的可以不用看啦(老)————————————
但模型的数据与预测所需资源过大,跑起来也比较费时,这里用Sergey Ovchinnikov 提供的“alphafold_single_sequence.ipynb”的代码,快速跑通看一下它的效果。
先是导入和安装各种库
%%bash
git clone https://github.com/deepmind/alphafold.git
mv alphafold alphafold_
mv alphafold_/alphafold .
%%bash
wget -qnc https://storage.googleapis.com/alphafold/alphafold_params_2021-07-14.tar
tar -xf alphafold_params_2021-07-14.tar
rm alphafold_params_2021-07-14.tar
mkdir params
mv params_* params/
%%bash
pip -q install biopython
pip -q install dm-haiku
pip -q install ml-collections
pip -q install mock
pip -q install py3Dmol
from typing import Dict
import os
import mock
import numpy as np
import pickle
import py3Dmol
from alphafold.common import protein
from alphafold.data import pipeline
from alphafold.data import templates
from alphafold.model import data
from alphafold.model import config
from alphafold.model import model
定义一个结构预测函数。
def predict_structure(
fasta_path: str,
fasta_name: str,
output_dir_base: str,
data_pipeline: pipeline







 博客介绍了AlphaFold2的开源及其对结构生物学的影响。通过Google Colab的链接,可以直接预测蛋白质结构,简化了预测流程。文章提供了一段示例代码,演示如何利用AlphaFold2预测一个特定序列的蛋白质结构,整个过程大约需要2分钟。此外,还展示了如何通过py3Dmol查看和下载预测结果的pdb文件。
博客介绍了AlphaFold2的开源及其对结构生物学的影响。通过Google Colab的链接,可以直接预测蛋白质结构,简化了预测流程。文章提供了一段示例代码,演示如何利用AlphaFold2预测一个特定序列的蛋白质结构,整个过程大约需要2分钟。此外,还展示了如何通过py3Dmol查看和下载预测结果的pdb文件。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章
















 1200
1200

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








