作业题目
去gencode数据库拿到最新的人类的gtf文件,仅仅是挑选蛋白编码基因即可,约2万个,然后把基因名字按照字母顺序排好,取前面的三分之一,对它进行一些基因分布特征的检验,比如是否集中于某条染色体,或者其它一切你能想到的检验。
下载数据
wget -c 'http://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/release_38/gencode.v38.chr_patch_hapl_scaff.annotation.gtf.gz'
挑选蛋白编码基因
#只挑选1-22,X,Y,MT染色体上的蛋白编码基因
$ zcat gencode.v38.chr_patch_hapl_scaff.annotation.gtf.gz |awk '!/^#/ && $3=="gene" && /protein_coding/ && /^chr/' |wc -l
19955
取前面的三分之一,即19955/3,约6652个
统计排序
把基因名字按字母排序,取前1/3,然后统计其在染色体上的分布情况
$ zcat gencode.v38.chr_patch_hapl_scaff.annotation.gtf.gz |awk '!/^#/ && $3=="gene" && /protein_coding/ && /^chr/' |perl -anle '{/gene_name\s+"(.*?)"/;print "@F[0]\t$1"}' | sort -k 2 | head -6652 |cut -f 1 |sort |uniq -c |sort -k 1nr |awk 'BEGIN{print "chr\tnum"}{print $2"\t"$1}'
chr num
chr1 770
chr19 512
chr2 502
chr17 433
chr11 430
chr6 417
chr3 410
chr12 391
chr7 369
chr5 344
chrX 343
chr16 334
chr10 308
chr4 308
chr9 293
chr8 271
chr15 238
chr14 228
chr20 199
chr22 163
chr13 121
chr18 99
chr21 79
chrY 24
对分布情况可视化
gene_chr = read.table('gene_chr.txt', header = T)
barplot(gene_chr$num,names.arg=sub('chr','\\1',gene_chr$chr),xlab='Chromosomes',ylab='num of chrosome',col = rainbow( length( gene_chr$num )) ) #绘制条形图
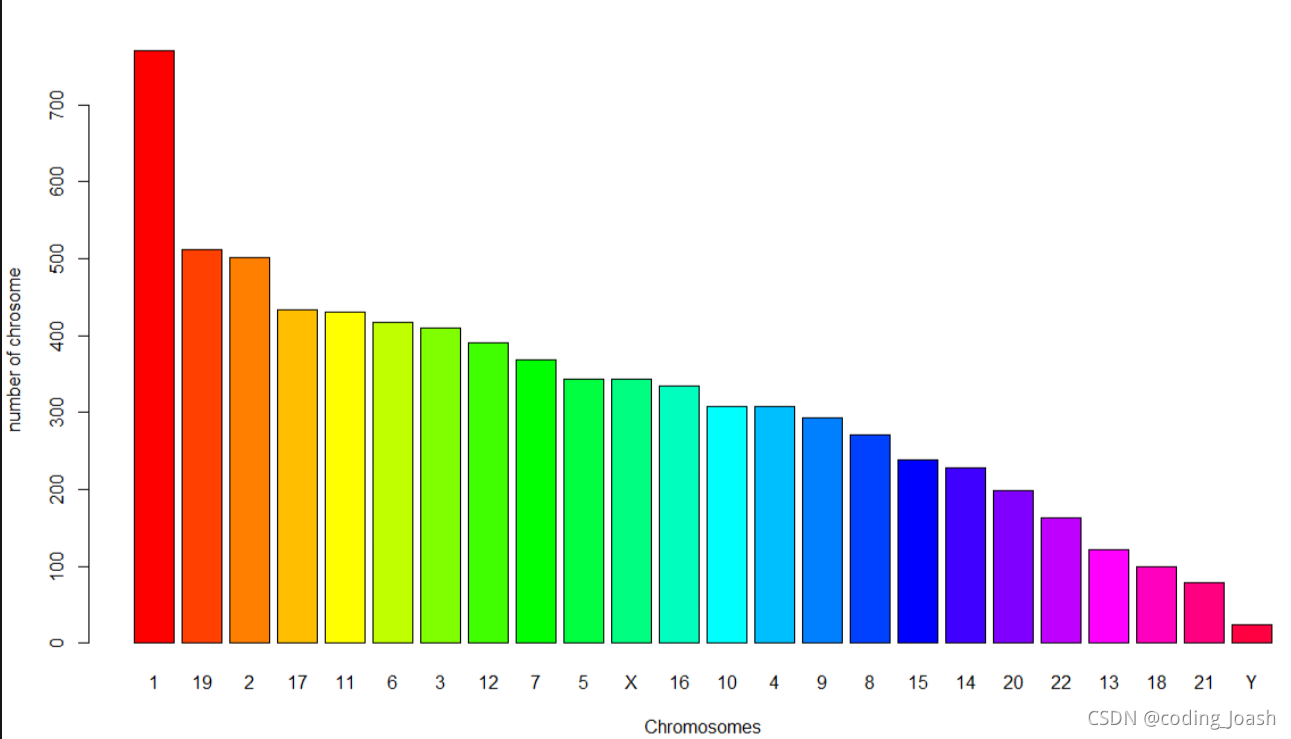







 该博客介绍了如何从gencode数据库中筛选人类蛋白编码基因,并按字母顺序排列取前三分之一进行分析。统计结果显示,这些基因在染色体上的分布不均,其中chr1、chr19、chr2等染色体上数量较多。通过可视化手段展示了这一分布特征,有助于理解基因在染色体上的分布模式。
该博客介绍了如何从gencode数据库中筛选人类蛋白编码基因,并按字母顺序排列取前三分之一进行分析。统计结果显示,这些基因在染色体上的分布不均,其中chr1、chr19、chr2等染色体上数量较多。通过可视化手段展示了这一分布特征,有助于理解基因在染色体上的分布模式。
















 1632
1632

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








