一、写在前面
10x Genomics 于 2025 年 6 月发布了 Space Ranger v4.0,这是 Visium 空间转录组分析管线的重要升级。在该版本中,新增了基于 H&E 图像的细胞分割功能(cell segmentation),可直接对 Visium HD(含 Visium HD 3’)数据进行分割。但尴尬也随之而来,现在为止,scanpy/seurat还没有现成的函数可以直接读取细胞分割后的结果:
-
Seurat::Load10X_Spatial 目前只认旧的
tissue_positions.csv; -
Scanpy 的
squidpy.read.visium()也还没适配新的cell_segmentations.geojson
于是,我们写了一个读取函数,能把Spaceranger v4 的细胞分割矩阵 + 细胞/细胞核边界 + 面积全部装进 SpatialData 对象,后续可以直接Scanpy、Seurat、Squidpy等下游分析。
二、分割输出文件说明
Space Ranger v4 在细胞分割运行完以后,会自动在输出目录下生成一个名为 segmented_outputs 的文件夹。该目录通常包含以下主要内容(详见下图):
-
filtered_feature_cell_matrix.h5:分割后每个细胞的基因表达矩阵(HDF5 格式),包含按细胞重新汇总的 UMI 计数。
-
cell_segmentations.geojson:细胞轮廓的 GeoJSON 文件,记录每个细胞边界的多边形坐标,用于空间可视化。
-
nucleus_segmentations.geojson:细胞核轮廓的 GeoJSON 文件(若分割了核,则包含核边界)。
-
raw_feature_cell_matrix.h5:未过滤的原始细胞表达矩阵。
-
其余还包括 Loupe 浏览器文件(
.cloupe)、聚类注释的 GeoJSON 等文件。
Space Ranger v4 segmented_outputs 目录结构示例(红色箭头所示分别为细胞边界信息 (.geojson)、细胞表达矩阵 (.h5)、细胞核边界信息。通过分析这些文件,可以获得单细胞级别的空间表达和形态信息。

三、Python 读取示例
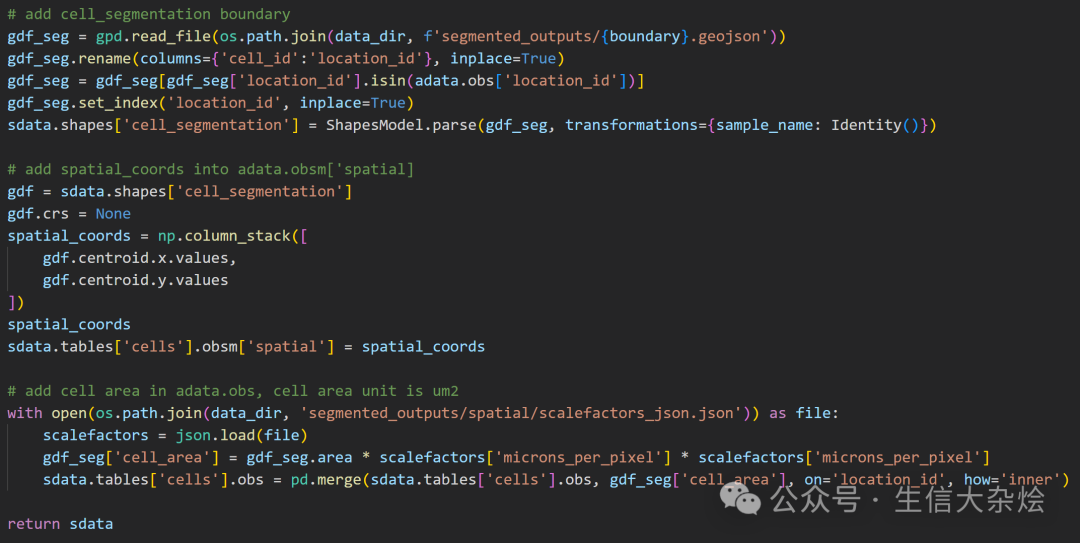
我们自定义一个读取函数,结合 spatialdata库来解析这些输出。以下是一个示例函数(假设已安装 spatialdata、scanpy、geopandas 等库):
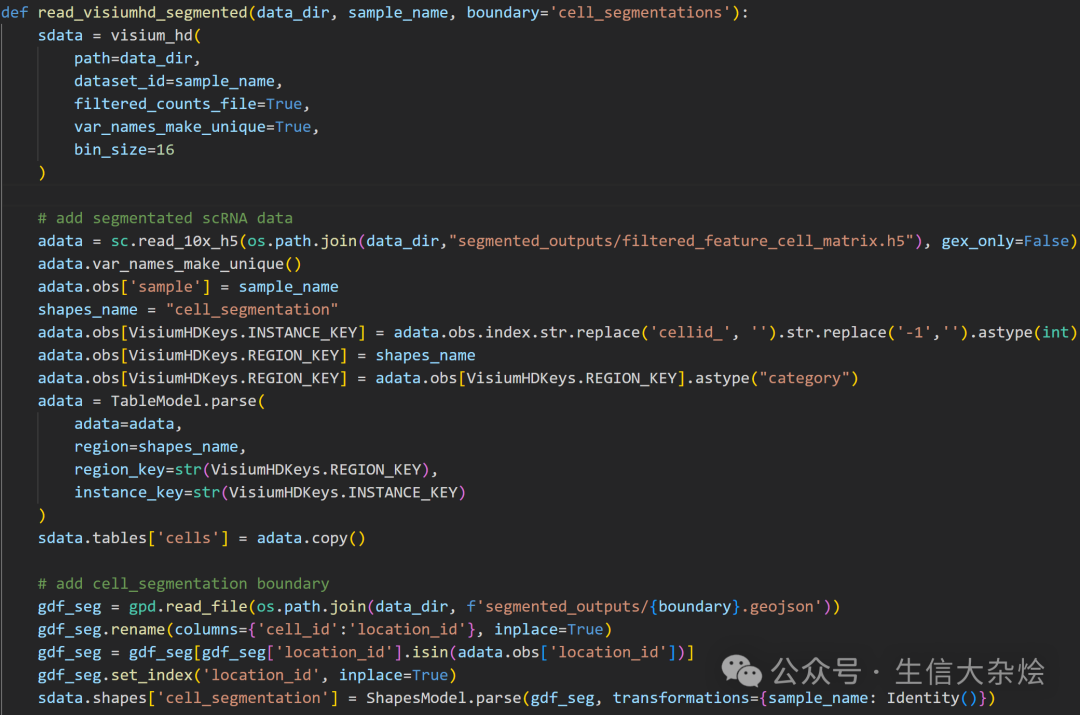
-
上述函数首先调用
visium_hd()(来自 SpatialData 库)加载原始 Visium HD 数据(包括原图像和默认的 binning 表格)。 -
然后使用
scanpy.read_10x_h5读取segmented_outputs/filtered_feature_cell_matrix.h5,得到分割细胞的表达矩阵。 -
通过设置
VisiumHDKeys.INSTANCE_KEY和REGION_KEY,将每个细胞作为独立区域(region)导入到SpatialData对象中。 -
接着用
geopandas.read_file读取cell_segmentations.geojson(或nucleus_segmentations.geojson),提取细胞(或细胞核)边界多边形信息,并用ShapesModel.parse将其解析为空间形状数据。 -
计算每个多边形的质心坐标并赋值给
adata.obsm['spatial'],同时根据scalefactors_json.json计算细胞面积,并将其并入adata.obs。
总结
Space Ranger v4.0 对 HD 数据集成细胞分割,赋予用户在近单细胞分辨率下分析空间转录组的能力。虽然目前 Seurat/Scanpy 等软件尚未官方支持该输出格式,但我们可以通过自定义加载函数,如上所示,将 Space Ranger 的分割结果导入分析流程中。感兴趣的读者可以结合更多空间分析工具(如 SpatialData),进一步探索细胞型分布、细胞间相互作用等信息。
欢迎大家帮忙阅读,点赞和转发(老板要求要有阅读量),需要代码原文件可以后台私信我哈!
























 2万+
2万+










