
关键词:基因组变异检测;全基因组测序;流行病学;
文献简介
- 标题(英文):Evidence for SARS-CoV-2 Delta and Omicron co-infections and recombination
- 标题(中文):SARS-CoV-2 德尔塔和奥密克戎合并感染和重组的证据
- 发表期刊:《CLINICAL AND TRANSLATIONAL REPORT》
- 作者单位:华盛顿大学医学中心等
- 发表年份:2022
- 文章地址: https://doi.org/10.1016/j.medj.2022.10.002

图1 文献介绍
病毒可以通过重组获得新的特性。SARS-CoV-2 德尔塔和奥密克戎变异株之间的重组可能导致一组新的突变,对病毒的传播性和严重程度产生未知影响。
在2021年11月到2022年2月期间,SARS-Cov-2在美国出现德尔塔和奥秘可容变异株共同传播并导致共感染。研究者通过对流行期29719个阳性样本进行研究。最终发现18例患者存在德尔塔和奥密克戎共感染,其中一例患者检测到德尔塔和奥密克戎重组病毒的证据。
测序流程
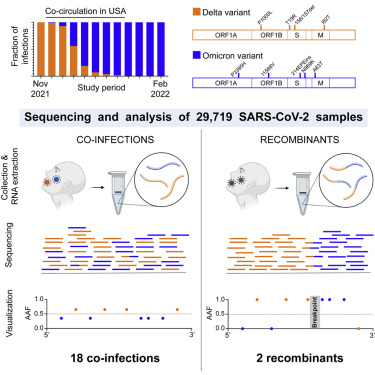
图2 工作流程图
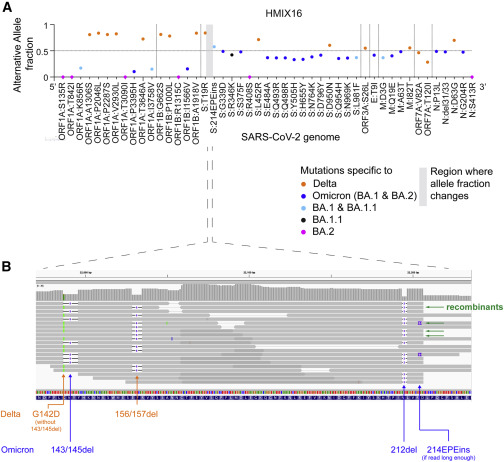
图3 HMIX16 中德尔塔-奥密克戎重组的证据
(A) Graph representing the alternative allele fraction for each mutation of sample HMIX16. Forty-seven mutations are plotted in order of their position on the SARS-CoV-2 genome from 5′ to 3′. Genes are separated by dashed vertical lines. Sixteen mutations specific to Delta are represented in orange. Nineteen mutations specific to Omicron and shared by its sublineages BA.1 and BA.2 are represented in blue. Five mutations specific to BA.1 (and BA.1.1) are in light blue. One BA.1.1 mutation is in black, and 6 mutations specific to BA.2 are in magenta. The gray box represents the region where the alternative allele fraction changes.
(B) An IGV view of the alignments for HMIX16, subsampled to only include read pairs where the first in pair covers the S:156/157del position and the second in pair covers the S:212del position. Read pairs representing three mutation profiles are present: (1) supporting Delta mutations only (7 read pairs), (2) supporting Omicron mutations only (10 read pairs), and (3) supporting a Delta-Omicron recombinant (4 read pairs) marked with a green arrow. Mutations specific to Delta are represented in orange. Mutations specific to Omicron are represented in blue. Read pairs that do not span these mutations are not shown.
该研究通过对29719样本进行测序,确定并验证了18例与德尔塔和奥密克戎共感染的病例,并在其中一例患者中发现了德尔塔和奥密克戎重组的证据。然而,无法确定是两种病毒同时感染该患者或是仅感染了德尔塔和奥密克戎重组毒株。无论是那种情况,重组德尔塔-奥密克戎重组体没有显示出增加了宿主间的传播能力。在数据分析部分,Sentieon的Haplotyper算法被用于变异检测。

图4 Sentieon的作用
Sentieon软件团队拥有丰富的软件开发及算法优化工程经验,致力于解决生物数据分析中的速度与准确度瓶颈,为来自于分子诊断、药物研发、临床医疗、人群队列、动植物等多个领域的合作伙伴提供高效精准的软件解决方案,共同推动基因技术的发展。 截至2023年3月份,Sentieon已经在全球范围内为1300+用户提供服务,被世界一级影响因子刊物如NEJM、Cell、Nature等广泛引用,引用次数超过700篇。此外,Sentieon连续数年摘得了Precision FDA、Dream Challenges等多个权威评比的桂冠,在业内获得广泛认可。
文献讨论

图5 文献讨论
该研究验证了18例Delta和Omicron新冠病毒变体合并感染的病例,并通过一系列证据排除了污染的可能性。这些证据包括样本重复提取测序结果的一致性、高比例的支持每种变体的序列读数、无交叉污染的样本测序结果以及其中一个合并感染案例中发现的重组病毒低频证据。该研究还指出了当前关于合并感染研究的不足,即难以区分真正的合并感染和污染,以及这些序列通常不被上传到公共数据库。尽管存在这些挑战,研究强调了识别合并感染并进行深度测序的重要性,这将有助于进一步了解冠状病毒中的重组现象。
总结
综上所述,研究发现德尔塔-奥密克戎重组病毒很少见,且没有证据表明德尔塔-奥密克戎重组病毒与奥密克戎毒株相比具有更高的宿主间传播能力。





















 192
192

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








