目录
1.在服务器上安装rdkit和python(3.7以上的版本)(推荐使用anaconda,此处感谢之前师兄的帮助)
1.背景知识:
合成可及性评分 (SAScore) 是一种用于计算化学和药物发现的评分系统,用于评估合成给定分子的难易程度。分数是根据分子的各种结构特征和性质计算的。
SAScore 算法通常评估以下因素:
1.官能团:已知具有合成挑战性或需要多个合成步骤的特定官能团的存在可能会导致更高的 SAScore。
2.环系统:具有复杂环系统或稠合环的分子通常具有较高的 SAScore,因为与线性或简单结构相比,它们更难合成。
3.复杂性:SAScore 计算中考虑了分子的整体结构复杂性,包括立体中心的数量、取代模式和键类型。
4.SAScore 算法为每个分子分配一个数值,分数越高表明合成可及性越低。它有助于优先考虑更容易合成的分子,从而有助于选择和设计具有更好成功合成机会的化合物。
除以上内容外:RDKit | 通过评估合成难度筛选化合物 ;基于图注意力机制的有机化合物可合成性预测
2.计算SAScore的平台:
1.RDKit:RDKit是一个用于化学信息学的开源框架,它提供了多种功能,包括计算SAScore。RDKit可以使用Python编程进行访问和操作,并且具有语言广泛的化学信息学工具。
2. ChemAxon:ChemAxon提供了许多化学信息学工具工具,包括计算SAScore的功能。他们的负载可以用于计算并评估化合物的合成可及性,并提供了一套完整的化学信息学解决方案。
3.OpenEye:OpenEye提供了一系列化学信息学工具,包括用于计算合成的可及性工具。他们的工具集可以用于计算SAScore以及其他与药物发现和合成相关的属性和指标。这些软件提供了计算SAScore的功能,可以帮助研究人成员评估化合物的合成的可及性,并进行化学合成的规划和决策。请注意,这些工具可能需要购买或获得许可证才能使用,并且可能需要一定的编程知识和技能才能使用它们进行计算和分析。
3.基于Linux系统计算SAScore的方法:
1.在服务器上安装rdkit和python(3.7以上的版本)(推荐使用anaconda,此处感谢之前师兄的帮助)
安装过程:window和Linux下RDkit包的安装 ;rdkit安装指南-常见问题解决(rdkit找不到包、下载时加载0%) ; RDKit安装
由于我的两个号下一个是有rdkit但是python2.7,一个是没有rdkit但是python3.7,我选后者进行介绍(前者我安装python3.10目前没成功:python3.10安装 ;python官网)
如何知道账号下情况的呢,如果什么都不尝试,直接使用下文提到的代码,会出现如下报错:
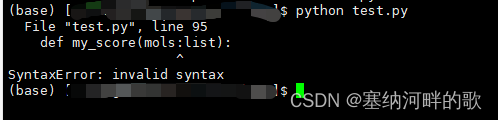
解决方案:SyntaxError: invalid syntax ; Python一直报错:SyntaxError: invalid syntax 的原因及解决办法
最大的可能就是python版本不对(输入下面后查看)
python
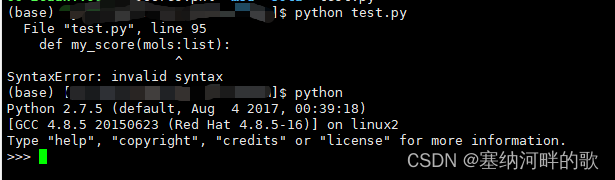
另一个是没有rdkit软件包的情况,由于我已经安装成功,现在就没办法展示了,反正也会报错,,,
( ok,在你排错完后,可以继续阅读下面内容)
在有符合要求的python版本和有anaconda的情况下:
conda install -c rdkit rdkit
大概过个几秒钟会出现这个框里的选项,我也不知道选什么,但是【y】可行
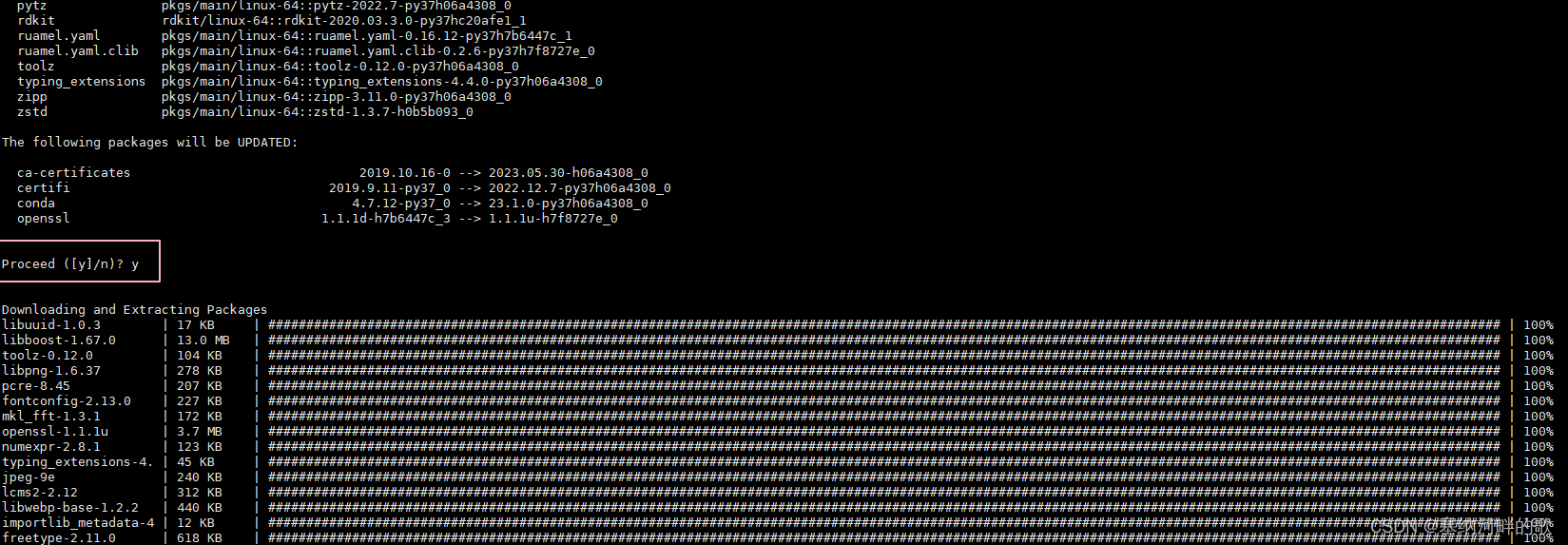
顺利完成如下所示:

2.获得化合物的SMILES码
这个软件是通过比对SMILES码进行计算打分的,因此需要先把结构式/化学式转化成SMILES码
那么你就需要了解一些可转换的途径:如何获得化合物的SMILES式
推荐使用第一个:SwissTargetPrediction (体验感不错,上手很快,我第一次接触,半小时从入门到完成,做了20个结构式)我大概介绍下:
利用SwissTargetPrediction进行成分靶点预测
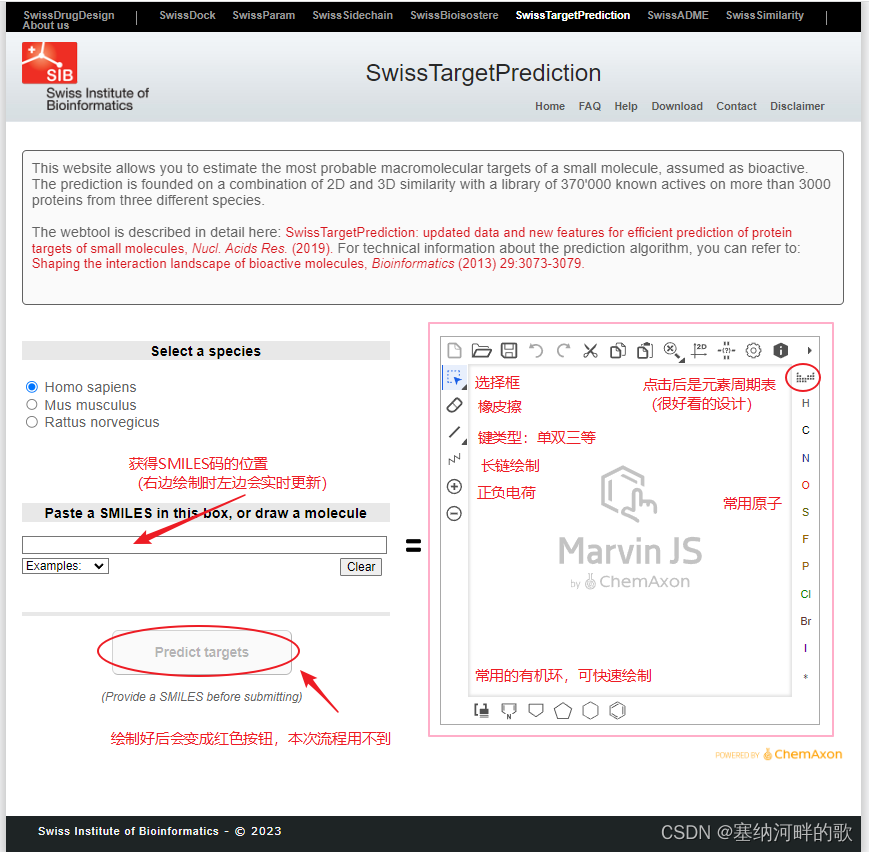
右侧绘制完后(如果电荷有误会被标注红色,很棒的设计!),在左侧的SMILES码位置全选,右侧结构会自动优化(无论你画的多标准/奇怪),再次检查后,复制SMILES码即可。
如果转换出来不确定,可以以常见化合物先去pubchem官网比对下:pubchem官网
3.运行代码进行计算
此处python代码及脚本源于前辈(万分感激):GitHub-SA_Score; RDKit | 合成可行性分数--SA SCORE ;化合物分子可合成性打分 SA_Score;rdkit QED定量评估类药性
在Linux上新建一个rdkit文件夹,下载fpscores.pkl.gz 到本地后上传到服务器账号下(不要解压!!!不要解压!!!不要解压!!!),运行前辈( GitHub-SA_Score;RDKit | 合成可行性分数--SA SCORE )给的脚本即可
如果手贱解压了就会这样(别问我咋知道的)
gzip -xvf fpscores.pkl.gz
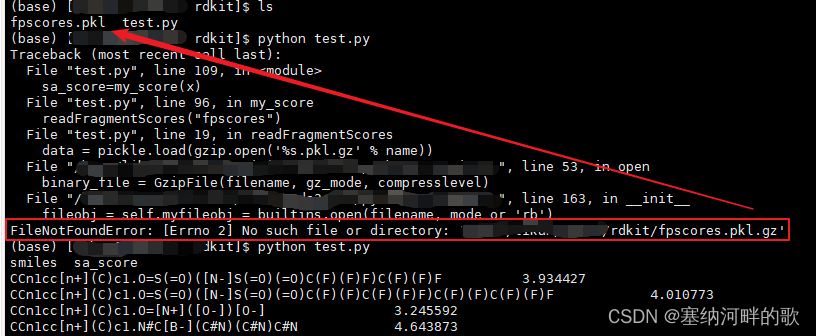
运行脚本的命令是(test.py是脚本的文件名)
python test.py
如果以上都跟着我的流程完成后,下面就会显示SMILES码和对应的分数
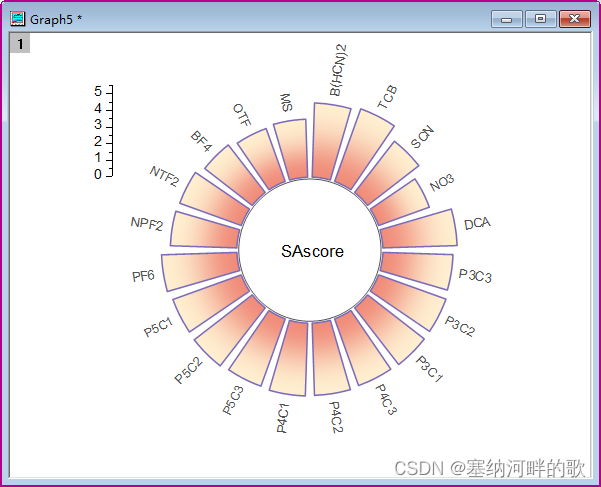
再把数据导到origin里作图即可(我试了几个,径向条形图效果最好,再修改显示即可)
花了差不多6-8h了解学习,走了很多弯路,看了很多用处不大的链接,这里仅推荐了有用的内容。再花2h整理码出来学习的思考和排错过程,思路可能不是特别清晰,但是作为新手操作的保姆级小白教程还是很不错的!




 本文提供了一篇保姆级教程,详细介绍了如何在Linux系统中使用RDKit框架计算化合物的SAScore,涉及SAScore的背景知识、安装RDKit和Python、获取SMILES码以及运行计算代码的全过程。
本文提供了一篇保姆级教程,详细介绍了如何在Linux系统中使用RDKit框架计算化合物的SAScore,涉及SAScore的背景知识、安装RDKit和Python、获取SMILES码以及运行计算代码的全过程。
















 2395
2395

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








