QaTa-COV19
V1: 该数据集由4603张COVID-19胸部x光片组成;该数据集首次包含了用于COVID-19感染区域分割任务的真值分割掩码。加上对照组的胸部x光片,QaTa-COV19由120,968张图像组成。图像位于“QaTa-COV19/ images /”文件夹下,ground-truth分割蒙版位于“QaTa-COV19/Ground-truths/”文件夹下。在4603张图像中,其中2951张具有相应的ground-truth分割掩码,其格式为mask_FILENAME.png。
V2: Qatar - cov19数据集扩展,该数据集由9258张COVID-19胸部x射线组成,其中包括用于COVID-19感染区域分割任务的ground-truth分割掩码。图像具有相应的真值分割掩码,可以在mask_FILENAME.png中找到。(目前LViT和Ariadne’s Thread都是拿这个训练的,其中LViT的一个创新点就是为这个数据集添加了文字批注,Ariadne’s Thread的一个创新点就是人为修正了LViT的批注的一些错误)
Early-QaTa-COV19:该数据集是QaTa-COV19数据集的一个子集,该数据集由1065张胸部x光片组成,其中包括无或有限症状的COVID-19肺炎病例,用于早期发现COVID-19。
Control_Group: 控制组的胸部x光片可以在Control_Group/文件夹下找到。在这个文件夹中有两个控制组。对照组i仅包括12,544张正常(健康)胸部x光片。另一方面,对照组ii包括116,365张正常胸部x线片和14张不同胸部疾病图像。在Control Group II中,CHESTXRAY-14文件夹包括CHESTXRAY-14数据集的训练集和测试集。除此之外,小儿患者的细菌性和病毒性肺炎也可以在这个文件夹中找到。
文本+图像
我直接随机抽取几个图片,mask和几个描述
covid_1.png
Bilateral pulmonary infection, two infected areas, all left lung and middle lower right lung.
双侧肺部感染,两处感染区,均为左肺和右肺中下。
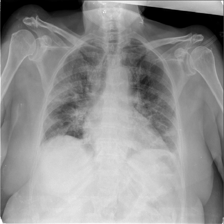
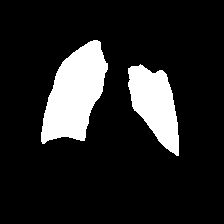
covid_1043.png
Unilateral pulmonary infection, one infected area, middle left lung.
单侧肺部感染,一个感染区,左肺中部。

covid_1238.png
Unilateral pulmonary infection, one infected area, lower right lung.
单侧肺部感染,一个感染区域,右下肺。

mask_covid_1423.png
Bilateral pulmonary infection, two infected areas, all left lung and all right lung.
双侧肺部感染,两个感染区域,左肺和右肺都感染了。
再来点纯文本描述↓
双侧肺部感染,两个感染区域,全部在左肺和中下部右肺。
双侧肺部感染,两个感染区域,全部在左肺和全部在右肺。
双侧肺部感染,两个感染区域,下部左肺和上部中下部右肺。
单侧肺部感染,一个感染区域,下部右肺。
双侧肺部感染,两个感染区域,下部左肺和中下部右肺。
单侧肺部感染,一个感染区域,下部左肺。
双侧肺部感染,三个感染区域,全部在左肺和上部中下部右肺。
单侧肺部感染,一个感染区域,中部右肺。
单侧肺部感染,一个感染区域,中下部左肺。
双侧肺部感染,三个感染区域,上部中下部左肺和全部在右肺。
双侧肺部感染,两个感染区域,中下部左肺和下部右肺。
双侧肺部感染,两个感染区域,下部左肺和下部右肺。
双侧肺部感染,三个感染区域,上部中下部左肺和上部中下部右肺。
双侧肺部感染,两个感染区域,上部中下部左肺和下部右肺。
双侧肺部感染,四个感染区域,上部中下部左肺和上部下部右肺。
单侧肺部感染,一个感染区域,中部左肺。
然后在看文本标签的时候发现其实里面大部分都是重复的,虽然七千多条数据但是只有三百多种标注,gpt总结一下它们的特征大概是↓
它们都涉及肺部感染,描述了感染的部位和区域,包括单侧感染和双侧感染,感染区域的位置(上、中、下)以及左右肺叶的涉及。这些描述中包含了不同的感染情况和位置组合。
关于语言描述这块:

模型在给出包含更详细位置信息的文本提示符时获得了更好的分割性能。
同时,当使用两种类型的文本提示时,即Stage3单独和Stage1 + Stage2 + Stage3性能几乎相同。这意味着文本提示中最详细的位置信息对提高分割性能起着最重要的作用。但这并不意味着文本提示符中其他粒度的位置信息对分割性能的改善没有贡献。即使输入文本提示只包含最粗略的位置信息(Stage1 +)在表3中的Stage2项中,我们提出的方法比没有文本提示的方法获得的Dice分数高1.43%
tips
(1)值得一提的是,多模态训练方法对数据量要求不算高
SIIM-ACR
SIIM-ACR Pneumothorax Segmentation | Kaggle
呃呃,这个数据集寄了
COVID Rural
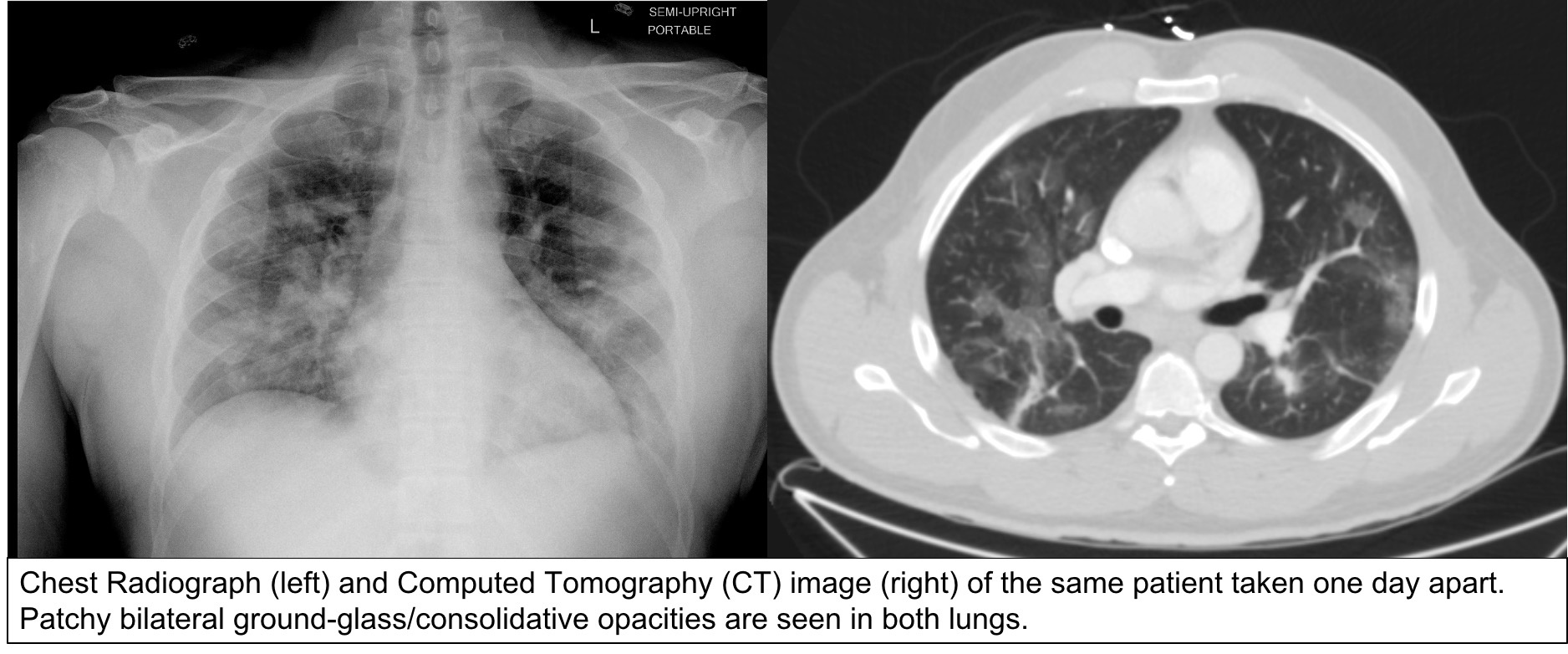
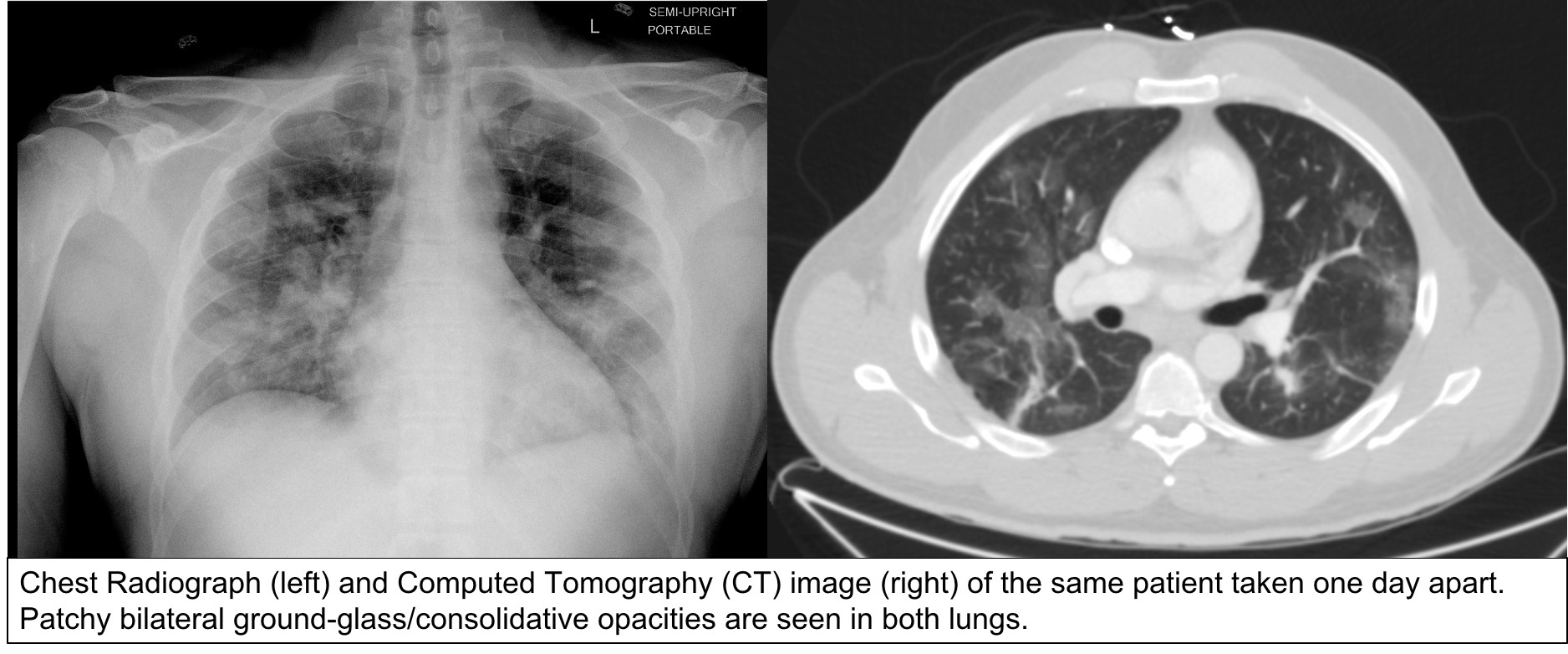
Chest Imaging with Clinical and Genomic Correlates Representing a Rural COVID-19 Positive Population (COVID-19-AR) 事实上,文献表明,关于农村人口COVID-19结局的数据非常有限,而已经确定的是,这些人群的关键合并症的表达差异很大。我们发表了一组COVID-19检测呈阳性患者的放射学和CT成像研究。每位患者都有一组有限的临床相关数据,包括人口统计学、合并症、选定的实验室数据和关键的放射学发现。这些数据与从同一种群的临床分离株中提取的SARS-COV-2 cDNA序列数据交联,并上传到Genbank存储库。我们相信这个集合将有助于定义适当的相关数据,并从这个通常代表性不足的人群中为全球研究界提供样本。
这个数据集在文章LoVT中使用过,因为下载太麻烦所以我摆烂不找图片了,反正跟文本描述关系也不是很大
文本
然后这是它的文本描述:
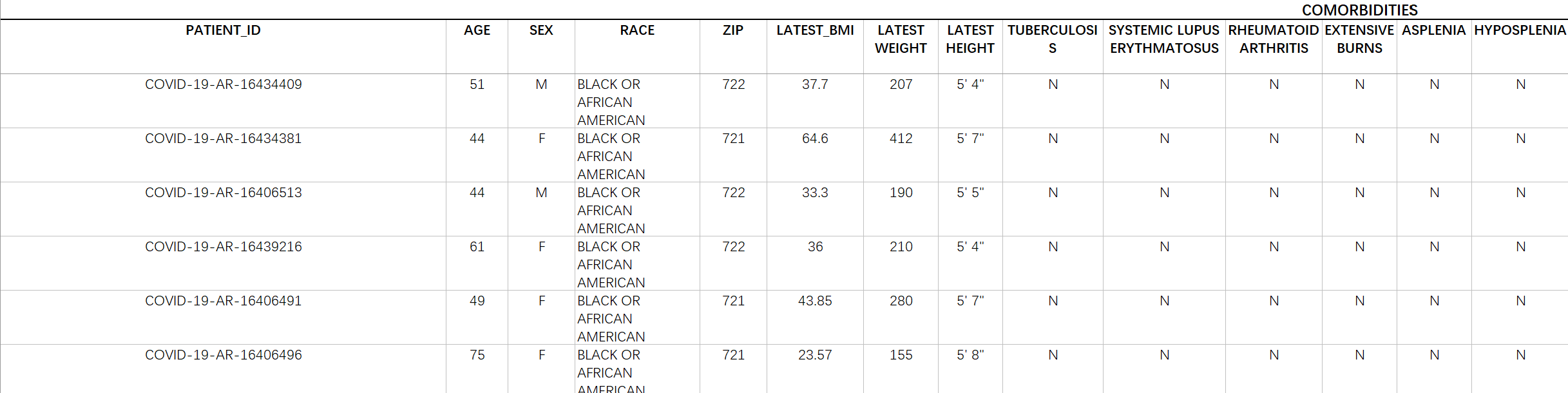
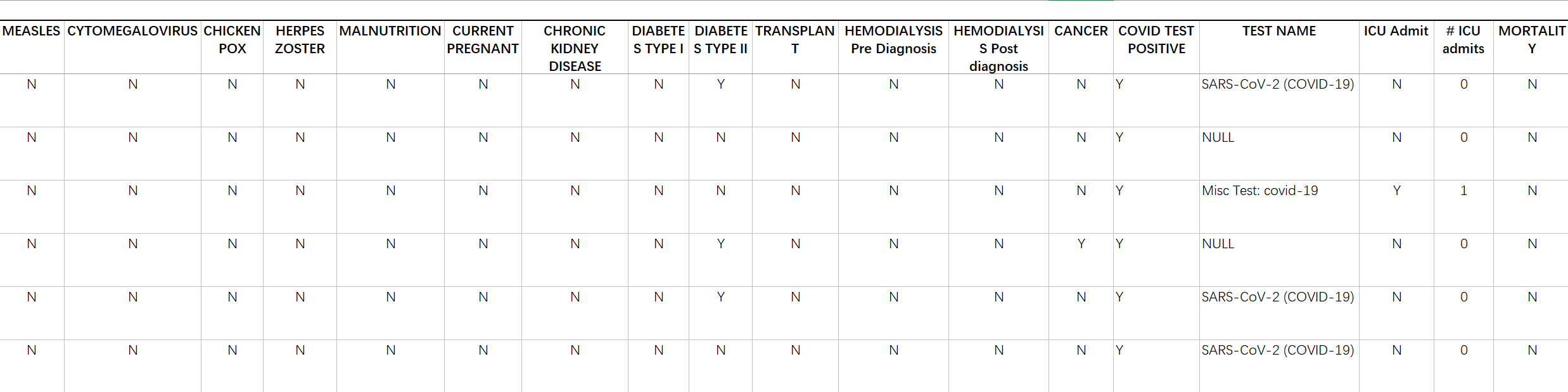
第一行从左到右描述依次是:
病人ID 年龄 性别 种族 邮编 最新BMI 最新体重 最新身高 结核病 系统性红斑狼疮 类风湿性关节炎 广泛烧伤 脾切除术 脾功能减低 麻疹 巨细胞病毒感染 水痘 带状疱疹 营养不良 目前怀孕 慢性肾脏疾病 糖尿病类型I 糖尿病类型II 器官移植 透析前诊断 透析后诊断 癌症 COVID检测阳性 检测名称 ICU入院 ICU入院次数 死亡率
我寻思这种数据收集得挺不容易的,所以真要创新我还是优先考虑第一种
继续是脑部的调研↓这里是纯图像无文本的
汇总
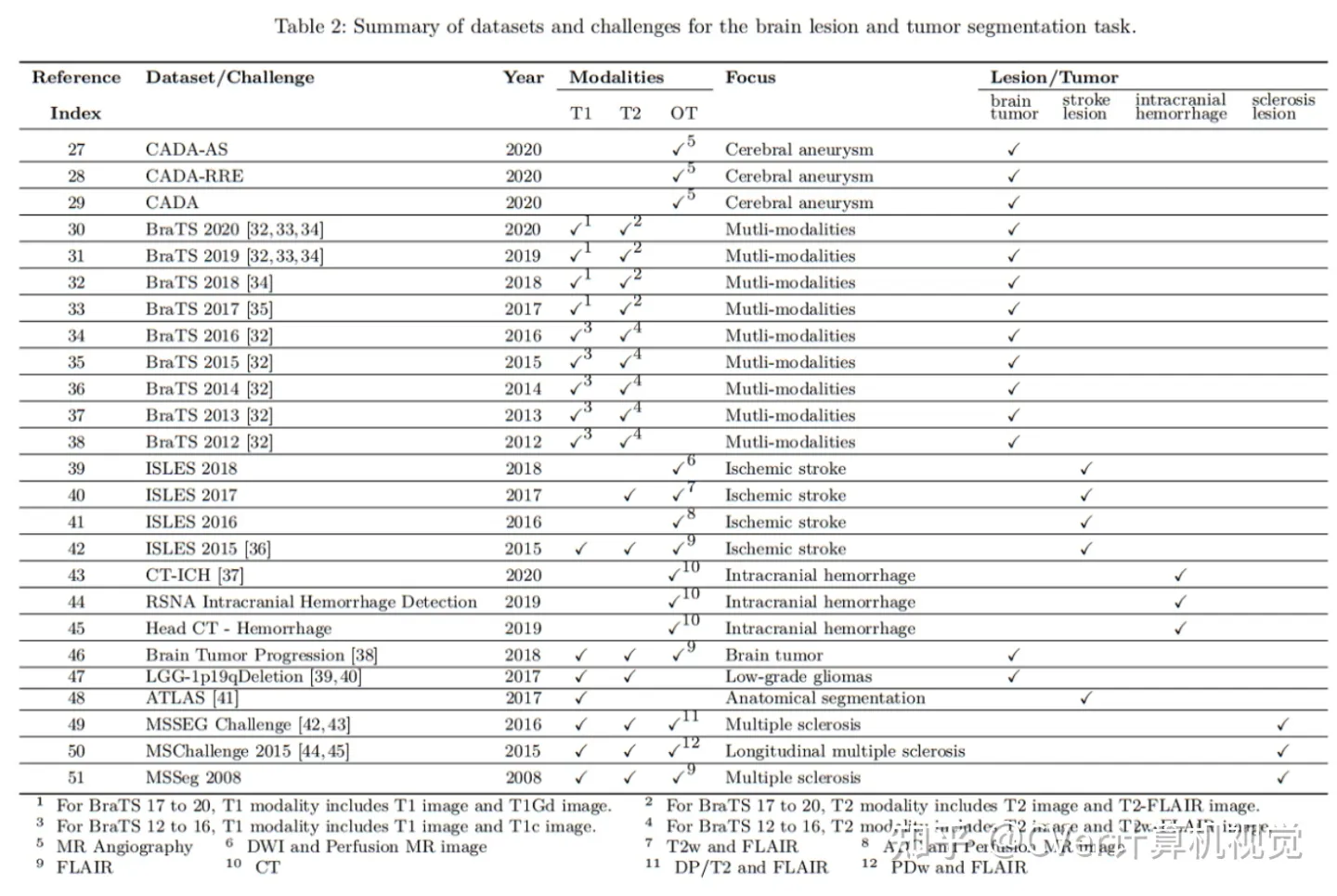
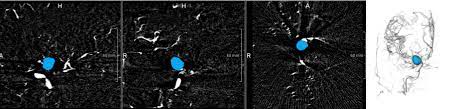
CADA
该数据集显示对比度增强的脑血管树和一个或多个动脉瘤种子点。此外,分割掩码以NIFTI (.nii.gz)掩码图像和STL (. STL)几何文件两种形式提供,并被视为ground truth。
格式:MRI
Brain US
Brain US Dataset | Papers With Code
这个大脑解剖分割数据集有1300个2D美国扫描用于训练,329个用于测试。在2010年至2016年期间接受治疗的20名不同的受试者(年龄为1岁)共获得1629张体内b型US图像。该数据集包含有IVH和没有IVH的受试者(健康受试者,但有发生IVH的风险)。美国扫描是使用飞利浦美国机器与C8-5宽带弯曲阵列换能器收集的,使用冠状和矢状扫描平面。对于每个收集到的图像,由专家超声仪手动分割心室和膈膜。我们将这些图像随机分成1300张训练图像和329张测试图像进行实验。请注意,这些图像的大小为512 × 512。
best:88.840
格式:DICOM
BRaTS
格式:MRI
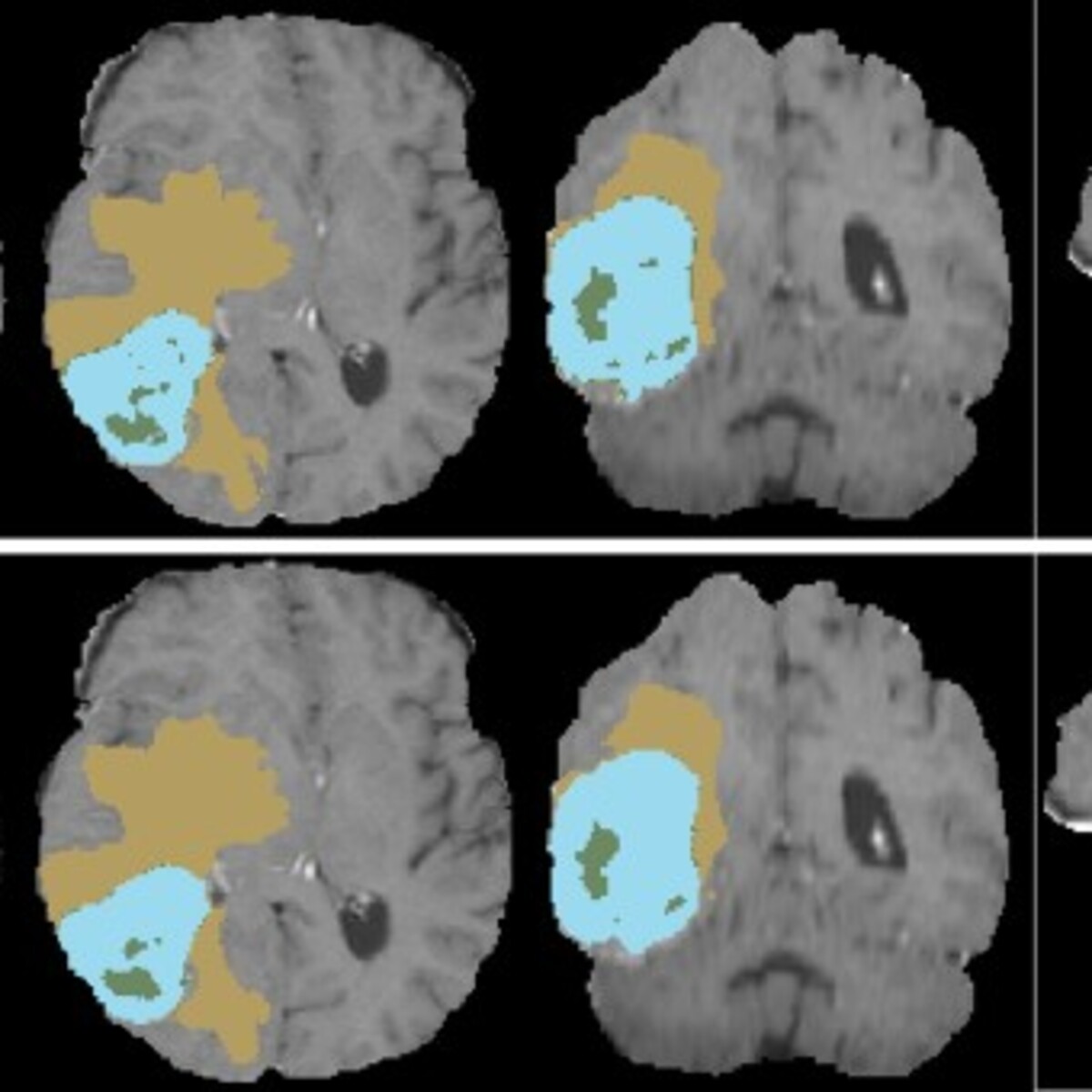
2021
BRaTS 2021 Task 1 Dataset | Kaggle
BraTS 2021脑肿瘤分割数据集介绍_brats 数据集_黄渡猿的博客-优快云博客
2019
BRATS 2019 Benchmark (Brain Tumor Segmentation) | Papers With Code
acc:0.817
BRATS 2018 Benchmark (Brain Tumor Segmentation) | Papers With Code
acc:0.870
BraTS每个病例包含四个模态的磁共振成像(Magnetic Resonance Imaging,MRI),每个模态的维度为240×240×155(L×W×H)
四种模态:
T1
T1成像,利于观察解剖结构,病灶显示不够清晰
T1ce
在受试者做磁共振之前向血液内注射造影剂,使成像中血流活跃的区域更加明显,是增强肿瘤的重要判据
T2
T2成像,病灶显示较为清晰,判断整颗肿瘤
FLAIR
T2压水像(抑制脑脊液的高信号),含水量大则更亮眼,可以判断瘤周水肿区域
ATLAS
转存失败重新上传取消
2.0
dice:0.610 ± 0.260
该挑战的目标是经验评估MR图像中病变分割的自动化方法。参与者的任务是自动生成T1w MR图像的病灶分割蒙版。
ISLES
都刷到96了,没必要
BrainPTM
Brainptm-2021 - Grand Challenge

白质束的准确定位是神经外科手术计划和导航成功的关键
MRI
mean dice score 0.7249
Instance
我们从10名经验丰富的放射科医生那里收集了200个带有精细标签的3D volume,100个用于训练数据集,70个用于封闭测试数据集,30个用于开放的验证数据集。采用DSC、HD、RVD作为分割的评价指标。这一挑战也将促进颅内出血治疗、研究者之间的互动和跨学科的交流。
DSC、HD、RVD:0.7953 ± 0.1718
看到这里感觉最合适的数据集是X光的,然后其实MRI也不错,不过之前没人做过倒是可能会有数据对齐的问题,这里确实可以琢磨琢磨。图片多模态的暂时就先不考虑了(?)
BONBID-HIE2023
今年才开始的新任务,新比赛
缺氧缺血性脑病的波士顿新生儿脑损伤数据集
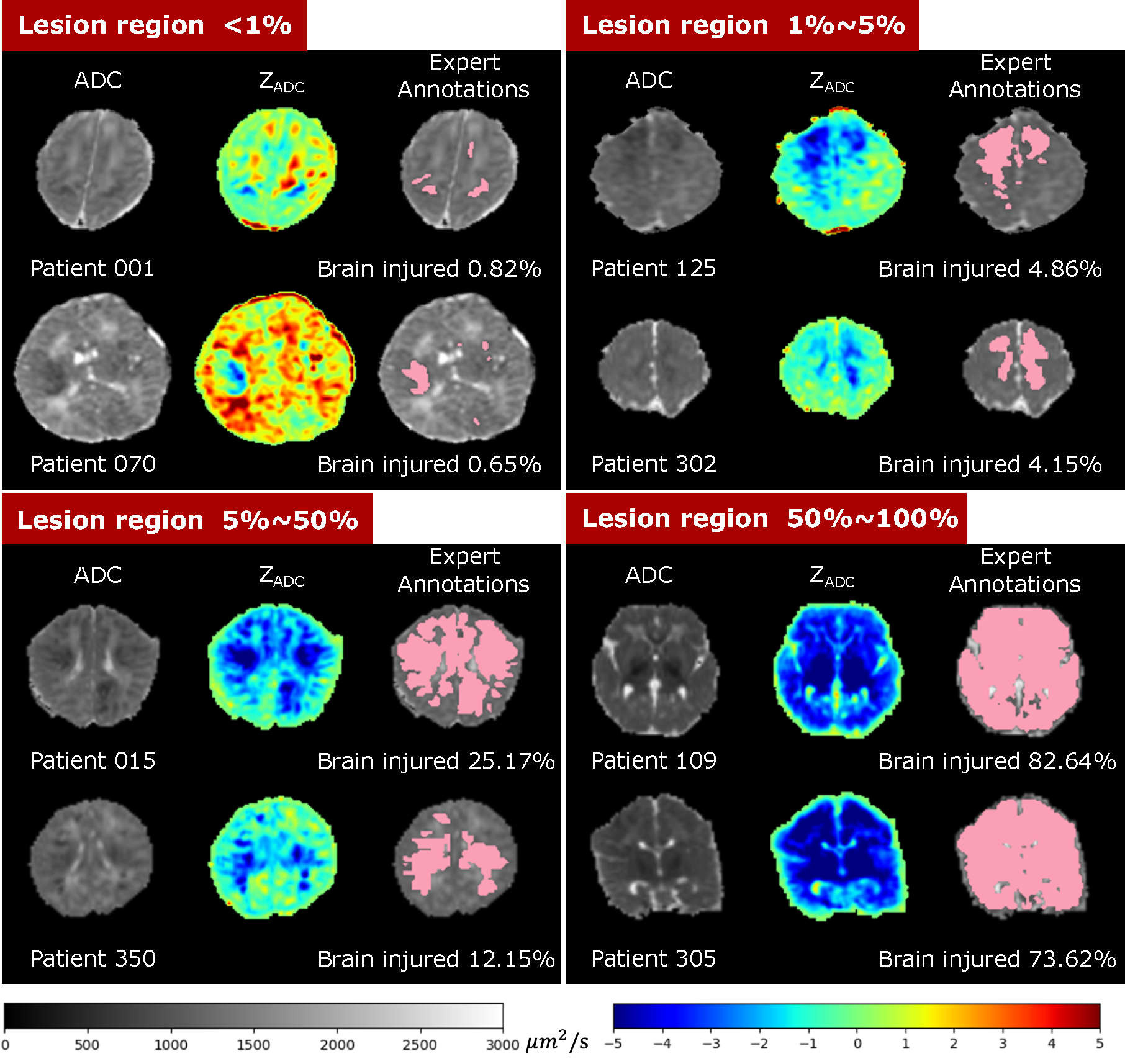
脑MRI中与hie相关的脑异常通常是弥漫性的(即多灶性),并且很小(超过一半的患者病变占脑体积的<1%)。HIE MRI数据的分割与其他分割任务(如具有大病灶和局灶性病变的脑肿瘤)明显不同,并且可以说比其他分割任务更具挑战性。例如,到目前为止,与U-Net和其他最先进的关于这种疾病的出版物的Dice重叠率保持在0.5左右,而脑肿瘤的Dice重叠率超过0.8。为实现准确的早期预后和医疗诊断,这是一项迫切但尚未得到满足的需求
数据集内容:
1ADC_ss:颅骨剥离表观扩散系数(ADC)图。
2Z_ADC: ZADC映射。
3LABEL:专家病变注释。
0.6115 ± 0.2567
好了又查了一圈脑部确实没有X光的数据集,现在要去看MRI怎么和文本对齐了。否则就Brain US的超声图像。
现在MRI面对的问题是现有CVPR2023的唯二的两篇3d多模态都是基于点云的,有点盲区了。但是个人认为还是能通过非点云方式解决的,主要是参考它数据对齐的方式
然后超声的那个应该是小孩子在子宫里的大脑,没有一个相对固定的位置,所以很难定位。








 文章介绍了QaTa-COV19数据集,包含大量COVID-19胸部X光片和对应的感染区域分割掩码,用于研究和早期识别。文章还提到了LViT和AriadnesThread等模型对该数据集的应用。
文章介绍了QaTa-COV19数据集,包含大量COVID-19胸部X光片和对应的感染区域分割掩码,用于研究和早期识别。文章还提到了LViT和AriadnesThread等模型对该数据集的应用。

















 1976
1976

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








