Cox回归分析具体示例与步骤
Cox回归分析是一种用于研究多个因素对生存时间影响的统计方法,广泛应用于医学、金融等领域。以下是一个具体的示例和详细的操作步骤,帮助你在SPSSAU(在线SPSS)平台上进行Cox回归分析。
示例背景
假设我们有一组医学数据,研究患者的生存时间与年龄、性别、治疗方式和淋巴结转移之间的关系。数据包括以下变量: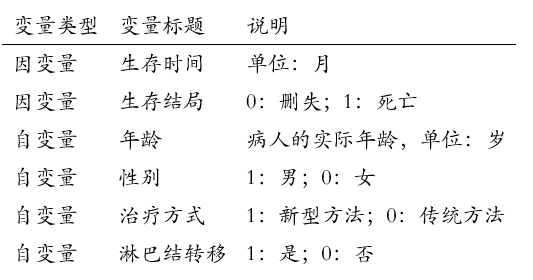
具体步骤
- 准备数据并进行Cox回归
- 将数据导入SPSSAU(网页SPSS)平台。
- 在仪表盘中依次选择【实验/医学研究】→【Cox回归】模块。
- 从左侧的标题框选中【生存时间】变量拖拽至【Y1(生存时间)】分析框中。
- 选中【生存结局】变量拖拽至【Y2(生存状态,0表示生存,1表示死亡)】分析框中。
- 将分类自变量【性别】【治疗方式】【淋巴结转移】拖拽至【X(定类,画图)[可选]】分析框中。注意,多分类因素平台默认以第一个水平作为参照进行哑变量设定。
- 将连续自变量【年龄】拖拽至【X(定量)[可选]】分析框中。
- 本例无分层考虑,因此【分层项】暂忽略。
- 进行PH等比例风险检验
- 首先对各因素是否满足PH等比例风险假定的条件进行检验。
- SPSSAU平台提供PH假定的显著性检验方法,适用于定类与定量数据。
- 当p值大于0.05时认为满足PH假定;如果p值小于0.05则说明不满足PH假定。
- 当不满足PH假定时,可将不满足条件的自变量作为分层变量,用其余变量进行Cox回归;或考虑采用依时协变量的Cox回归模型分析。
- 建立Cox回归模型与因素分析
- 评价Cox回归模型拟合质量。
- 检验各因素显著性。
- 解释和分析影响因素,以及HR(风险比)。
结果解读
- 模型拟合质量:通过查看模型的拟合优度指标,如-2对数似然值、AIC等,评估模型的拟合效果。
- 因素显著性:查看各因素的p值,判断其对生存时间的影响是否显著。
- HR(风险比):解释各因素对生存时间的影响程度,HR大于1表示增加风险,小于1表示降低风险。




















 1572
1572

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








