摘要
生命系统中的亚细胞扩散反映了细胞过程和相互作用。光学显微镜的最新进展允许以前所未有的精度跟踪单个物体的纳米级扩散。然而,从亚细胞环境中分子和细胞器的扩散中自动提取功能信息是劳动密集型的,并构成了重大挑战。在这里,我们介绍了 DeepSPT,这是一个集成在分析软件中的深度学习框架,用于以快速有效的方式解释对象的扩散二维或三维时间行为,而不受知论。为了展示其多功能性,我们将 DeepSPT 应用于病毒感染早期事件的自动映射,在识别内体细胞器、网格蛋白包被的凹坑和囊泡这三种结构时,其预测的F1准确性分别达到了81%、82%和95%,并且在几秒钟内而不是几周内。DeepSPT 仅从扩散中有效地提取生物信息的事实表明,除了结构之外,运动还编码分子和亚细胞水平的功能。
F1评分
一种衡量模型准确性的指标,它综合了精确率(precision)和召回率(recall)。精确率是指模型预测为正的样本中实际为正的比例,召回率是指所有实际为正的样本中被模型正确预测为正的比例。F1评分是精确率和召回率的调和平均值,取值范围在0到1之间,值越高表示模型的预测越准确。
介绍
细胞过程的直接观察通常通过荧光显微镜和单颗粒追踪 (SPT) 技术实现。这些技术提供了必要的时空分辨率,以定位和追踪单个生物大分子的扩散——从小蛋白质和病毒到细胞器或整个细胞——在二维 (2D) 和三维 (3D) 环境中观察到的扩散是高度复杂的,并且表现出相当大的时空和粒子间异质性,反映了各种生物因素,例如内吞阶段、局部环境、聚合状态以及与细胞骨架、膜、分子马达、细胞器等元素的相互作用。对这种内在异质性的稳健分析对于理解潜在的生物物理过程至关重要,但也带来了巨大的挑战,仍然是从单颗粒研究中提取定量见解的主要瓶颈。
时空分辨率(Spatiotemporal Resolution)
时空分辨率指的是在时间和空间两个维度上对生物分子运动的精确测量能力。高时空分辨率意味着可以在非常短的时间间隔内(如毫秒级)和非常小的空间尺度上(如纳米级)精确地跟踪生物分子的位置。
时空和粒子间异质性(Spatiotemporal and Interparticle Heterogeneity)
时空异质性指的是生物分子在不同时间和空间位置上的扩散行为存在显著差异。粒子间异质性指的是不同生物分子之间的扩散行为也存在显著差异。
从SPT实验中提取扩散行为依赖于拟合均方位移(mean squared displacement,MSD)。这些方法通常将整个轨迹转化为一个单一的描述符,如扩散系数,但会丢失轨迹中的重要时间信息。我们和其他人最近的进展引入了特征提取和扩散指纹的概念,以分析异质行为,但这种方法没有提供时间分割。
实现时间分割——解锁生物过程固有的丰富时间数据的先决条件——是一项巨大的挑战:
- 手动标注需要大量的专业知识,并且对于大型数据集来说耗时极长,尤其是在三维情况下。
- 诸如滚动MSD和分治法等方法提供了自动时间分割,但它们依赖于窗口轨迹,这引入了时间敏感性与准确性之间的权衡,并且它们还依赖于用户定义的系统特定参数。
- 隐马尔可夫模型(HMMs)可以分割轨迹,但只有当扩散指标(通常是步长)在状态之间显著变化时才能实现。
在过去几年中,开发了其他SPT分析技术,如状态转移分析(MC-DDA、anaDDA和SMAUG),以及其他分析工具,如ExTrack、vbSPT、Momboisse等、Spot-ON和TARDIS。
均方位移(Mean Squared Displacement, MSD)
常用的分析扩散行为的方法。它通过计算粒子在不同时间间隔内的位移平方的平均值来描述粒子的扩散特性。
扩散指纹(Diffusional Fingerprinting)
通过提取轨迹的特征来描述扩散行为的异质性。这些特征可以包括扩散系数、步长、方向性等。
时间分割(Temporal Segmentation)
时间分割是指将轨迹分割成具有不同扩散行为的子段,这对于理解生物过程中的动态变化至关重要。
对扩散的准确分析挑战确定了当前在时间分割方面的最先进技术是基于机器学习。这与机器学习在生物学多个任务中展现出卓越强大的广泛趋势相一致,包括蛋白质结构预测、生命图像分析、基因组工程和药物发现。机器学习在生物学中的优势源于其能够自主学习有意义的特征表示,旨在直接从高维噪声数据中优化特定任务的性能。在最少的人为干预下,它利用微妙的规律、领域知识和传统方法常常无法访问的非线性关系。目前用于扩散时间分割的机器学习方法包括各种模型,从使用滑动窗口的随机森林到端到端深度递归神经网络。虽然这些工具在特定状态集的独特、用户定义的扩散特征下效果良好,但在表现出任意数量状态并具有广泛分布的扩散特征的系统中仍然较少探索,而这些特征常见于复杂的细胞环境。此外,目前只有少数方法可以扩展到三维轨迹。
实现异质扩散的时间分段对于克服SPT中当前的分析瓶颈至关重要;然而,解码异质行为与生物分子身份、共定位伙伴、细胞定位或生物事件时间点之间的关联可能依赖于更微妙的特征关系。虽然上述工具箱,无论是否基于机器学习,都可以提供扩散行为的分段,但它们并不是为了将生物运动与生物功能关联而设计的。目前,在荧光显微镜中识别这种生物背景是一个挑战,需进行专业分析、并行多色以及通常是超分辨率成像。这种实验设计、专业分析和生物分子实体的荧光标记在劳动力和材料上都消耗巨大,并且有损生物功能。这些挑战因为使用两到三个成像通道进行定量成像时的光谱重叠限制而更加复杂化。
扩散行为的时间分析可以通过作为正交探针来克服这些挑战,从而提取生物功能、共定位或身份,减少对荧光标记的需求,从而简化实验工作流程。然而,迄今为止,拆解异质扩散行为以提供这样的生物背景仍然基本上没有被开发。
异质性扩散
异质性扩散指的是在细胞内,不同生物分子的扩散行为存在显著差异。
正交探针(Orthogonal Probe)
一种在科学研究中用于独立验证或分析特定现象的工具或方法。在实验设计中,采用与主要研究方法不同的、独立的手段来验证结果或提取信息。这种方法有助于减少单一方法可能带来的偏差,从而提高研究结果的可靠性和准确性
在这里,我们介绍了DeepSPT,这是一种多功能的基于深度学习的工具箱,旨在快速、准确和自动地进行SPT行为的时间分析。DeepSPT促进了从2D或3D轨迹中提取生物学见解,仅基于所跟踪物体的扩散特征,正如通过捕捉内体的身份、共定位伙伴、网格蛋白包被凹坑(CCPs)的细胞定位以及检测病毒逃逸到细胞质的时间点所示。简单直观的图形用户界面(GUI)允许用户执行DeepSPT的每个核心功能:分割、扩散指纹识别和训练特定任务的分类器以预测生物信息,同时输出出版质量的图形。
内体身份(Endosomal Identity)
内体是细胞内的一种膜结构,参与物质的运输和处理。内体身份指的是区分不同类型内体(如早期内体、晚期内体)的特征。
共定位伙伴(Colocalization Partners)
在细胞内与目标生物分子共同存在或相互作用的其他生物分子。
网格蛋白包被凹坑(Clathrin-Coated Pits,CCPs)
细胞膜上的一种结构,参与物质的内吞作用。它们由网格蛋白组装而成,能够包裹细胞外的物质并将其内化。
结果
DeepSPT
DeepSPT是一个深度学习框架,包括三个顺序连接的模块(图1a):
- 一个时间行为分割模块(temporal behavior segmentation module):该模块将单个粒子的轨迹分割成具有不同扩散行为的子段。例如,它可以识别轨迹中的正常扩散、定向运动、受限运动和亚扩散等不同状态。
- 一个扩散指纹模块(diffusional fingerprinting module):该模块将每个识别出的扩散行为段转换为一组描述性扩散特征,这些特征可以包括扩散系数、步长、方向性等。这些特征被称为“扩散指纹”,可以用于进一步的分析。
- 一个任务特定的下游分类模块(task-specific downstream classifier module):该模块利用实验数据来学习特定于研究系统的任务。例如,它可以学习如何根据扩散行为预测生物分子的身份、细胞定位或生物事件的时间点。
作为输入,DeepSPT接受任何粒子追踪器的输出:随时间变化的x、y和z坐标定位数据,这些数据形成了一组轨迹数据集。前两个模块可以直接应用于由x、y、(z)和时间(t)坐标特征化的任何轨迹数据集,适用于各种生物系统。最后一个模块利用实验数据学习特定于研究系统的任务。
用户可以使用任何单独的模块或完整的流程:
-
单独使用模块:用户可以选择单独使用任何一个模块,例如,仅使用时间行为分割模块来分析轨迹中的不同扩散状态。
-
使用完整流程:用户也可以使用完整的DeepSPT流程,从轨迹分割到特征提取,再到特定任务的分类,以获得全面的分析结果。
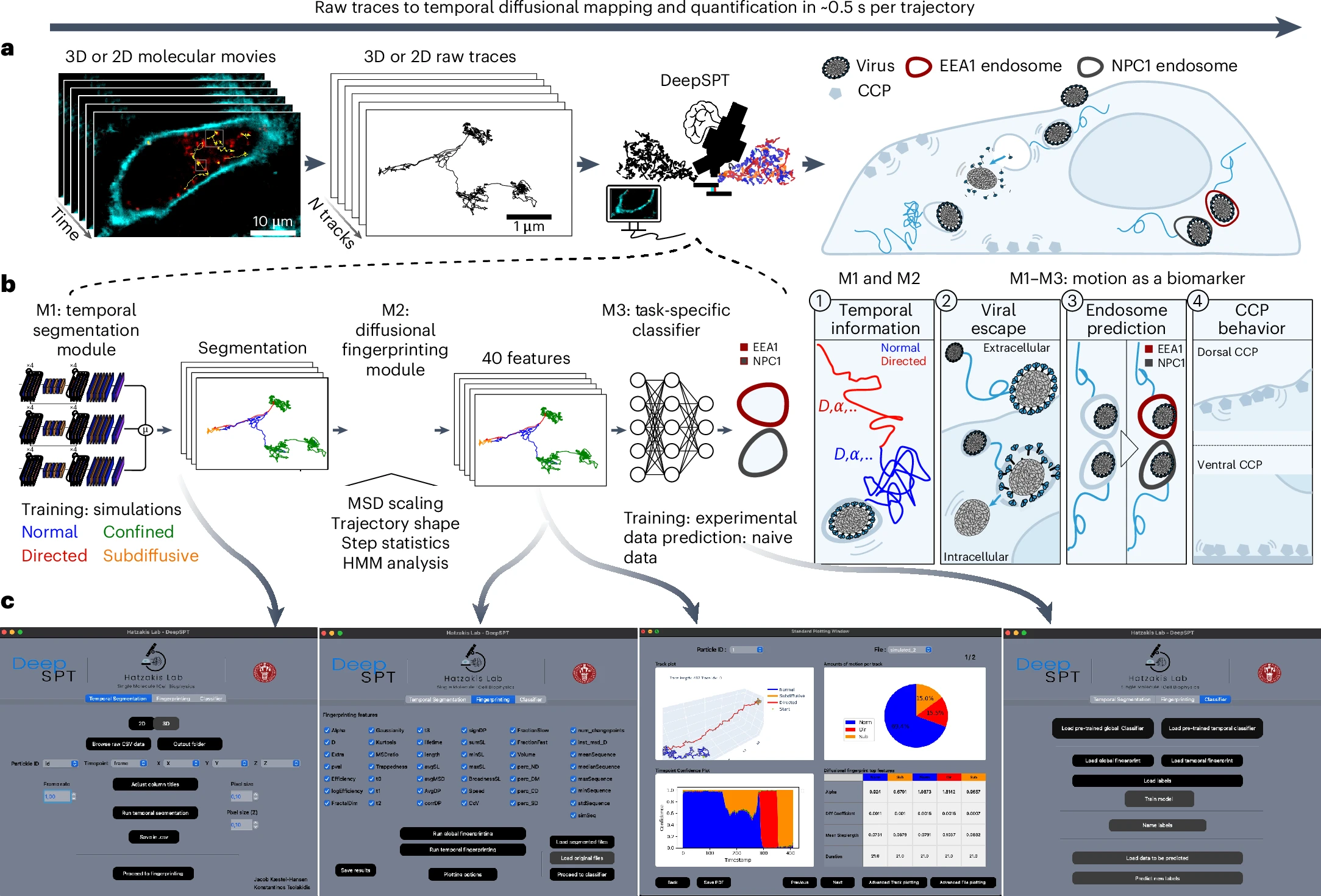
a. 深度 SPT 管道的示意图:通过荧光显微镜成像获得的二维或三维分子影片为每个粒子生成一组 x、y、(z) 和 t 局部定位信息,从而形成单粒子轨迹数据集。这些轨迹直接输入到深度 SPT 中,深度 SPT 包括一个时间行为分割模块(M1)、扩散特征识别模块(M2)和一个特定任务分类器(M3)。深度 SPT 的各个模块在放大图中可见:
- 首先,时间分割模块按时间点对扩散行为进行分类,适用于任何扩散行为(如正常扩散、定向扩散、受限扩散或亚扩散)。
- 其次,由扩散特征识别模块通过多个扩散描述符对分割后的轨迹进行量化。
- 最后,利用每个轨迹的时间信息和扩散指纹进行任务特定分类器的训练,以学习感兴趣的问题,例如基于货物的扩散行为识别内体身份。
整个深度 SPT 管道每个轨迹的计算时间约为 500 毫秒。
b. 深度 SPT 管道所支持的选定生物应用的示意图:
- 时间扩散行为的分割、分析与量化。基于扩散行为的变化,利用深度 SPT 揭示生物学见解的应用
- 生物事件的时间点识别,例如检测病毒逃入细胞质
- 直接使用内体运动或仅从其货物的移动来预测内体身份
- 预测 CCP 的细胞定位。
c. 深度 SPT 多模块管道集成的独立、免费使用的图形用户界面的截图:用户友好的图形用户界面允许用户直接在原始轨迹上执行深度 SPT 的所有核心功能,包括分割、扩散指纹识别、实时绘图和数据评估、分类器训练以及输出适合发表的图形。
时间行为分割模块(temporal behavior segmentation module)
组成:时间行为分割模块由三个预训练的不确定性校准U-Net组成,这些U-Net适应于使用二维或三维单颗粒轨迹,接受二通道或三通道一维卷积。
U-Net是一种深度学习架构,最初用于医学图像分割。该网络对图像中的每一个像素点进行分类,最后输出的是根据像素点的类别而分割好的图像。它由一个编码器(下采样)和一个解码器(上采样)组成,通过跳跃连接(skip connections)将编码器和解码器连接起来。
不确定性校准(Uncertainty Calibration)
不确定性校准是一种技术,用于调整模型的预测不确定性,使其更接近实际的不确定性水平。这有助于提高模型的可靠性和可解释性。
功能:时间分割模块将单颗粒轨迹转换为特征明显的不同扩散行为的子段,直接处理来自x、y、(z)和t坐标的输入,使用全卷积网络(fully convolutional networks)的集合。
全卷积网络(Fully Convolutional Networks)
完全由卷积层组成,没有全连接层。它能够处理任意大小的输入,并生成与输入大小相同的输出。这使得全卷积网络特别适合于像素级的任务(每个输出像素对应输入图像中的一个像素),如图像分割。
输出:除了预测的扩散行为外,轨迹中的每个时间点还会被分配一个概率估计,用于识别每种扩散类型。
本研究主要集中在生物系统中报告的扩散行为,包括:
- 正常扩散(normal),典型的无阻碍随机运动;
- 定向扩散(directed),常常由分子马达展示;
- 受限扩散(confined),在有限空间内,具有反射边界的运动,如小膜结构;
- 次扩散运动(subdiffusive),更受限的运动,通常观察到在密集的人细胞质环境中。
该模块的训练使用了一个包含900,000条轨迹的广泛数据集,展示了广泛分布的扩散属性,包括跨越四个数量级的扩散系数变化、不同的轨迹持续时间、变化的定位误差以及在其生命周期内展示多种随机长度扩散行为的轨迹。这个广泛的训练集扩大了DeepSPT在不同生物系统和实验条件下的适应性。DeepSPT可以被训练来识别其他扩散属性和多样化的运动类型,或者在均匀运动的情况下简单地预测一个统一的全局扩散状态。
扩散指纹模块(diffusional fingerprinting module)
扩散指纹模块将每个识别的扩散行为段转化为一套全面的40个描述性扩散特征,这些特征不仅包括之前工作中提到的17个特征(参考文献13),还扩展到40个特征,增加了时间特征。参考文献13和DeepSPT中的扩散指纹模块都是分析异质行为的工具,尽管它们不提供分段功能。DeepSPT的扩散指纹模块具有双重目的:它有助于用户解读个体行为段的量化,并生成对于下游分类任务至关重要的特征表示。
描述性扩散特征(Descriptive Diffusional Features)
描述性扩散特征是一组用于描述扩散行为的量化指标,如扩散系数、步长、方向性等。这些特征帮助用户理解和解释扩散行为的特性,同时也为机器学习模型提供输入。
时间特征(Temporal Features)
时间特征是描述扩散行为随时间变化的特征,如时间依赖的扩散系数、时间依赖的步长等。
任务特定的下游分类模块(task-specific downstream classifier module)
功能:该模块直接在实验数据(这些实验数据已经被上述两个模块转换为一组特征)上进行训练和预测。
-
预测轮状病毒感染的早期阶段的重要时间点:该模块可以预测轮状病毒感染过程中病毒逃逸到细胞质中的时间点。
-
区分早期内体和晚期内体:该模块可以基于扩散行为识别内体的身份,区分早期内体和晚期内体。
-
定位网格蛋白包被凹坑(CCPs)和囊泡到细胞的背侧或腹侧膜:该模块可以预测CCPs和囊泡在细胞内的位置,是位于背侧膜还是腹侧膜。(图1b)
输入:输入数据是经过时间行为分割和扩散指纹分析后的特征集。
输出:该模块输出每个类别的概率估计,仅利用扩散特性进行预测。
快速、自动化的时间扩散行为分析
为了展示DeepSPT时间分割能力的有效性和普遍适用性,我们采用了五种不同的评估方案。
- 首先,我们使用了保留方案(Holdout Scheme)来评估在训练过程中未使用的轨迹上的性能(见图2a-c)。这种方法通过将数据集分为训练集和测试集,用训练集来训练模型,然后用测试集来评估模型的性能。
- 其次,使用比训练时更广泛的扩散参数值分布的模拟轨迹来测试模型的泛化能力。这意味着模型需要能够处理在训练阶段未见过的参数范围。
- 第三,将DeepSPT的性能与现有的最先进的时间分割算法进行比较。
- 第四,在AnDi挑战中的五种不同扩散行为上,将DeepSPT与两个深度学习模型进行基准测试。AnDi挑战是一个关于异常扩散行为分析的竞赛,提供了一系列复杂的扩散行为数据。
- 最后,研究DeepSPT对超出扩散行为之外的行为类别进行分类的能力。这表明DeepSPT不仅能够区分不同的扩散行为,还能够识别其他类型的行为。
在保留验证中,研究者在包含20,000条模拟轨迹的测试集上评估了时间分割性能,其中80%的轨迹表现出异质性运动,20%表现出均匀运动(见方法部分)。这些轨迹涵盖了广泛的扩散参数和四种运动类型(见图2a、方法部分和补充图1和2)。DeepSPT不仅准确地识别了时间变化点(见图2a),还产生了时间分辨的概率估计,这些估计可以作为可调整的后处理参数(见图2a中的DeepSPT输出随时间变化的情况)。为了提高可靠性并减少过度自信,研究者使用温度缩放(temperature scaling)对这些概率估计进行了校准(见补充图3)。图2a中布朗运动轨迹的短段看似定向运动,这一事实突显了精确分段的挑战(补充图4)。
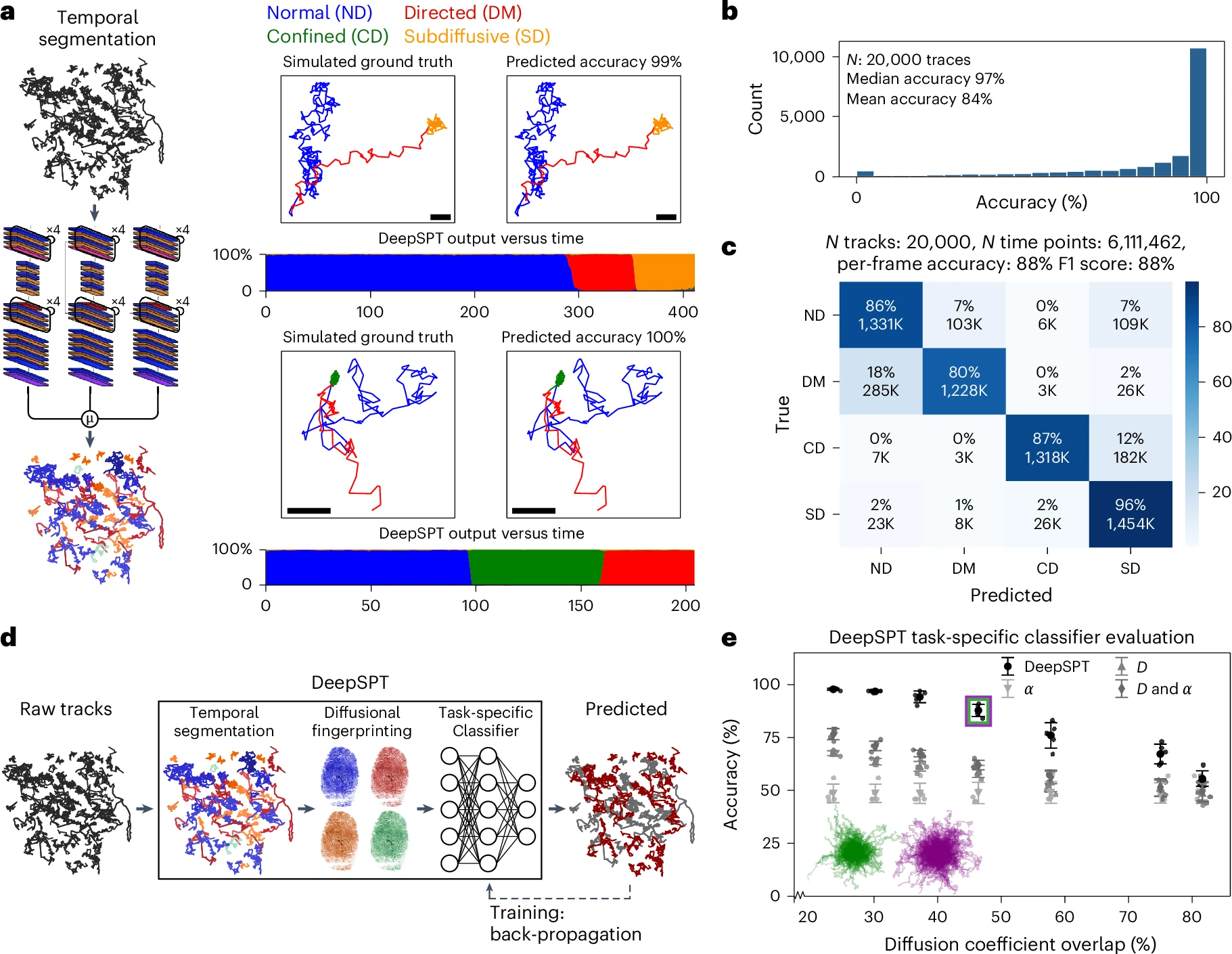
a. DeepSPT的时间分割模块的插图,展示了两个关于合成的3D轨迹和异质扩散的DeepSPT预测示例。
- 左侧:模拟真实值的2D投影,按基础扩散行为着色。
- 右侧:轨迹按DeepSPT的预测着色。尺度棒500纳米。
- 底部:每个时间点针对每种建模扩散类型的不确定性校准概率估计(DeepSPT输出与时间),提供对模型确定性的透明度(见补充图2和3以获取附加示例和不确定性校准)。
b. 测试集中每个单独3D轨迹相关精度的直方图(N轨迹,20,000;N时间点,6,111,462;方法)。
c. 基于b中20,000个测试集轨迹内所有预测的混淆矩阵(N时间点,6,111,462),总计>600万单独时间点预测。对角线条目为正确预测,非对角线表示混淆类别。每个条目报告预测的绝对数量(K=1,000)和对给定类别中标签数量的归一化。
d. DeepSPT分类管道的插图。每条轨迹通过分割模块被时间分割成四种扩散行为的每一种,通过扩散指纹模块转换为描述特征,组合成一组独特的时间和扩散特征,随后被输入到特定任务的下游分类器中。
e. DeepSPT分类管道与使用MSD特征的分类器进行基准测试:扩散系数(D)、非定常扩散指数项(α)或两者(D和α)在具有重叠分布的两类轨迹的模拟数据上。分类精度在瞬时扩散系数的增量重叠程度下进行评估。紫色和绿色轨迹描绘了在扩散系数中约45%重叠的轨迹,用紫色和绿色框表示。误差条表示标准偏差。DeepSPT在扩散系数达到75%的重叠时显著优于所有三种基于MSD特征的方法(所有P值<0.001,使用双侧Welch t检验,N=5每种条件;补充表2),在82%重叠时DeepSPT显著优于D和α(所有P值<0.05;补充表2)。
-
分类性能的量化评估:
-
整体准确性:DeepSPT在所有四种运动类型(正常扩散、定向运动、受限扩散和亚扩散)上的分类性能表现为每条轨迹的中位数准确率为96%,每帧的平均准确率为84%(见图2b和c)。
-
特定运动类型的准确性:
-
对于三种运动类型(正常、定向和受限/亚扩散),平均准确率为91%(见扩展数据图1)。
-
对于两种运动类型(正常/定向与受限/亚扩散),平均准确率为97%(见扩展数据图1)。
-
对于均匀运动,平均准确率为91%(见补充图5)。
-
-
F1值:DeepSPT在3D和2D数据集上均达到了88%的F1值(见图2c和扩展数据图1)。
-
推理时间:在所有情况下,其每个轨迹的推理时间均小于40毫秒。
-
-
不同运动类型的分类准确性:模型在区分不相似的运动类型时表现出极小的混淆,突出了DeepSPT区分受限和自由运动类型的能力(见图2c和扩展数据图1)。
-
亚扩散运动的分类准确率为96%。
-
定向运动的分类准确率为80%。
-
正常扩散和受限扩散的分类准确率分别为86%和87%。
-
-
模型的鲁棒性:
-
受限区域大小的影响:增加受限区域的大小会增加模型的混淆度并降低准确性,但DeepSPT仍然保持超过91%的中位数准确率(见补充图6)。
-
运动类别数量的影响:当模型被要求识别较少的运动类别时,区分不同行为类型的能力变得更加明显(见扩展数据图1)。
-
不同条件下的鲁棒性:DeepSPT的强大鲁棒性能在多种扩散特性、状态转换率、轨迹持续时间、跟踪误差和定位误差下得到了进一步验证,即使对于训练集中未包含的参数范围也是如此。
-
特定条件下的表现:DeepSPT在轨迹长度超过20帧、定位误差等于或小于实际扩散步长以及存在大量跟踪误差的轨迹上表现出色(见扩展数据图2-4和补充图7),这表明其对各种实验设置的适应性和鲁棒性,以及优化成像对于准确分割和精确输出扩散度量的重要性。
-
为了评估DeepSPT在分割具有四种扩散行为的异质性扩散轨迹上的能力,我们对DeepSPT进行了基准测试:
- 一种高性能的基于长短期记忆(LSTM)的方法:在处理四种、三种和两种扩散行为时的分类准确率分别为44%、58%和72%
- 广泛使用的滚动均方位移(rolling MSD)方法:在处理四种、三种和两种扩散行为时的分类准确率分别为34%、51%和65%
隐马尔可夫模型(HMM)专注于不需要大幅度变化步长即可检测扩散行为变化的方法。由于LSTM方法有二维限制,所有方法都在2D轨迹上进行了测试。相比之下,DeepSPT在相同分类下的准确率达到了88%、91%和97%,超越了当前最先进的方法,并突显其在匿名分割和异质扩散分类方面的改进能力。
长短期记忆(Long Short-Term Memory,LSTM)
特殊的递归神经网络(RNN),能够学习长期依赖关系,适用于处理时间序列数据。
滚动均方位移(Rolling Mean Squared Displacement,rolling MSD)
一种分析扩散行为的方法,通过计算轨迹中每个时间点的均方位移来评估扩散特性。
隐马尔可夫模型 (Hidden Markov Model,HMM)
一种统计模型,用于描述由一系列隐藏状态生成的观测序列,适用于处理具有时间依赖性的数据。
此外,我们将DeepSPT与2021年AnDi挑战赛上表现最好的两个模型进行比较。这两个模型分别是深度学习方法E和J。方法E使用的是与全连接层网络相结合的递归神经网络,而方法J则基于卷积神经网络。AnDi挑战调查了五种行为:
- 退火瞬态时间运动
- 连续时间随机游走
- 分数布朗运动
- 莱维走动
- 缩放布朗运动
DeepSPT针对这些扩散行为进行了重新训练,并在两组数据上进行了测试。在具有恒定长度和单一变化点的数据(参考文献28)上进行基准测试时,2D轨迹的方法E和J的中位准确率分别为79%和80%,而DeepSPT则达到了94%。对于3D轨迹,方法J不适用,方法E的中位准确率为68%,而DeepSPT达到了98%(补充图10)。在具有恒定轨迹持续时间但在五种AnDi扩散行为之间有多个变化点的异质轨迹上进行基准测试的2D轨迹,方法E和J的中位准确率分别为64%和59%,而DeepSPT达到了80%。对于3D轨迹,方法E的中位准确率为66%,而DeepSPT为82%(补充图11)。DeepSPT的改进分割准确度源于其设计和训练,以便对随机轨迹长度的任意变化点数量进行分割,而AnDi挑战则专注于固定长度轨迹的单一变化点建模。DeepSPT在AnDi任务3和异质扩散中的适应性和准确性提升,突出了其在异质扩散的无关分割及其对多个系统的可扩展性的优势(在扩展数据图5和6中提供了额外指标)。
我们随后对DeepSPT在二维实验数据集上进行了定性评估(补充图12和13)。具体而言,对于人胰岛素(HI),我们用Atto-655对其进行标记,并使用二维活细胞旋转盘共聚焦荧光显微镜记录其在HeLa细胞中的时空定位。通过DeepSPT,我们报告胰岛素的细胞内运输主要表现出亚扩散行为,但也包括定向运动的部分。定向运动与马达蛋白的扩散模式一致,表明活跃的细胞运输,确立了DeepSPT作为研究不同实验背景下运输机制的潜在工具。
生物分子身份的分类需要添加时间分段。我们结合了DeepSPT的所有模块,以展示其利用微妙扩散变化进行分类异质行为的能力。DeepSPT的分段和指纹识别模块的整合使得任何轨迹都可以转化为包含时间和扩散特征的特征表示,随后可以喂入下游分类器(图2d)。为了展示DeepSPT集成方法的描述能力,我们评估了DeepSPT在两类具有重叠分布的模拟轨迹(1,000条轨迹)上的分类性能,即,每个人群的扩散背后都是相似分布的参数。在保持所有扩散特征不变的情况下,除了扩散系数,我们通过分层五折交叉验证评估了不同类别之间扩散系数重叠程度变化时的分类准确性。DeepSPT的准确率达到了98%,即使在扩散系数重叠约57%时,仍保持76%的准确率。这一性能显著(通过Welch的t检验;图2e)优于基本的MSD特征,其准确率范围约为49%到76%(图2e)。对于在模拟轨迹间切换一次且约有75%扩散重叠的轨迹的变点预测准确性与五个基准方法的比较(图2e),显示DeepSPT明显优于这些基准方法(扩展数据图7),突显了DeepSPT辨别和利用扩散特征微妙差异的能力。
使用运动加速病毒逃逸的检测
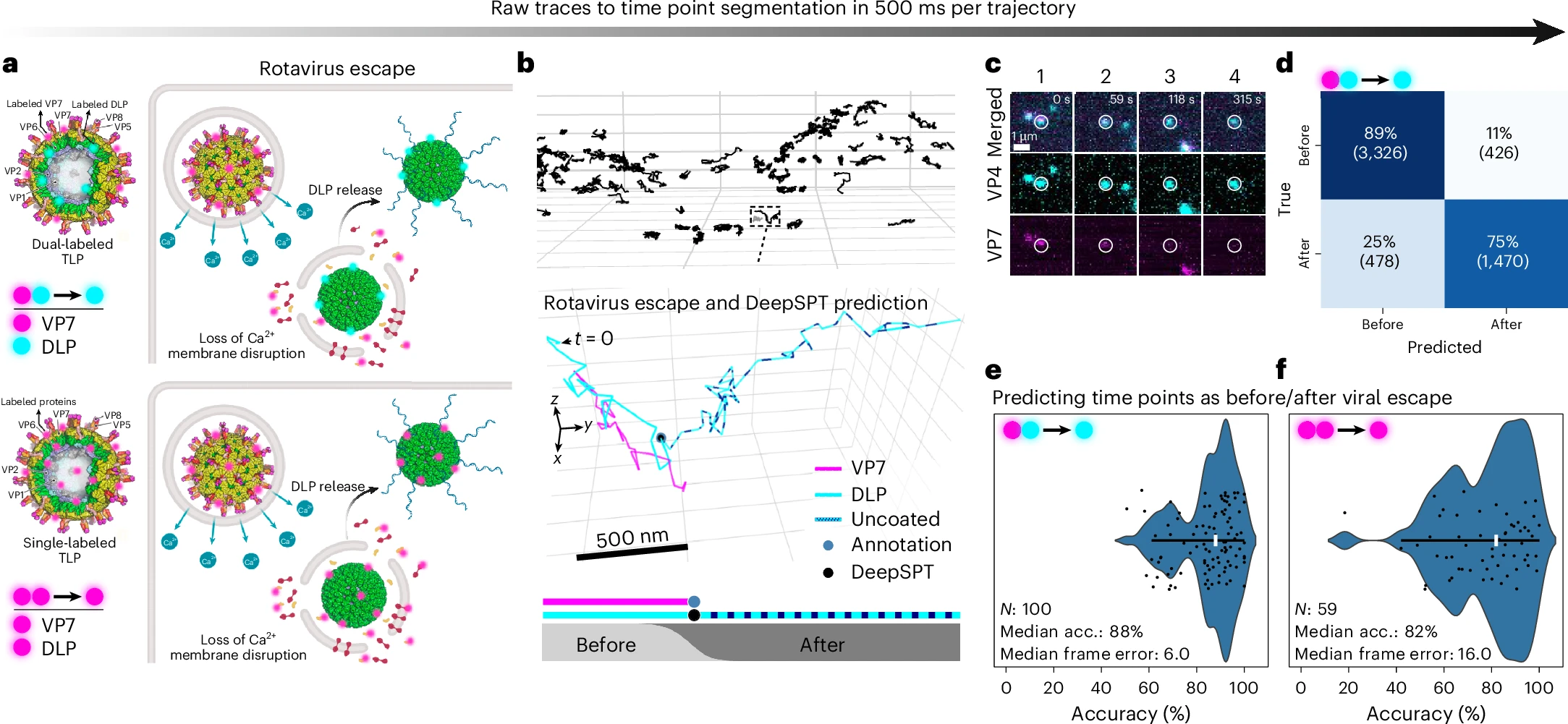
a. 轮状病毒细胞进入通路的典型阶段。从与质膜的相互作用、膜包裹、膜通透化、钙依赖的脱壳到逃逸至细胞质,RNA可以在此开始生产。展示了单个轮状病毒追踪的实验方法。
- 顶部:通过重组构建的带有荧光标记的DLP和VP7的双标记轮状病毒。
- 底部:通过Atto565对自由赖氨酸的单色化学标记。
b. 采用活细胞LLSM获得的单个轮状病毒颗粒的3D轨迹。放大图展示了使用平行多色成像的轮状病毒示例。VP7信号丢失的时间点,表明脱壳和病毒逃逸(蓝点),被DeepSPT(黑点)正确识别。底部的插图显示了DLP和VP7信号的1D表示,标注了VP7的丢失和DeepSPT预测。SoftMax输出提供了‘脱壳前’和‘脱壳后’的时间分辨概率估计。
c. 从放大图中轨迹周围区域的3D活细胞LLSM数据的总强度投影。插图包含DLP(青色)和VP7(品红色)的平行成像。编号的列显示了病毒生命周期中观察到的不同阶段,从共定位到脱壳。
d. 一混淆矩阵展示了DeepSPT分类性能,将时间点预测为‘脱壳前’或‘脱壳后’与实际共定位分析进行比较,条目被归一化至双标记轮状病毒的真实标签(左上,真实脱壳前;右上,假脱壳后;左下,假脱壳前;右下,真实脱壳后)。
e,f. DeepSPT分类准确率的直方图,显示在单个轨迹中准确预测‘脱壳前’或‘脱壳后’的时间点的百分比:双标记轮状病毒显示中位准确率88%(100条轨迹,N = 1的载玻片实验,4部电影)
- (e)单色标记轮状病毒显示中位准确率82%(59条轨迹,N = 5的载玻片实验,13个视频)
- (f)DeepSPT每条轨迹需要500毫秒的处理时间来将轨迹转化为特征表示,并对时间点进行分类(分类约1毫秒),因此与手动标注相比,加速分析至少四个数量级。面板a使用BioRender.com创建。
我们验证了DeepSPT在提取轮状病毒(rotavirus)的3D活细胞SPT数据中的操作有效性(图3a和方法)。轮状病毒进入细胞的过程涉及(图3a):
- 糖脂介导的病毒与细胞膜的结合
- 囊泡吞噬和内化
- 病毒引发的膜通透性增加
- 钙依赖性外部蛋白的脱壳
- 膜的破坏
- 病毒基因组的胞质传递,以便随后进行RNA合成
之前在BSC-1细胞中使用共聚焦成像的SPT结果表明,脱壳步骤与扩散行为的变化相关,暗示运动可能是生物行为的一个潜在标志。
为了测试DeepSPT仅通过运动检测脱壳和细胞质传递的能力,研究者使用3D活细胞晶格光片显微镜(lattice light-sheet microscopy,LLSM)来成像重组轮状病毒的细胞进入过程,该病毒在VP7(一种外壳蛋白)上标记有Atto565,并在双层颗粒(DLP)上标记有Atto647,或者在包括VP7和DLP的整个病毒上标记有Atto565(图3a,b)。通过LLSM捕获的轨迹(图3b)使用广泛使用的粒子跟踪器u-track9输出,并通过DeepSPT的模块进行时间分割和扩散指纹分析,以滚动窗口的方式进行序列化表示。优化的成像条件使得定位误差远低于步长(补充图14)。这些处理后的轨迹随后通过基于序列到序列的模型分类为“去壳前”或“去壳后”,将时间坐标转换为时间解析的预测,作为特定任务的分类器(补充视频1和2)。对于双标记的轮状病毒,脱壳事件的真值是通过确定不同标记的DLP和VP7的共定位程度来确定的。图3b(放大图和c)展示了轮状病毒去壳的代表性例子,以及DeepSPT的一致预测和原始数据的快照。
双层颗粒(Double-Layered Particles, DLPs)
轮状病毒(Rotavirus)结构中的一个重要组成部分。轮状病毒是一种非包膜病毒,具有三层同心蛋白层结构。双层颗粒由病毒的内层和中间层组成:
内层结构:
最内层由120个病毒蛋白2(VP2)组成,排列成12个十聚体,形成一个50-60纳米直径的二十面体核心颗粒。
这个内层包含病毒的基因组,由11段双链RNA(dsRNA)组成。
中间层结构:
内层被260个VP6三聚体包围,形成中间层。
VP6三聚体与内层的VP2紧密接触,共同构成了双层颗粒(DLPs)
晶格光片显微镜(Lattice Light-Sheet Microscopy,LLSM)
一种先进的成像技术,能够在活细胞中进行快速、低光毒性的三维成像。
DeepSPT准确识别了89%的“去壳前”和75%的“去壳后”时间点,在100个双标记轮状病毒轨迹中,平均准确率为85%,中位准确率为88%。这种高准确率转化为仅六帧的中位误差来确定去包壳时间点(图3d,e)。DeepSPT与HMM、滚动均方位移、参考文献13和AnDi挑战赛中的最佳表现方法进行基准测试,显示DeepSPT获得了对轮状病毒逃逸的更准确识别(扩展数据图8和补充图15和16)。此外,可以使用DeepSPT中的启发式方法研究轮状病毒在去包壳前后的行为特征(补充图15)。与传统方法不同,传统方法通常需要几分钟到几小时的手动分析每条轨迹,而DeepSPT自动化了识别过程,将每条病毒轨迹的时间缩短到毫秒级,提供了巨大的加速并最小化任何人为偏见。
DeepSPT基于DLP轨迹中捕获的运动,以每条轨迹500毫秒的速度输出这些预测,从而使得第二个VP7通道多余(补充视频1和2)。在对同一荧光染料标记的轮状病毒进行测试时,DeepSPT显示出相似的表现,获得了82%的中位准确率和78%的平均准确率(图3f)。值得注意的是,与手动标注需要约8个工作日以获取基于560 nm通道强度损失的真实注释相比,DeepSPT只需不到一分钟。通过将运动作为病毒去包膜的标记,DeepSPT简化了实验设计和准备,避免了构建双标记病毒的需要。因此,DeepSPT释放了可用的两个到三个成像通道之一,从而增加了荧光显微镜实验中的信息内容。据我们所知,这些结果构成了首次基于运动检测病毒逃逸到细胞质的实例,而不需要多色标记。
运动中的共定位和细胞定位
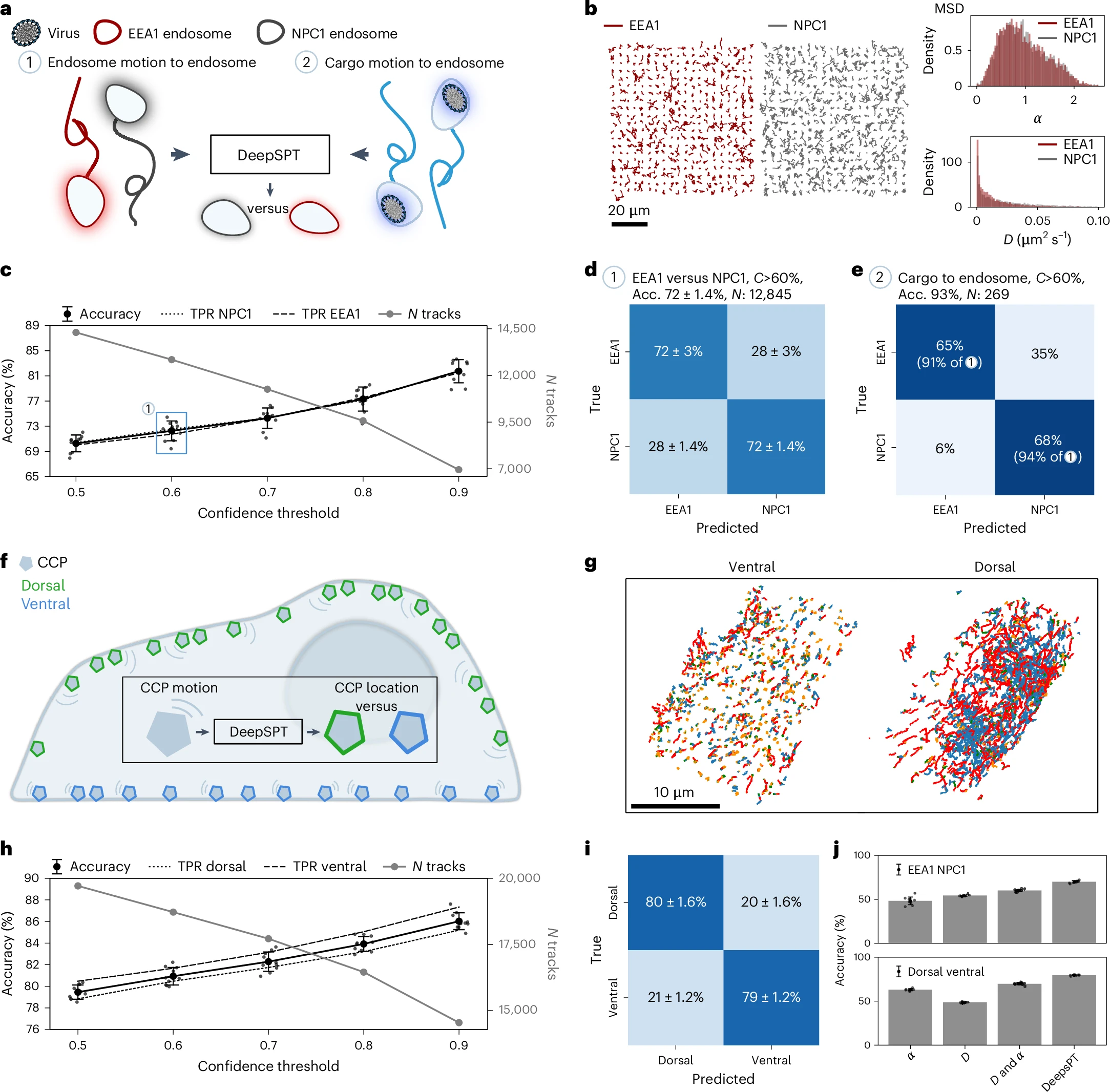
a, 仅使用内涵体或其货物的扩散来预测内涵体身份的示意图。
b, 左:通过LLSM获取的EEA1-mScarlett-(红色)和NPC1-Halo-JFX646阳性(灰色)轨迹的二维投影,显示出视觉上相似的轨迹。右:EEA1阳性(红色)和NPC1阳性(灰色)区域异常扩散指数(上图)和扩散系数(下图)的分布,显示出几乎无法区分的分布(N = 4,770 EEA1阳性和N = 9,534 NPC1阳性)。
c, 左:准确性和真正阳性率(TPR)与DeepSPT分类的置信阈值(方法)的双轴图(右:轨迹数量)。提高阈值增强准确性,但降低接受的轨迹。误差条表示标准差。
d, 在60%阈值下EEA1与NPC1阳性区域的DeepSPT分类的混淆矩阵,显示准确率为72 ± 1.4%(N = 4次实验,35部影片)。误差条表示标准差。
e, 使用轮状病毒货物轨迹的DeepSPT分类EEA1与NPC1阳性区域的混淆矩阵,在60%阈值下使用置信阈值(C)。括号中的准确度相对于d的结果进行了标准化(269轨迹来自N = 12次实验,44部影片)。
f, 仅使用扩散行为来预测AP2复合物细胞定位的DeepSPT示意图。
g, 来自玻片下方和上方500 nm的AP2轨迹的二维投影。超过500 nm以上其寿命的20%的轨迹被认为是背侧的,其余被认为是腹侧的。轨迹由DeepSPT分割进行颜色编码。
h, 关于DeepSPT分类AP2的双轴图,左侧为准确性(TPR)和轨迹数量(右)与置信阈值的关系。误差条表示标准差。
i, DeepSPT对AP2的预测的混淆矩阵,显示在50%置信阈值下准确率为79.5 ± 0.6%(19,712轨迹;12,213背侧和7,499腹侧来自N = 5次实验,13部影片)。误差条表示标准差。
j, 根据MSD的DeepSPT与传统特征的基准:EEA1与NPC1阳性区域和背侧与腹侧AP2的扩散系数(D)和异常扩散指数(α)。误差条表示标准差。DeepSPT显著优于MSD分析(P值<0.00001,使用双侧Welch t检验,N = 10每个条件;补充表3)。
识别生物分子身份、共定位伙伴或仅基于扩散特性推断亚细胞定位的能力,可以减少对多色成像的需求以及与创建表达相关荧光细胞标记的细胞系相关的劳动密集型工作。例如,早期和晚期内体的分化需要多色标记,因为它们可能看起来具有相似的尺寸、以相似的空间密度分布并显示出几乎相同的扩散系数。传统上,它们的识别需要通过针对在特定类型内体中富集的内源性蛋白标记的抗体对每种内体类型进行标记,或者通过异位表达这些标记。基于多色标记,已经通过分析固定样本中内化货物的分布和腔体形态,努力推导内吞机制的一般原则。
在这里,我们评估DeepSPT是否可以仅基于扩散特性判断内涵体的身份,从而减少多色标记的需要(图4a)。我们使用双色活细胞LLSM跟踪由基因编辑内源性标记的早期内涵体(EEA1-mScarlett)和由NPC1-Halo-JFX646标记的晚期内涵体(补充视频3)。它们的轨迹显示出无法区分的扩散系数和α值(图4b及补充图18和19),这对内涵体身份预测提出了挑战。在一个十倍分层交叉验证方案中,随着决策置信度阈值的变化,DeepSPT在将EEA1阳性与NPC1阳性区室分类时的准确度范围为70 ± 1.3%到82 ± 1.8%。提高置信度阈值提高了准确性,但减少了接受的轨迹数量(图4c)。在60%的置信度阈值下,DeepSPT识别内涵体类型的准确率为72 ± 1.4%(图4d),对EEA1阳性区室的召回率为72 ± 3%,对NPC1阳性区室的召回率为72 ± 1.4%(图4d)。DeepSPT明显优于常用的MSD分析,后者在内涵体分类中的准确率为48 ± 4%、55 ± 1.6%和60 ± 1.4%(补充图20和21),使用α值的变异、扩散系数(D)的变异或同时结合α和D的变异。DeepSPT仅利用内涵体货物的扩散特征,在直接预测EEA1阳性和NPC1阳性区室时分别实现了91%和94%的召回率(图4e)。将DeepSPT与文献13、AnDi挑战中的E法(method E)进行基准测试,采用α值的变异、扩散系数(D)的变异或同时结合α和D的方法,仅通过扩散识别内涵体身份,结果表明DeepSPT明显优于所有方法(扩展数据图9)。DeepSPT通过仅基于早期与晚期内涵体的运动或其货物的运动进行区分的能力,加速了数据采集和分析,同时最小化了潜在干扰和/或多色标记的需要。
为了评估DeepSPT是否可以推断其他系统的身份,我们首先将其应用于一个新的数据集,该数据集包含在细胞表面形成的CCP和涂层囊泡的单粒子轨迹的组装。通过使用3D实时LLSM跟踪克拉苏蛋白AP2适配器复合物(基因编辑了其sigma亚基并标记了eGFP),获得了这些结构的动态组装和细胞内位置。获取的AP2轨迹的2D投影定性地表明,AP2的扩散特性与细胞位置相关联55——背面与腹面细胞表面(图4f,g),这一点通过DeepSPT对3D轨迹的时间分段进行了定量证实(补充图19)。在十倍交叉验证方案中,DeepSPT准确预测了AP2的细胞位置,在不同的置信阈值下,结果的准确率在79.5±0.6%到86.0±0.8%之间(图4h)。在未应用任何置信过滤器的情况下(也就是50%的阈值),DeepSPT对AP2的细胞位置进行了分类,其中两个类别的召回率大约为80%(图4i)。相比之下,使用MSD特征定位AP2的细胞位置的准确率达到了62.5±1.8%、48.7±0.8%和70±1.3%,其中背面轨迹的最大召回率为60±2%(图4g和补充图20及21)。尽管参考文献13和AnDi挑战的E方法对AP2的分类优于基于MSD的特征,DeepSPT显然优于所有基准方法(扩展数据图10)。
DeepSPT在识别四个实验数据集中的生物信息时,显示出高于或与基准方法相似的准确性,这些数据集跟踪个体酶(补充图22和23)、转录因子(补充图24)和药物纳米载体(补充图25),突显了DeepSPT在多种系统中的适用性。DeepSPT利用系统间微妙的扩散变化,这些变化常被普通工具忽略,以精确输出复杂系统中的生物信息,同时也允许对每个特征重要性的研究,从而提供机制性洞见(补充图26)。DeepSPT在各种生物背景、成像协议和实验条件下实现了这一点。
讨论
生物分子在细胞内的扩散表现出空间和时间的异质性,并且在不同的生物系统和功能上有所差异。从活细胞成像中提取定量时间信息目前是一个分析瓶颈,通常依赖于特定系统的分析或甚至手动标注。DeepSPT通过提供一个框架,克服了这一瓶颈,能够快速、精确且以最小的人为干预从原始轨迹转变为定量时间信息,无论是针对2D还是3D扩散。DeepSPT的多模块架构利用并超越了我们之前在扩散指纹识别或其他深度学习工作上的成果,结合了分割模块、指纹识别模块以及下游分类模块。分割模块在具有广泛分布的扩散特性的轨迹上训练,始终在对模拟和实验数据中不同异质性扩散行为的分割中优于现有的最先进工具箱。实施的不确定性校准概率估计增强了DeepSPT输出的透明性,使得对生物系统了解有限的用户也能判断模型的确定性。扩散指纹识别模块的特征从17个扩展到40个,确保对扩散指标和时间信息进行更全面的量化,用户可以轻松进行特征选择。根据我们所知,分类模块的实施提供了基于扩散特性单独得出的生物信息的首次输出。
DeepSPT目前将粒子追踪器的输出作为输入,例如u-track9或Trackpy56,这些工具已经考虑了光子闪烁,而DeepSPT并不是为了解决闪烁或追踪丢失错误而设计的。然而,DeepSPT在即使是产生定位误差(大小与平均步长相同)、多个链接错误以及持续时间仅约20帧或更短的轨迹的成像设置下,也展示了强健和准确的预测能力(扩展数据图2-4和补充图7和14)。DeepSPT训练使用了四个数量级的扩散系数和具有高度变化持续时间的行为段,旨在捕获大量的过渡速率,但没有训练以捕获快于成像帧率的状态过渡。DeepSPT的多模态性可以为无监督数据探索提供有价值的见解,提供基于深度学习的预训练扩散分割和针对任何2D或3D轨迹的扩散启发式。同时,利用广泛的特征集,DeepSPT实现了准确且可解释的下游监督学习,并具有特征选择和特征重要性评估的潜力,以确定关键的机制见解。端到端模型在有足够训练数据的情况下,可能会获得类似或改进的预测,尽管这可能会导致训练时间更长以及可解释特征的丧失13。通过例如利用定位形状、运动模糊、定位强度、信号与背景比率或轴特定不确定性,改变DeepSPT架构或网络结构可能会提高性能;DeepSPT的可访问源代码对此非常重要。对人类干预的最小需求凸显了DeepSPT在不同实验室之间增强结论可重复性和稳健性的潜力。开源并免费向公众提供,使未来用户能够根据个人研究需求进行定制分析。
精确的时间分段结合DeepSPT扩散特性的全面量化,加上其训练好的下游分类器,使病毒去壳事件的快速预测成为可能——取得的结果在几秒内,而不是人工注释所需的几周。这种四个数量级的加速,不仅标志着深度学习辅助的病毒去壳识别,也将单颗粒发现中的瓶颈从数据分析转移到了数据采集。它甚至引入了早期病毒感染阶段几乎实时分析的潜力。
在二维或三维中的微妙扩散变化虽然被常见工具忽视,但被DeepSPT利用,以在各种生物背景、成像协议和实验条件下精确输出复杂系统中的生物信息。例如,DeepSPT仅基于其各自的三维扩散特征或其货物,辨别出EEA1阳性和NPC1阳性隔室,准确率为72%,显著超过常用的MSD分析(准确率为50-60%)。这些发现促使进一步的机制研究,以探索不同的扩散行为是否源于不同的外部相互作用伙伴、内源性内涵体隔室之间的物理差异或其他变量。DeepSPT同样在三维数据中准确定位了AP2的细胞位置,准确率为80%,显著超过常见分析的约50-70%准确率。AP2的不同扩散行为突显了成像设置中谨慎选择的重要性。在胰岛素内化的二维数据上,DeepSPT发现胰岛素主要表现出亚扩散行为,但也包含指示主动运输的定向运动片段(补充图12和13)。DeepSPT准确量化二维和三维中异质行为的能力,以及在多样化的生物系统和不同实验及成像条件下的表现,证明了其作为表征系统间异质扩散的平台的实用性。
DeepSPT在预测病毒解包事件、识别内吞体类型以及仅基于扩散区分共定位伙伴和细胞定位方面的能力,将传统的结构与功能范式扩展到一种新颖的运动与功能范式。这表明,除了结构,运动也可以作为功能和身份的指示器。这一发展为采用运动作为生物标志物以及进行最小标签分析开辟了新途径——有效地用时间扩散分析替代荧光标签。这种转变可以简化实验设计,减少准备时间,降低光毒性,或通过将冗余的荧光标记重新分配用于其他应用来潜在地丰富实验。为了简化DeepSPT在不同实验室的实施,并将我们的用户群扩展至更广泛的受众,我们提供了一个独立的直观GUI,允许用户执行DeepSPT的所有功能,包括输出分析数据、扩散特征、分割与分类以及生成出版质量的图形。该GUI还允许用户方便地单独使用任何模块,或者将它们结合使用。互动式的内置轨迹查看器允许对轨迹和扩散分割进行2D和3D可视化。开源实现使专业用户能够针对其特定需求优化和增强管道。DeepSPT在实验室的广泛实施可以促进创建详尽的库,详细记录细胞、亚细胞结构和生物分子的特征运动。这种开放源代码的扩散库将为科学界提供一种新的工具,帮助探索通过时间扩散行为进行的4D细胞生物学。
方法
DeepSPT的扩散指纹模块
包含并扩展了参考文献13中的工作,最近的一项研究提供了一组扩散指标,以将单颗粒轨迹转化为固定长度的可解释特征表示,这些特征作为唯一识别符,即扩散指纹。这项工作将扩散行为的描述特征从17个扩展到40个,更重要的是,提供了时间特征和顺序表示,以实现时间分辨的预测(补充表3)。后面的章节对此进行了概述。
限制半径和定向速度
由于DeepSPT允许准确的扩散行为分段,因此有可能使用为特定运动类型调整的MSD方程,例如![]() ,其中r是限制半径,A1和A2是形状参数,d是维度数,D是扩散系数,t是时间,以及
,其中r是限制半径,A1和A2是形状参数,d是维度数,D是扩散系数,t是时间,以及![]() 用于限制和定向速度(v)。MSD分析中的偏移对应于来自定位误差的MSD恒定贡献。因此,可以提取整个轨迹或子轨迹的定向运动的速度项和限制运动的限制半径。
用于限制和定向速度(v)。MSD分析中的偏移对应于来自定位误差的MSD恒定贡献。因此,可以提取整个轨迹或子轨迹的定向运动的速度项和限制运动的限制半径。
使用向量点积(DPs)进行方向性分析
两个向量之间的点积(dot product,DP)能够反映它们之间的角度和它们的大小乘积,特别是对于归一化的向量,DP返回角度的余弦值(−1, 1),其中平行向量的值为1,正交的为0,反平行的为−1。对于给定的轨迹,由三组连续坐标(p)在轨迹中的时刻(i)形成的两个向量(和
);p0,p1,p2可用于计算沿轨迹的滚动DP,以研究方向性的持久性。由于进行滚动DP计算需要使用三组连续坐标构建两个向量,因此得到的滚动DP向量比原始轨迹短两个元素。为了使滚动DP向量的长度与原始轨迹匹配,DP向量的开头填充了两个零。使用三种策略将每帧的DP序列聚合成一个值,以补充扩散指纹分析:
- 平均(MeanDP)用于显示方向性中的任何平均趋势,正常扩散的结果一般接近于0,方向扩散大于0,亚扩散小于0;
- 持续性(persistDP)用于研究连续向量通常是否具有方向持久性,具体计算是连续出现的正或负的比例;
- 符号分析(AvgSignDP)计算向量DP符号为正的百分比。
附加步长描述统计
步长包含了关于单个粒子轨迹的许多信息,这里我们增加了额外的描述统计,例如最小步长(MinSL)、最大步长(MaxSL)、步长分布的广度(BroadSL),即 MaxSL - MinSL;以及步长分布的变异系数,即标准差与均值的比率,用于测量分布相对于均值的变异性。被捕获的分数和快速移动的分数是特定于系统的,但分别计算为步长在 0.1 微米以下和在 0.4 微米以上的百分比。瞬时扩散系数的计算是使用相邻位置之间的均方位移(MSD),即 MSD/2dt。
轨迹的体积/面积
2D或3D轨迹的面积或体积(为了一致性,称为体积)是封闭轨迹的凸包的体积,其中2D或3D的坐标为x、y、(z)。轨迹或子轨迹的体积直接反映了轨迹的形状,并包含了轨迹所探索的体积量的信息,以及所探索区域的形态特征。因此,体积可用于识别限制运动的粒子与更自由运动的粒子,并提供一个用于限制体积的度量,并使用Python包SciPy进行计算。
时间特征
为了向分类器提供轨迹中的时间变化信息,时间分段被压缩为独特的特征。四个特征是基于花费在正常扩散、定向运动、受限扩散或亚扩散中的时间百分比构建的,以及一个基于扩散变化次数的特征。为了反映扩散变化的历史,扩散行为的序列被编码为:正常扩散,0;定向运动,1;受限扩散,4;亚扩散,6;例如,0146表示从正常扩散开始每种行为采样一次。编码值的选择是故意的,旨在将相似的运动类型编码为相似的数值,同时在每种运动类型的数值之间编码独特的距离。根据这种编码构建了六个特征:平均值和中位数用于表示最可能的运动类型,最大值和最小值用于表示采样到的运动,标准差和相邻序列值之间的距离中位数用于反映采样运动类型的变化和相似性。
DeepSPT 的时间分割模块
DeepSPT 中的时间分割模块由三种 U-Net 组成,提供从原始轨迹到轨迹分割的端到端转换。我们选择 U-Net 是基于我们的假设,认为它们对局部感受野的关注、共享特征图、分层特征组合以及在归纳偏见中具有平移不变性,使其特别适合于建模扩散的特性。每个 U-Net 模型在 300,000 条模拟轨迹的数据集上进行训练,该数据集的详细信息见“扩散的随机模拟”和“生成异质扩散”部分,其中 80% 为异质运动,20% 为同质运动。
著名的 U-Net 的端到端架构可以看作是编码器网络的降采样、解码器网络的升采样以及随后的分类器。各个 U-Net 包含1D 卷积层和最大池化的编码网络,以及 1D 卷积层和最近邻升采样的解码网络,编码器和解码器通过跳过连接相连。编码和解码后直接跟随一系列卷积层,然后是一个集成平均 SoftMax 输出。集成中每个模型的 SoftMax 输出通过平均组合以产生最终的 DeepSPT 预测。特定的超参数由 Optuna 的树结构 Parzen 估计器找到,并根据仅在超参数搜索中使用的测试集上的表现选择最佳超参数集(未包括集成大小)。对于 2D 时间分割,所选超参数包括四个降采样步骤和四个升采样步骤。编码器包括两个层,解码器包括一个层,具有三个底层和四个输出层。编码器、底层和解码器的卷积核大小为 7,扩张率为 2,而输出层使用的卷积核大小为 3,无扩张。在输入特征之后的通道维度设置为 130,在降采样期间应用 2 的乘法因子,在升采样期间应用除法。对于 3D 时间分割,超参数包括三个降采样步骤和三个升采样步骤。编码器由三个层组成,解码器由四个层组成,具有四个底层和两个输出层。编码器、底层和解码器的卷积核大小为 5,扩张率为 2,而输出层使用的卷积核大小为 3,无扩张。在输入特征之后的通道维度设置为 48,在降采样期间应用 2 的乘法因子,在升采样期间应用除法。该模型采用 Pytorch 编写。
DeepSPT模块的训练和评估概述
DeepSPT时间分割模块(M1)的训练数据由三个独立的模拟扩散数据集组成,每个数据集包含300,000条轨迹,其中80%为异质扩散,20%为均质扩散。这些轨迹的构建在‘扩散的随机模拟’和‘生成异质扩散’部分中进行了描述。M1的训练使用Adam优化器和交叉熵损失函数。M1的准确性评估使用保留的测试集进行。DeepSPT扩散指纹模块(M2)是一套广泛的扩散启发式方法,直接从任何数据中得出,因此不需要训练。扩散指纹特征的描述能力通过分类性能和特征重要性评估进行评估。DeepSPT任务特定分类器(M3)模块的训练数据是任务特定的,训练直接在实验数据上进行,评估通过交叉验证。
DeepSPT 在 2021 年 AnDi 挑战数据的时间分割模块
DeepSPT 在为 AnDi 挑战数据重新训练时,其原理和架构与‘DeepSPT 的时间分割模块’部分中解释的相同。每个 U-Net 模型都是在 400,000 个模拟轨迹的数据集上进行训练的,如‘用于 AnDi 挑战数据的时间分割评估的模拟测试集’部分所述,涵盖了 AnDi 任务 3 和高度异构的轨迹。同样,特定的超参数是通过 Optuna 的树结构 Parzen 估计器从一个独立的验证集中找到的。
温度缩放神经网络
被发现即使在错误预测中也过于自信。这种过度自信可以通过不确定性校准来缓解,使得置信度估计更接近实际的正确性概率。温度缩放就是这样一种方法,尽管其简单性,仍然被发现有效。温度缩放在分类器的SoftMax之前的最后一层引入一个常数,并将该常数调节以最小化负对数似然,因为近期的研究表明这使得能够近似实际的后验概率分布。不确定性校准的衡量指标包括预期校准误差,这实际上是完美校准和实际校准之间的一范数误差;尖锐度,测量k个类别的最大类别概率分数与1之间的距离,因为完美分类器对正确预测的类别概率分数为1;最后,负对数似然(NLL),这里NLL相对于随机预测被报告为NLL改善,以便提供更直观的指标。
随机模拟扩散
轨迹是通过在0.0001和0.5 µm² s⁻¹之间对随机扩散系数进行对数均匀采样生成的。由于扩散的尺度不变性,D的广谱采样等同于以不同大小的时间步长(t)进行采样,从而允许模型学习不同D和t的扩散行为特征。模拟轨迹从x = y = 0开始生成,长度均匀抽取在5帧和600帧之间,挑战模型甚至在较短的轨迹中捕捉规律。按Pinholt等人的研究模拟了正常、定向和亚扩散,采用的不同参数与Wagner等人、Pinholt等人和Kowalek等人的研究相似。除了三个参数,使得这些参数的分布更加广泛:α,一个测量运动保持性的异常指数项,范围为0-0.7;步骤长度与定位误差比定义为![]() 和
和![]() ,
,和
以均匀方式在 1 到 16 之间采样;最后,主动运动对扩散的影响程度
![]() 在5到25之间均匀生成。限制运动与以往的工作也有不同,因为本研究中的限制半径与轨道持续时间无关,反映了这样一种情况:反射边界区域的半径在实验进行过程中不会增长。相反,我们模拟了与轨道的持久性无关的限制。限制的面积或体积在二维中由椭圆定义,在三维中由椭球定义,允许任何方向,长半轴和短半轴均匀采样在5纳米到250纳米之间。对于三维情况,两个短半轴被选择为等长,从而在任何给定方向上产生一个大范围的限制面积/体积。
在5到25之间均匀生成。限制运动与以往的工作也有不同,因为本研究中的限制半径与轨道持续时间无关,反映了这样一种情况:反射边界区域的半径在实验进行过程中不会增长。相反,我们模拟了与轨道的持久性无关的限制。限制的面积或体积在二维中由椭圆定义,在三维中由椭球定义,允许任何方向,长半轴和短半轴均匀采样在5纳米到250纳米之间。对于三维情况,两个短半轴被选择为等长,从而在任何给定方向上产生一个大范围的限制面积/体积。
生成具有异质扩散行为的异质扩散
轨迹被模拟为均匀轨迹,通过在扩散行为中添加随机变化点的采样,最多达到四种状态。因此,给定的轨迹被分隔成随机的子轨迹,其所需的最小长度为五帧,因此,轨迹的长度必须大于变化点和最小长度的乘积。在轨迹内部随机采样变化点是故意选择的,而不是让状态遵循用户定义的马尔可夫模型,以确保DeepSPT的决策不会受到学习一个在自然界中不一定存在的潜在马尔可夫模型的影响,而是保持DeepSPT完全不受影响。
优化基准LSTM性能
为了提高用于基准测试的基于注意力的LSTM的性能,LSTM使用原始出版物29的超参数进行重新训练,并在用于训练DeepSPT中的三个个体U-net之一的300,000个模拟轨迹上进行重新训练。
将模拟扩散移动到三维
之前很多人的工作完全集中在二维扩散上,而鉴于我们的3D活细胞晶格光片实验,我们需要将之前的工作扩展到三维。模拟正常扩散和亚扩散运动很容易扩展到二维和三维情况,因为扩散轴是独立的。检测运动是使用余弦和正弦进行模拟,以确保在x和y方向上的方向性,而速度作为一个幅度项,我们通过考虑单位球体将其扩展到三维情况,因此添加的速度变为,
和
,其中θ是极角,ϕ是方位角。
具有重叠分布的异质扩散的两个种群的模拟
两组500个轨迹的种群,每条轨迹的持续时间在150到200个时间点之间均匀抽样,使用上述的四种扩散类型的异质运动模拟框架进行构建。两个群体的步长与定位误差的比率均匀抽样在6到16之间。群体1的活动运动比率均匀抽样在5到12之间,并且亚扩散运动的α在0.3到0.6之间均匀抽样。群体2的活动运动比率均匀抽样在8到15之间,亚扩散运动的α在0.4到0.7之间均匀抽样。除此之外,在扩散系数的增量方面,这两个群体的构建是相同的。扩散系数在0.004 µm² s⁻¹和0.0008 µm² s⁻¹之间以对数均匀分布进行抽样,增量为0.005 µm² s⁻¹。每次随机轨迹模拟后,计算瞬时扩散系数的分布,并计算两个分布的重叠程度作为一人口中的直方图交集归一化后的总轨迹。为了建立具有真值的扩散变化点预测测试集,将这两个模拟人口的轨迹合并为5000条具有单个变化点的轨迹。这些轨迹可以同时抽样两种行为,可以从任一人口开始,变化点随机分布,但距离轨迹的任一端至少要有五个时间点。选定合并的两个群体在扩散系数上约有75%的重叠。
用于评估 AnDi 挑战数据的时间分割的模拟测试集
两个测试集是基于2021年AnDi挑战构建的。首先,使用2021年AnDi挑战任务3的开源框架直接模拟轨迹,该框架构建了持续时间为200个时间点的轨迹,每个轨迹由在AnDi挑战中随机选择的五种扩散行为中的两段异常扩散组成。这五种扩散行为包括退火瞬态时间运动、连续时间随机游走、分数布朗运动、莱维游走和缩放布朗运动。其次,轨迹通过将AnDi挑战开源框架中的异常扩散行为组合成异质轨迹来模拟,这些轨迹样本具有多个扩散行为和多个变化点。具体来说,对于每条200个时间点的轨迹:
- 通过在3到6之间均匀抽样选择段的随机数量;
- 每段的长度在5到200之间均匀抽样,同时确保段的持续时间总和为200;
- 每段均匀抽样五种扩散行为之一,同时确保相邻的段不能表达相同的行为,因为重新抽样行为类型会生成更少、持续时间更长的段,从而简化分析;
- 对于每种运动类型,使用AnDi挑战开源框架生成轨迹;
- 根据Muñoz-Gil等人的研究,标准化轨迹以确保时间上位移的单位标准差,然后按从单位正态分布中抽取的随机因子进行缩放;
- 在每个时间点独立地通过从正态分布中随机抽样的加成进行位移,以模拟定位误差。该正态分布的均值为零,标准差对应于每个维度(x、y和z)中平均步长的50%。
DeepSPT-时间分割中的跟踪误差影响
通过模拟1000条轨迹在不同尺寸的盒子内进行200帧的广泛分布的扩散参数,以评估追踪误差对DeepSPT时间分割精度的影响,并使用Trackpy进行追踪。该人群的扩散特性如“扩散的随机模拟”部分所定义,最大帧数为200帧,扩散系数范围为0.01至0.05 µm² s⁻¹。轨迹的初始位置在盒子内随机抽样(有放回)。任何离开盒子的轨迹将被移除。此外,还有10,000个不属于任何轨迹的定位随机分布在盒子内并均匀分布在帧中,每帧平均产生50个假阳性检测。这些轨迹和假阳性随后被视为单独检测的定位,并使用Trackpy进行追踪,搜索范围为1.5 µm且无记忆。盒子的尺寸选择为20000、2000、1000、500、200、150和100 µm,随着盒子尺寸的减小,追踪误差增加。每个维度进行三次模拟,每次都有一组独立的1000条轨迹。
基于时间分辨的任务特定下游分类器,使用时间和扩散特征
对轮状病毒轨迹进行的分割分为“去包膜前”和“去包膜后”阶段,使用的是基于时间分辨扩散特征的序列到序列模型,该特征是通过Windows中的时间分段和扩散指纹模块计算得出的。原始轨迹可以视为每个时间点有三个并行通道(xyz)的时间序列;这些被转换为具有相同长度但现在有40个通道(每个时间点在时间分段和扩散指纹模块中的一个特征)的时间序列,采用以给定轨迹中的每个时间点为中心的31帧窗口。双标签轮状病毒的去包膜时间点的基准真相是基于VP4和VP7之间共定位的丧失构建的。对于单标签轮状病毒,标签基于VP7信号丧失后强度下降的终点的手动注释(补充图17)。这两种情况产生二元目标时间序列,如果共定位的丧失发生在第一帧或最后一帧,则会被过滤掉。序列到序列模型架构由一个双向的五层门控递归单元组成,后面跟随一个全连接前馈层。由于其双向性,门控递归单元的输出长度是输入长度的两倍,因此在完全连接层之前将其对半分开并通过求和组合以匹配输入长度。在训练之前,通过均方根距离评估轨迹的相似性,均方根距离小于0.6微米的轨迹在图中被归类为连接组件。模型训练使用十折分组交叉验证(验证和测试集各占10%)进行,以确保相似轨迹在同一折中,同时保存在验证集上具有最高平均召回率的模型。
仅基于扩散特征进行预测的特定任务下游分类器
在所有情况下,分类器接收固定长度的轨迹表示,并为每个类别输出一个概率估计。固定长度的表示是通过使用时间行为分割和扩散指纹模块从原始轨迹构建的,共有40个描述性扩散特征(方法)。对输出的概率估计进行过滤是通过要求估计的概率大于给定的阈值,否则轨迹被视为被预测为‘未知’。两个模拟人群的预测(图2e)是使用Scikit-learn61中的简单逻辑回归模型,使用‘lbfgs’作为求解器,允许的迭代次数为10,000,并在五折分层交叉验证中进行评估,测试集大小为10%。EEA1阳性和NPC1阳性区室的预测(图4)是使用简单的多层感知器模型进行的,该模型由输入层(大小40)和输出层(大小2)组成,并使用SoftMax激活。训练采用对少数类的随机过采样,以减轻多数类偏差,并在十折分层交叉验证中进行评估。使用病毒载荷对EEA1阳性和NPC1阳性区室(图4)的预测是使用相同的多层感知器模型,在90%/10%的训练/验证分割下进行训练,并进行少数类过采样,保存具有最高验证准确度的模型。AP2的细胞定位预测(图4)与EEA1阳性和NPC1阳性区室的预测完全相同。
时间上持久的基于距离的共定位分析
共定位是基于在获取通道中轨迹之间的时间一致性接近度而定义的。对于每个成像通道中的感兴趣轨迹,计算与感兴趣的次要通道中轨迹的锁步欧几里得距离。需要在用户定义的搜索距离阈值内连续帧的最小数量,以被定义为共定位段。为了减少在两个轨迹之间的距离闪烁作弊峰值,这些峰值会打断真实的共定位段,因此允许在给定的距离阈值上有一定数量的帧(称为“宽容”),在两侧的帧和共定位段将被连接。为了同时增加已注册共定位段的确定性并减少注册暂时的、虚假的共定位,在‘distthreshold’、‘min_coloc’和‘foregiveness’的基础上添加了多个过滤器:强制执行最低总共定位长度、最低平均同步欧几里得距离和个别坐标轴之间的最低皮尔逊相关性。通过设置连续帧的最低数量为5、搜索距离阈值为750纳米以适应任何通道间的像差、宽容值为5帧、皮尔逊相关性阈值为0.8、最低共定位长度为5帧及最低平均同步欧几里得距离为750纳米,识别共定位轮状病毒和内涵体。初步通过识别长时间共定位来校正色差,然后计算VP4和VP7信号之间的xyz色差,并将所有VP7轨迹根据其平均xyz偏移进行调整。初始参数为连续帧的最低数量为5、搜索距离阈值为750纳米、宽容值为3帧、皮尔逊相关性阈值为0.9、最低共定位长度为20帧以及最低平均同步欧几里得距离为500纳米。在校正色差后,使用连续帧的最低数量为3、搜索距离阈值为400纳米、宽容值为2帧、皮尔逊相关性阈值为0.9、最低共定位长度为20帧和最低平均同步欧几里得距离为600纳米来执行轮状病毒VP4与VP7信号的共定位。
通过3D平面拟合推断AP2相对于盖玻片的位置
在三维空间中,LLSM获得了AP2坐标。这些坐标围绕LLSM成像方向的y轴旋转了30°,以考虑LLSM的检测角度,使用标准旋转矩阵M = ((cos(θ), 0, sin(θ)), (0, 1, 0), (−sin(θ), 0, cos(θ))),其中θ为弧度制的旋转角度。旋转后的坐标指向细胞的腹侧,z轴方向为正。使用AP2一般定位于质膜,旋转后的xy坐标以5微米的网格大小进行分箱(所有箱子左侧包含),对于每个箱子,提取最低的z坐标,代表最背侧的AP2坐标。为了处理背侧z坐标中的离群值,计算每个背侧z坐标的马哈拉诺比斯距离(使用所有背侧z坐标的均值和协方差),过滤掉距离为1.8或以上的坐标。得到的背侧z坐标用于通过最小化背侧z坐标与平面之间的平方距离和来拟合三维平面的参数,最终推断得出载玻片位置。所有AP2坐标都计算了到结果平面的距离。
统计测试
图2e和图4i中结果的比较是通过双侧Welch t检验进行的,以评估零假设,即相关条件的均值相等。选择Welch t检验是因为它作为位置检验的优势,以及它对具有不等方差和样本大小的群体的稳健性。Welch t检验是在SciPy中实现的,使用Welch-Satterthwaite方程计算自由度(补充表1和表2)。
Welch 的 T 检验
一种统计方法,用于确定方差和样本量可能不相等的两个组的平均值之间是否存在显著差异。Welch 的 T 检验适用于被比较的两组样本量和方差不同的情况。例如,如果一组样本量小且变异性高,而另一组样本量大且变异性低,则使用 Welch 的 T 检验可以更准确地分析平均值。这种检验在处理通常不符合其他统计检验所要求的严格假设的现实世界数据时特别有用。
用于 LLSM 成像的病毒和内涵体标记
用mScarlett(EEA1-mScarlett)编辑基因的早期内体抗原1的细胞和用JFX646标记的胆固醇转运蛋白Niemann Pick C1的激活版本(NPC1-Halo-JFX646)是来自Kang等人在Kirchhausen实验室生成的冷冻样本。细胞最初来自美国典型培养物保藏中心(CRL-8621)。具有克拉胜适配器复合物、在其σ亚单位上编辑过的AP2基因与eGFP的细胞是来自Cocucci等人的冷冻样本。为了成像,SVG-A细胞在每次实验前一天在35 mm培养皿内的直径为5 mm的载玻片上以约50%的汇合度铺板。细胞用10 µl的标记轮状病毒颗粒以约40 µg ml−1的浓度和约10的感染倍数孵育10分钟,然后直接转移到显微镜下。细胞在无酚红的培养基(FluoroBrite DMEM、25 mM HEPES和1% PenStrip)中成像,培养基中加入了可溶性荧光染料(或Alexa Fluor647或Alexa Fluor488羧酸)。在没有轮状病毒的实验中,使用FluoroBrite DMEM、25 mM HEPES和1% PenStrip以及5% FBS。为了对病毒进行标记,三层颗粒(TLPs)在总容量为50 µl的HNC(20 mM HEPES pH 8.0、100 mM NaCl和1 mM CaCl2)中稀释至0.4 mg ml−1,并加入5.5 µl的1 M NaHCO3(pH 8.3)。将此溶液与0.5 µl的0.76 mg ml−1 Atto488 NHS酯混合。反应在室温下进行1小时,然后用5 µl的1 M Tris pH 8.0终止。标记的TLPs然后使用Zeba旋转脱盐柱交换缓冲液,转移至含有20 mM Tris pH 8.0、100 mM NaCl和1 mM CaCl2的溶液中。对于再涂层颗粒的形成和标记,提纯了TLPs、DLPs、VP7和VP4。VP7和VP4在感染了杆状病毒载体的Sf9细胞中表达。VP7通过在甘露糖凝集素A和单克隆抗体(mAb159)上进行的连续亲和色谱法进行纯化(用EDTA洗脱)。纯化的VP7被去盐处理,放入含有2 mM HEPES(pH 7.5)、10 mM NaCl和0.1 mM CaCl2(0.1 HNC)的溶液中。对于VP4,收集的细胞通过冻融裂解并在加入完全不含EDTA的蛋白酶抑制剂(罗氏)后,离心在2,900g下澄清。VP4通过加入30%饱和度的硫酸铵沉淀,沉淀后重悬于含有20 mM Tris(pH 8.0)和1 mM EDTA的溶液中,然后加载到HiTrap Q柱(GE Healthcare)上,并在10至150 mM NaCl的梯度中洗脱。含有VP4的汇聚组分在20 mM Tris(pH 8.0)、100 mM NaCl和1 mM EDTA缓冲液中透析过夜。
如前所述,VP7和DLP被标记。VP7的浓度提高到1.7 mg/ml,总体积为100 µl,加入了11.1 µl的1 M NaHCO3(pH 8.3)。该溶液与0.76 mg/ml的Atto565-NHS酯的0.71 µl混合。反应在室温下进行1小时,然后用10 µl的1 M Tris(pH 8.0)终止。标记后的VP7被脱盐,转移到一个含有2 mM Tris(pH 8.0)、10 mM NaCl和0.1 mM CaCl2(0.1 TNC)的溶液中。接着,将50 µg DLP在HN中稀释到100 µl,再加入11.1 µl的1 M NaHCO3(pH 8.3)。此溶液再与0.5 mg/ml的Atto647N-NHS酯的1.5 µl混合。反应在室温下进行1小时,然后用10 µl的1 M Tris(pH 8.0)终止。样品通过0.5 ml的Zeba Spin脱盐柱脱盐,转移到一个含有20 mM Tris(pH 8.0)和100 mM NaCl(TN)的溶液中。我们将45 µg的DLP平均分配到五个1.5 ml的锥形管中(每管0.5 µl)。首先向最终浓度为100 mM的溶液中加入1 M醋酸钠(pH 5.2),然后加入82 µl的VP4(浓度为1.8 mg/ml),使最终反应体积内VP4的浓度为0.9 mg/ml,从而使VP4单体的数量超过DLP上的总180个位点33倍。向样品中添加了0.1 mg/ml的aprotinin溶液,使最终浓度达到0.2 µg/ml,然后在37 °C下孵育1小时。为了使VP7单体的浓度超过DLP上780个位点的2.3倍,我们将所需量的VP7(7.14 µl,储存于1.26 mg/ml的HNE中)与0.1体积的TC缓冲液(20 mM Tris,10 mM CaCl2,pH 8.0)和0.1体积的1 M醋酸钠(pH 5.2)预混合15分钟,再加入到DLP-VP4混合物中。样品在室温下孵育1小时,然后通过添加0.1体积的1 M Tris pH 8.0终止。五个管中的再涂覆颗粒被合并,并添加TNC至最终体积2.5 ml。重组TLP(rcTLP)通过在4 °C下用Beckman Coulter旋转器TLS 55以215,000g超离心,从多余的VP4和VP7中分离出来。我们去除2.0 ml的上清液,用TNC补充至2.5 ml并再次沉淀。小心去除上清液,留有100-200 µl。rcTLP沉淀重悬于剩余的缓冲液中,并存储在4 °C。
胰岛素标记用于SDCM成像
HI被标记为Atto-655-NHS酯。HI Atto-655-NHS (LysB29Atto-655-HI) 酯是根据以前的出版物准备的。简而言之,HI(21 mg,0.0036 mmol,3当量)溶解在pH调节至10.5的0.1 M Tris缓冲液(0.2 ml)中以实现完全溶解,随后在5分钟内逐滴加入DMF(0.3 ml)中的Atto 655-NHS酯(1.0 mg,0.00122 mmol,1.0当量),然后搅拌15分钟。反应通过液相色谱-质谱联用技术进行监测。随后,反应混合物用2.0毫升的Milli-Q水稀释,并将pH调节至7.8。该产品通过反相高效闪光色谱分离,使用Biotage SNAP超柱(C18,30克,25微米)。以5–50% CH3CN线性梯度在20分钟内以25 ml/min的流速使用与0.1%甲酸混合的CH3CN/H2O作为洗脱剂。每个分馏通过液相色谱-质谱联用技术进行分析。单取代产品单独收集,CH3CN在减压下利用旋转蒸发器去除,随后进行冷冻干燥(LysB29Atto-655-HI收率:5.6 mg,79%)。
当量(equiv)
通常用于描述物质在某种特定条件下的等效数量或性能。在不同的学科领域中,例如化学、物理、工程等,对当量的定义和应用有所不同。但总体来说,当量是一种为了方便计算或比较而设定的量化标准。在化学领域,当量常用来表示反应物或生成物的数量,尤其是与化学反应的计量关系有关时。例如,在化学反应中,某些物质以特定的比例参与反应,这些物质的当量就是它们在反应中的计量单位。
LLSM成像,实验设置和SPT
轮状病毒,EEA1-mScarlett。NPC1-Halo-JFX646和AP2跟踪实验使用自制的LLSM进行,方法与之前发表的工作相同。LLSM成像在样品扫描模式下进行,每个平面在z成像轴上的间距为0.25微米,生成由抖动的多贝塞尔光片照明组成的3D体积的分子视频。每个实验的曝光时间和帧速率在补充表4中列出。生成的z堆栈经过去斜处理,随后通过探测和连接点光源的光点,在MATLAB中使用自动跟踪算法产生xyzt轨迹集,该算法基于u-track,涉及3D高斯的最小二乘数值拟合,前面已描述。使用的设置与u-track的原始设置相同,唯一区别在于搜索半径下限设定为3个像素,上限设定为6个像素。为了评估LLSM中的定位误差,我们生成了模仿实验PSF(点扩散函数)在x、y和z维度的模拟PSF。这是通过对在光栅中测量的实验珠子拟合高斯函数来实现的,使用的样品扫描设置与收集实验数据时相同。模拟的PSF与背景噪声叠加,背景噪声与实验中观察到的相同,生成了100帧具有不同背景强度的z堆栈,以重现真实的噪声波动。该过程对五种不同的PSF强度重复进行。最后,使用点源检测方法分析模拟数据,并与已知的真实值进行比较。
SDCM成像和SPT
使用反向旋转盘共聚焦显微镜(SDCM)(奥林巴斯 SpinSR10,奥林巴斯)进行所有胰岛素的2D成像。SDCM使用油浸60×物镜(奥林巴斯)和数值孔径为1.4,连接到补充金属氧化物半导体(CMOS)相机(Photometrics PRIME 95B),有效像素大小为183纳米 × 183纳米。在成像之前,约10,000–20,000个HeLa细胞在37°C,5% CO2条件下在Ibidi IbiTreat 8孔板中培养2天。使用商业库存浓度稀释至1:20,000的LysoTracker Green DND-26被添加并在成像之前孵育1小时(37°C,5% CO2)。最后,为了成像胰岛素,向HeLa细胞中添加0.05 mg ml−1的LysB29Atto-655-HI进行1小时的孵育(37°C,5% CO2)。在任何SDCM成像实验之前,孔被用新鲜的预热10 mM HEPES和HBSS缓冲液清洗三次。为了同时对胰岛素和细胞区室进行SPT,使用640 nm和488 nm的激光进行了双重成像。SDCM实时细胞成像是在SDCM流媒体设置下以30.4 ms的曝光时间和3 EM增益进行的,结果是帧之间的间隔为36 ms,包括滞后时间。胰岛素以100%的激光功率在640 nm的激光下记录,细胞区室以10%的激光功率记录LysoTracker Green DND-26,在37°C下总共记录了2,000帧。使用基于Trackpy的内部跟踪脚本6进行LysB29Atto-655-HI的跟踪,物体直径为9个像素,搜索范围为5个像素,间隔关闭为1帧,平均乘数手动评估在0.6和1之间。轨迹的后处理使用偏心率(ecc)<0.3、强度>0和持续时间>20的阈值,以及训练用于根据跟踪脚本直接检测特征来区分视频中有/没有胰岛素的检测的逻辑回归模型,进一步过滤检测。为了评估SDCM中的定位误差,使用Crocker–Grier算法分析了一系列100幅荧光显微镜图像,每幅图像捕捉衍射极限颗粒。对于随时间检测到的每个颗粒,使用从Crocker–Grier算法获得的质心位置作为初始猜测,对PSF进行2D高斯函数拟合。拟合在10 × 10像素的感兴趣区域内进行。标准误差或均值定位误差表示定位误差,并受到信噪比的影响。在假设一个全局噪声项的情况下,该误差直接依赖于背景减去的颗粒信号的亮度。因此,定位误差在颗粒之间并不均匀,应该相对于颗粒亮度考虑。为此,我们将颗粒分类为亮度分位数,并报告整体定位误差以及每个分位数内的误差。
数据可用性
所有数据均可通过哥本哈根大学的存储库获取。




















 2317
2317

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








