LD(Local descriptor)是一种无需序列比对的方法,其效力在很大程度上取决于潜在的氨基酸分类。
1 划分区域
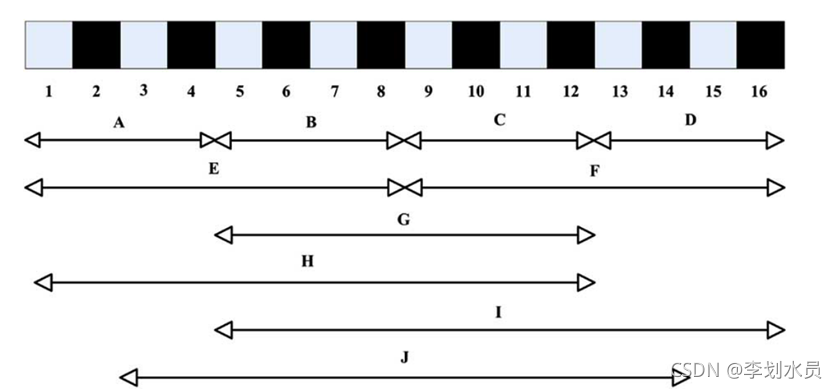
首先将每个蛋白质都会划分成10个区域,通过计算10个区域的 组成(Composition, C) 过渡(Transition, T) 分布(Distribution, D) 构建蛋白质序列特征向量。
10个区域的划分分别是4均等分(A-D),2均等分(E-F),中间50%(G),前75%(H),后75%(I),中间75%(J)
2 分类
依据氨基酸侧链的偶极性和体积,把20种氨基酸分为7个组。
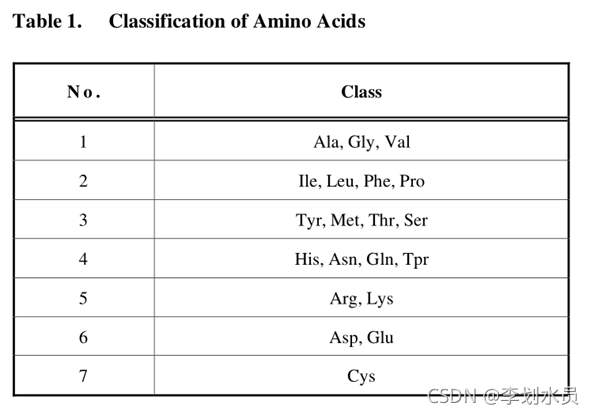
3 计算 C T D
根据依据氨基酸官能团在蛋白质一级结构中发生的变化计算三个描述符:
组成(Composition, C) 1*7
过渡(Transition, T) 1*21
分布(Distribution, D) 1*35 相加为1*63维
以序列VCCPPVCVVCPPVCVPVPPCCV为例
按照上表分类得到 17722
‘1’:[1] ‘7’:[2,3] ‘2’:[4,5]
C:为每组氨基酸在整条氨基酸序列中所占的比例,7组氨基酸共有7个值
C:C[1]=1/5=0.2
C[7]=2/5=0.4
C[2]=2/5=0.4
所以 C:[0.2,0.4,0,0,0,0,0.4]
T: 指一组氨基酸中的氨基酸残基和另外一组氨基酸中的氨基酸残基相邻组合的频率
按照分类大小排序
’17’:1 ‘27’:1 [PI表示第一个组,L表示第二个组,frequency表示该组合出现的频率,L表示这一区域的长度,Index是数组的位置]
T[Index]=Frequency*1/L-1
Index=int((21-(7-PI)*(7-PI+1)/2)+(I-PI-1) 5 ,10
T=[0,0,0,0,0.25,0,0,0,0,0.25,0……]
D:指各组氨基酸中所有氨基酸残基位置的1、第25%、第50%、第75%和整条蛋白质序列长度的比值。
选每一组1,25%,50%,75%,长度组成残基位置
第一组:[1] [2,3]
x: [1,0,0,0,1] x:[1,0,1,1,2]
Index[x-1]/L=0.2
D=[0.2, 0.2, 0.2, 0.2, 0.2, 0, 0……, 0.4, 0.6, 0.4, 0.4, 0.6]
最后拼接起来即可。





 本文详细介绍了局部描述符(LD)方法在蛋白质序列分析中的应用,该方法无需序列比对,主要依赖于氨基酸分类。首先,将蛋白质序列划分为10个特定区域,然后基于氨基酸的偶极性和体积进行分类。接着,计算组成(C)、过渡(T)和分布(D)三个描述符,构建63维特征向量。以实例VCCPPVCVVCPPVCVPVPPCCV说明了C、T、D的计算过程,展示了如何从蛋白质序列中提取这些特征。
本文详细介绍了局部描述符(LD)方法在蛋白质序列分析中的应用,该方法无需序列比对,主要依赖于氨基酸分类。首先,将蛋白质序列划分为10个特定区域,然后基于氨基酸的偶极性和体积进行分类。接着,计算组成(C)、过渡(T)和分布(D)三个描述符,构建63维特征向量。以实例VCCPPVCVVCPPVCVPVPPCCV说明了C、T、D的计算过程,展示了如何从蛋白质序列中提取这些特征。
















 117
117

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








