本文内容速览:
01
植物基因组编辑简介
植物基因组编辑技术历经了三十余年迭代,从早期特异性重组酶(如Cre和Flp)技术,到归巢核酸酶(meganucleases)技术、锌指核酸酶(zinc-finger nucleases,ZFNs)技术和转录激活因子样效应物核酸酶(transcription activator-like effector nucleases,TALENs)技术,再到当前广泛应用的CRISPR-Cas技术。CRISPR-Cas能成为当前基因组编辑的首选技术,主要有三个关键特性:第一,20nt的单链向导RNA(single guide RNA,sgRNA)赋予了CRISPR-Cas较高的特异性,即使在大型植物基因组中也能编辑特异性位点;第二,通过更换不同sgRNA即可编辑不同基因,实验操作简单;第三,能够添加多种功能蛋白,如脱氨酶、逆转录酶、转录增强子或抑制子和多种染色质重塑因子等,以满足不同个性化编辑需求。
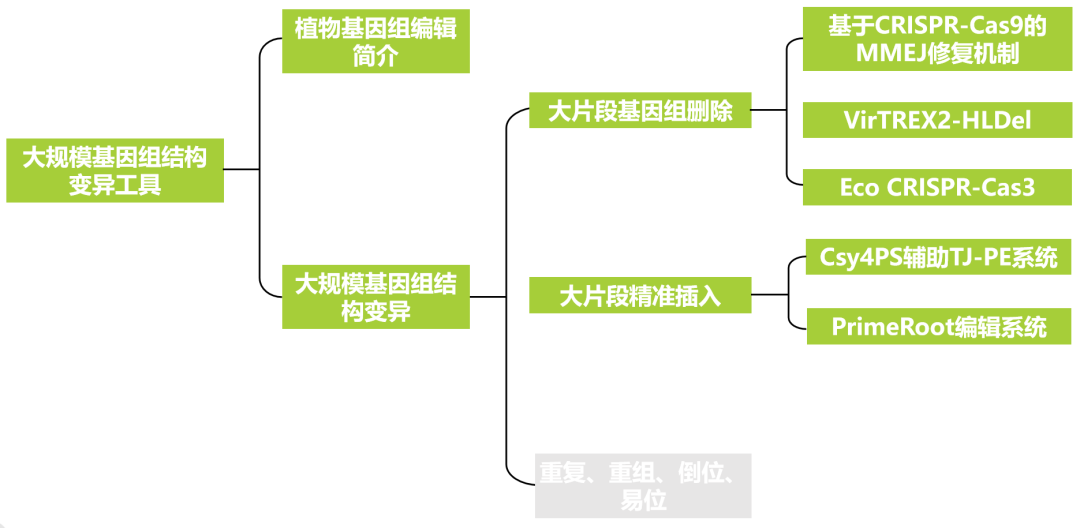
目前以CRISPR-Cas9为代表的基因组编辑工具主要适用于小规模的基因组编辑。该工具利用Cas9蛋白在特定基因组位点产生DNA双链断裂(DNA double-strand break,DSB),DSB诱导非同源末端连接(non-homologous end joining,NHEJ)修复,在特定基因组位点附近或位点内随机产生一个或几个碱基的插入、删除或替换。在CRISPR-Cas9工具的基础上改造获得的碱基编辑工具可实现精准的碱基替换,先导编辑工具可实现精准小片段的插入、删除或替换。这些基因组编辑工具功能多种多样,可编辑编码蛋白的基因、microRNA、lncRNA或启动子调控元件(如增强子)、可编辑多个基因或编辑一个基因内多个位点等等,它们在植物研究和作物育种中发挥着重要作用。
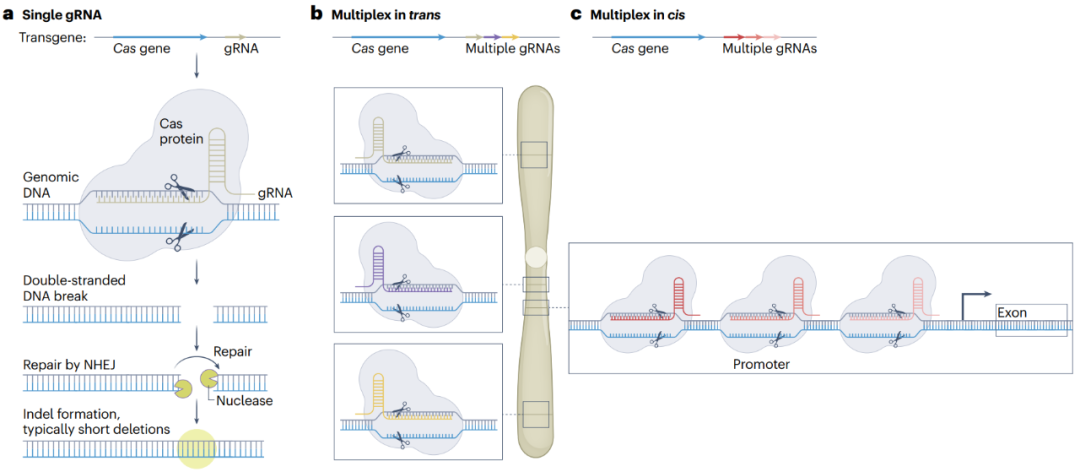
图1 目前以CRISPR-Cas9为代表的基因组编辑工具在特定基因位点内或附近可产生小片段的插入或删除(Gilbertson et al., 2025)。(a)由CRISPR gRNA引导的Cas蛋白切割基因组。在植物中,产生的DSB最常被NHEJ修复,导致1-10bp的短序列删除; (b)反式多重基因组编辑:两个或多个sgRNA将Cas蛋白同时引导到多个基因组位点;(c)顺式多重基因组编辑:两个或多个gRNA将Cas蛋白引导到基因组的同一区域(如基因的启动子),通常会导致gRNA靶位点之间产生较大片段的删除。
但值得注意的是,自然发生的结构变异(structural variation,SV)是作物育种和驯化所依赖的表型变异基础,植物基因组中的结构变异通过自然和人工选择产生了许多非常重要的农业种质资源。因此,有学者认为不能仅仅局限于在基因组中实现小片段的插入、删除或替换,植物基因组编辑的未来需要模拟自然进化过程中植物基因组的重塑机制,即实现大片段删除、插入、重复、重组、倒位和易位等结构变异(Gilbertson et al., 2025)。如果把常规小规模的基因组编辑比作“精雕细琢”,那用“大刀阔斧”来形容大规模基因组结构变异再合适不过。目前实现大规模基因组结构变异的基因组编辑工具刚开始崭露头角,今天伯小远就带领大家了解一下这些工具吧!
02
大规模基因组结构变异
2.1大片段基因组删除
大片段基因组删除可用于简化基因组,去除串联重复基因以及去除由生物合成基因簇编码的整个途径。
2.1.1 基于CRISPR-Cas9的MMEJ修复机制
2020年4月,华南农业大学刘耀光课题组在Plant Biotechnology Journal杂志上发表了一篇题为“Efficient CRISPR/Cas9-based plant genomic fragment deletions by microhomology-mediated end joining”的研究论文。作者提出了一种基于CRISPR-Cas9并利用微同源介导的末端连接(microhomology-mediated end joining,MMEJ)修复机制实现大片段基因组删除的策略。
作者实验室前期用CRISPR-Cas9系统获得了48个发生片段删除的水稻基因编辑群体,靶标设计未事先考虑微同源序列(MHS)。作者首先分析了这48个基因编辑群体的大片段基因组删除效率和删除片段大小,结果显示,发生MMEJ修复的群体与发生NHEJ修复的群体有相近的删除效率,但MMEJ修复产生的删除片段更大(图2a)。
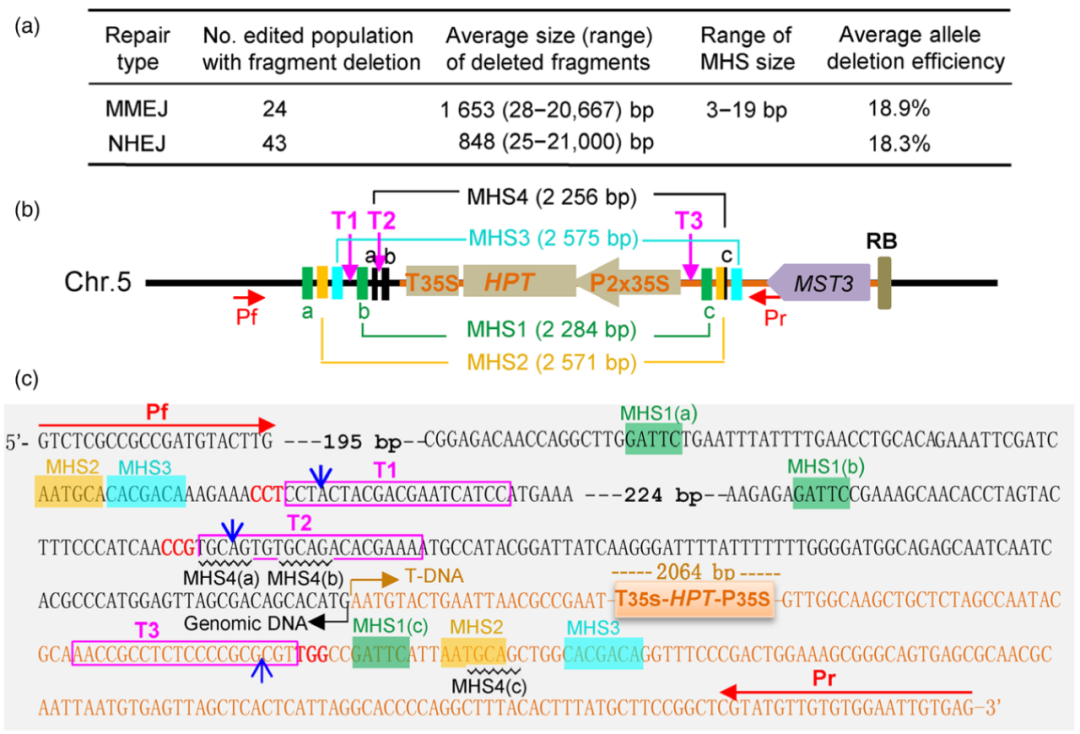
图2 基于CRISPR-Cas9 MMEJ修复机制介导的大片段基因组删除(Tan et al., 2020)。(a)水稻中MMEJ和NHEJ修复机制介导的大片段基因组删除效率和删除片段大小的比较。每个群体为同一载体转化获得的T0个体,其中包含一定比例的MMEJ修复或NHEJ修复介导的大片段基因组删除事件。共分析了48个具有大片段基因组删除的编辑群体,其中19个同时具有MMEJ修复和NHEJ修复介导的大片段基因组删除;(b)转基因水稻材料中抗性基因HPT表达框的示意图,显示了设计的靶标(T1-T3)和MHS(同一组的位点以相同的颜色显示);(c)HPT表达框的侧翼序列,不同颜色标识出不同的MHS和靶标,以及Cas9切割位置(即DSB位点,蓝色箭头)。Pf/Pr引物对用于检测大片段基因组删除。
为了验证MMEJ修复是否有利于大片段基因组删除,作者以一个转基因水稻材料中的抗性基因HPT表达框作为删除目标,通过改良的高效热不对称交错PCR(mhiTAIL-PCR)确定了HPT表达框侧翼的序列,利用在线微同源搜索工具RepFinder(http://skl.scau.edu.cn/repfinder/)分析出HPT表达框侧翼的5-7bp的MHS,再利用基因组编辑综合在线工具CRISPR-GE(http://skl.scau.edu.cn/)设计了三个尽量靠近MHSs的编辑靶标(T1-T3)(图2b、c)。作者构建三个靶标的编辑载体并转化上述转基因水稻,最终在21株T0代编辑植株中,获得了66.7%的HPT表达框删除的植株,其中MMEJ修复介导的删除效率高达52.4%,而NHEJ修复介导的删除效率仅为14.3%(图3b)。
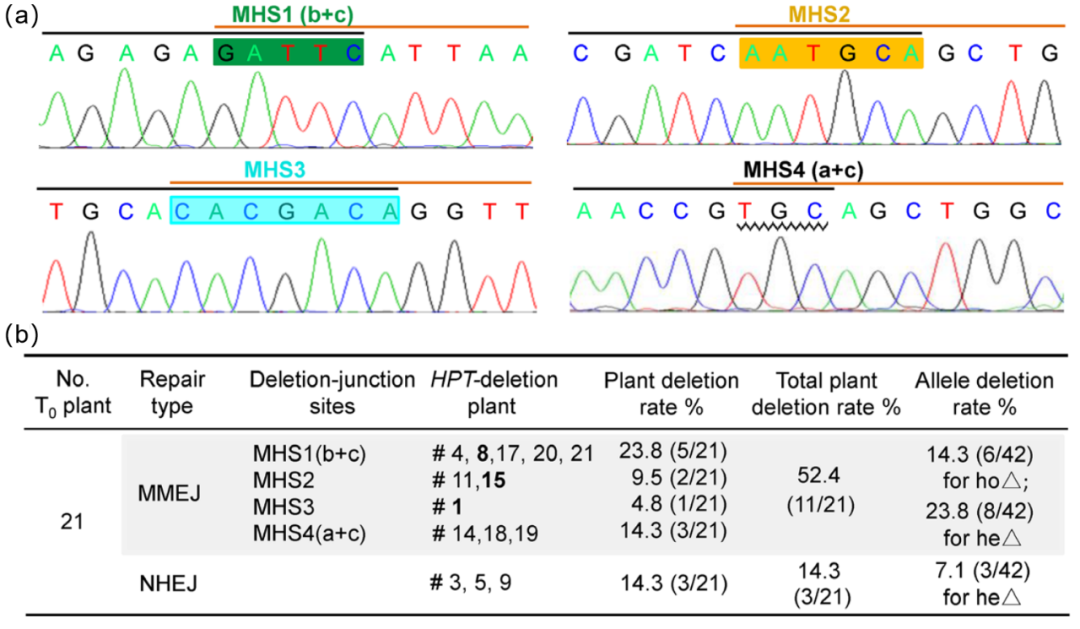
图3 基于CRISPR-Cas9 MMEJ修复机制介导的大片段基因组删除(Tan et al., 2020)。(a)测序峰图显示了11个MMEJ介导的基因组编辑植物中四种类型的删除事件;(b)MMEJ修复和NHEJ修复介导的大片段基因组删除效率。
作者进一步研究发现主要有三个方面影响基于CRISPR-Cas9 MMEJ修复介导的大片段基因组删除效率:(1)MHS与DSB位点距离较近时将有助于片段删除;(2)MHS的长度对MMEJ介导基因组片段删除效率的影响并不显著;(3)使用多个sgRNA同时产生多个DSB可能会增加大片段基因组删除的效率。
2.1.2 VirTREX2-HLDe
常规基于病毒递送的植物基因组编辑系统,一般需要先通过传统的农杆菌介导的方法获得组成型过表达Cas9的植物,再通过病毒载体如烟草脆裂病毒(tobacco rattle virus,TRV)载体,将sgRNA递送至过表达Cas9的植物中,绕过组织培养,最终产生基因编辑植株,但往往只能产生小片段的插入/删除突变。
2024年1月,美国明尼苏达大学Daniel F. Voytas课题组在Plant Physiology杂志上发表了一篇题为“Heritable,multinucleotide deletions in plants using viral delivery of a repair exonuclease and guide RNAs”的研究论文。作者开发了一种基于病毒递送的高效植物基因编辑系统—VirTREX2-HLDel,该系统在编码蛋白质的基因和顺式调控元件中实现了可遗传的大片段基因组删除,并且可以提高低效编辑位点的编辑效率。
研究表明,TREX2是一种外切酶,可加工Cas9产生的DSB 3'端,通过NHEJ或MMEJ修复后,就会产生更大的删除(Weiss et al., 2020)。增强型sgRNA(enhanced sgRNA,esgRNA)是sgRNA融合了tRNAIleu,可增强其在细胞间的移动性,提高基因编辑系统的编辑效率。作者假设,使用RNA病毒载体将TREX2和esgRNA传递给Cas9转基因植物,可能会产生可遗传的大片段基因组删除。为了验证这一假设,作者构建了两种载体系统:VirTREX2-HLDel-1和VirTREX2-HLDel-2,VirTREX2-HLDel-1是包含TREX2和esgRNA的TRV载体,在此基础上,VirTREX2-HLDel-2载体在esgRNA 3'端添加两个com RNA适配体,com RNA适配体可招募与TREX2融合的Com蛋白,增强TREX2在靶标位点的局部富集(图4B)。
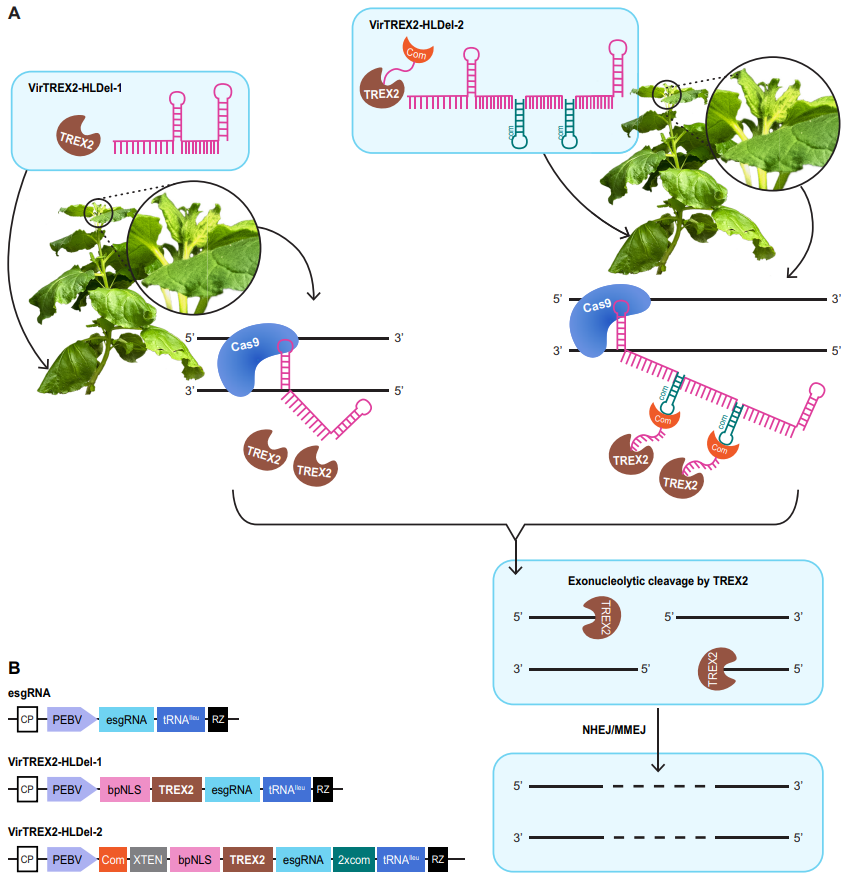
图4 VirTREX2-HLDel产生大片段基因组删除的原理(Liu et al., 2024)。(A)将VirTREX2-HLDel递送至Cas9转基因植物。当TRV在整个植物中系统性移动时,Cas9和esgRNA会在靶标位点产生DSB,TREX2可以切割DSB的3'端,通过NHEJ或MMEJ修复可以产生大片段基因组删除;(B)表达TREX2和esgRNA的TRV载体示意图。CP:外壳蛋白;PEBV:豌豆早期褐变病毒亚基因组启动子;bpNLS:二分核定位信号;XTEN:16个氨基酸的接头;RZ:终止核酶。
作者首先以NbPDS为对象设计了esgRNA,将这些载体和仅含esgRNA的对照载体转化到Cas9转基因烟草中。结果表明,VirTREX2-HLDel-1/2可遗传的大片段基因组删除效率均显著高于对照载体(图5A)。接着,作者针对烟草中的SHOOT MERISTEMLESS(STM)基因的核心K-box和MERISTEM LAYER 1 (ATML1)基因的L1-box这两个顺式调控元件,设计了靶向它们的esgRNA。将VirTREX2-HLDel-1载体和仅含esgRNA对照载体转化到Cas9转基因烟草中,结果表明,VirTREX2-HLDel可以在顺式调控元件中产生比仅含esgRNA的载体更大片段的删除(图5B、C)。作者通过在烟草中靶向miR164e和miR164h,证明了VirTREX2-HLDel可以编辑microRNA,产生大片段基因组删除。通过靶向编辑效率较低的ATML1-1启动子和ATML1-2启动子,证明了VirTREX2-HLDel可以显著提高低效编辑位点的编辑效率,并获得大片段基因组删除的多位点编辑植株。
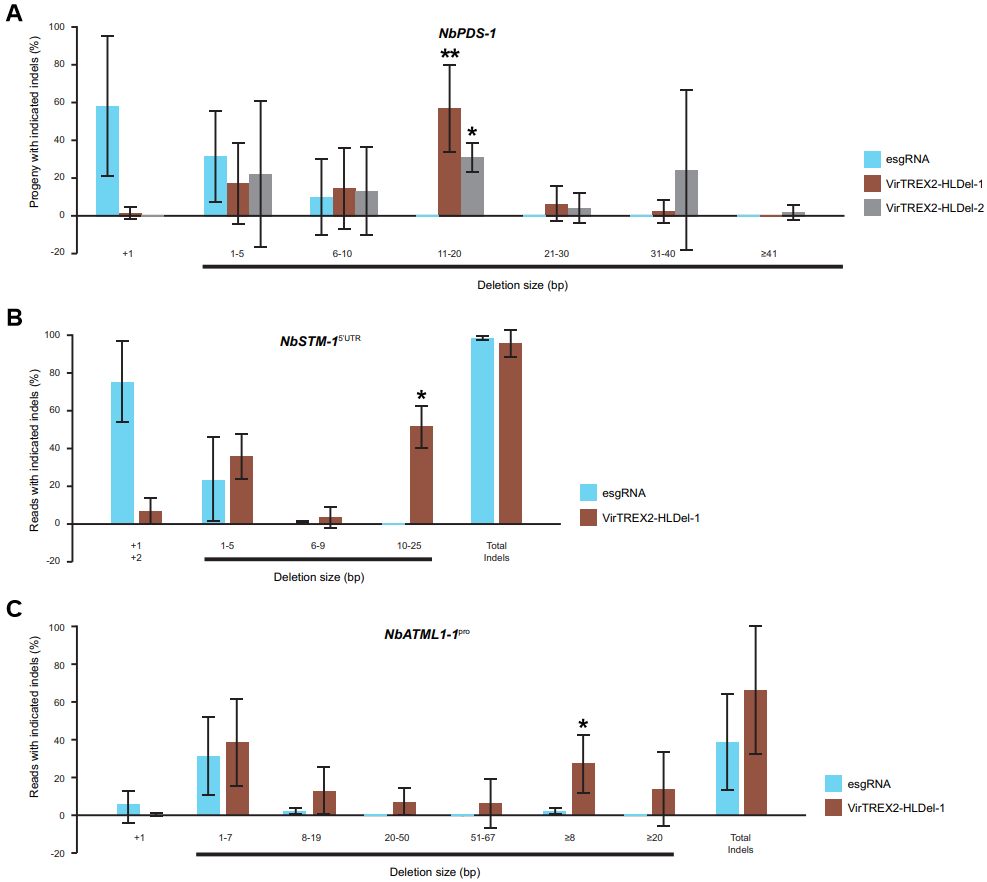
图5 VirTREX2-HLDel在编码蛋白的基因和顺式调控元件中产生可遗传的大片段删除(Liu et al., 2024)。(A)M1幼苗中NbPDS-1可遗传的大片段删除效率;(B)M1幼苗中STM核心K-box(NbSTM-15' UTR)可遗传的大片段删除效率;(C)M1幼苗中ATML1 L1-box(NbATML1-1pro)可遗传的大片段删除效率。
2.1.3 Eco CRISPR-Cas3
源自大肠杆菌的I-E型CRISPR-Cas3(Eco CRISPR-Cas3)系统可以实现大片段基因组删除,但由于该系统需要同时表达6个Cas蛋白和CRISPR RNA(crRNA),使其在植物中的应用极其困难。2025年6月,日本农业食品研究所Seiichi Toki课题组在bioRxiv杂志上发表了一篇题为“Heritable Large Deletions Using Type I-E CRISPR-Cas3 in Rice”的研究论文。作者成功在水稻中实现了Eco CRISPR-Cas3介导的可遗传的大片段基因组删除,这一突破为植物基因组编辑工具增添了重要的新成员。
为了研究Eco CRISPR-Cas3是否可以在水稻中实现大片段基因组删除,作者开发了两种载体(图6A、B):(1)将6个Cas基因表达框分别构建到两个双元载体上,Cas537载体表达EcoCas5、EcoCas3、EcoCas7和潮霉素磷酸转移酶(hpt),Cas6811表达crRNA、EcoCas6、EcoCas8、EcoCas11和新霉素磷酸转移酶Ⅱ(nptⅡ);(2)构建携带4个表达框的“一体化”双元载体,包括crRNA表达框、Cas758表达框(EcoCas7、EcoCas5、EcoCas8)、Cas1163表达框(EcoCas11、EcoCas6、EcoCas3)、hpt表达框,Cas758和Cas1163表达框通过T2A自切割肽融合多个Cas蛋白。在两个双元载体共转化水稻获得的愈伤组织中检测到39-48%的靶基因删除,而“一体化”载体转化水稻获得的愈伤组织中检测到55-71%的靶基因删除。通过液滴式数字RCR(droplet digital PCR,ddPCR)技术测定不同位置的删除效率,发现PAM序列上游7.0kb区域的删除效率为21-61%,这表明Eco CRISPR-Cas3能够以相当高的效率获得大片段基因组删除突变体。
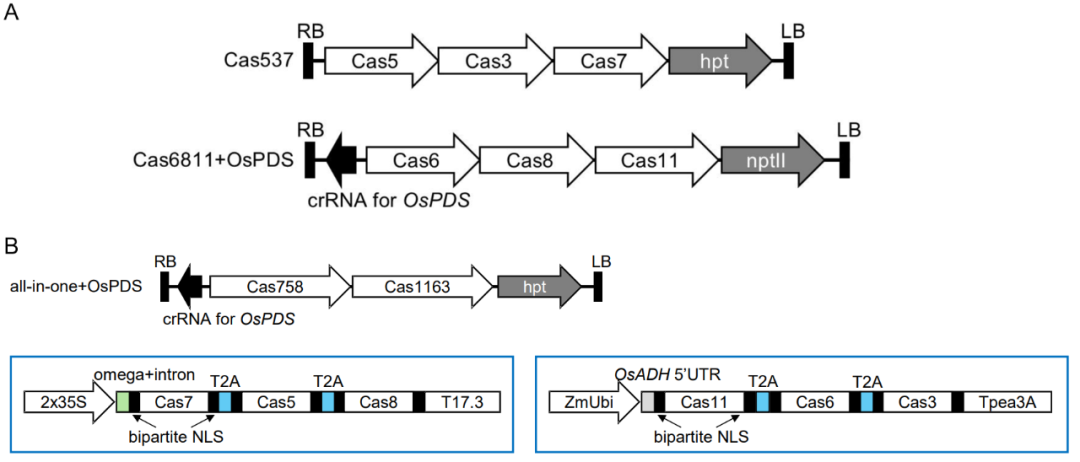
图6 靶向OsPDS的Eco CRISPR-Cas3载体结构示意图(Saika et al., 2025)。(A)两个双元载体结构示意图。hpt:潮霉素磷酸转移酶;nptⅡ:新霉素磷酸转移酶II;RB:右边界;LB:左边界;(B)“一体化”载体结构示意图。Cas758和Cas1163表达框的详细结构如蓝线框所示;绿方块代表CaMVΩ序列和蓖麻豆过氧化氢酶内含子;蓝方块代表T2A自切割肽;黑方块代表细胞核定位信号(NLS);灰方块显示了水稻乙醇脱氢酶的5'端非翻译区;2×35S:双CaMV 35S启动子;T17.3:水稻热休克基因17.3转录终止子;ZmUbi:玉米泛素启动子;Tpea3A:豌豆小亚基核酮糖-1,5-二磷酸羧化酶/加氧酶基因(rbcs)转录终止子。
在OsWx基因编辑实验中,作者获得了一株含有2.8kb基因组删除的T0代植株,其所有T1种子都表现出糯性表型,PCR分析确认了双等位基因突变的稳定遗传,这证明了Eco CRISPR-Cas3介导的大片段基因组删除能够稳定遗传给后代。
2.2大片段精准插入
植物蛋白研究中,大片段精准插入技术可将免疫标签精准嵌入编码目标蛋白的基因位点,用于蛋白检测和机制解析。在植物育种和合成生物学中,该技术则可实现特定农艺性状基因或信号与代谢通路元件的精准导入。与随机插入相比,精准插入的优势在于能够将外源基因置于基因组的“安全港”,既避免干扰内源基因功能,又能保证外源基因的稳定表达。
2.2.1 Csy4PS辅助TJ-PE系统
先导编辑(PE)可通过替换策略有效地插入小片段基因组序列,但在插入大于100bp的片段时面临困难。为实现更大片段插入,在人类细胞中开发了一种名为模板跳跃先导编辑(TJ-PE)技术,可以实现200-800bp的插入(Zheng et al., 2023),但该技术还没有运用到植物中。
2025年5月,河南农业大学苗春波、李俊周课题组合作在Plant Biotechnology Journal杂志上发表了一篇题为“Targeted insertion of large DNA fragments through template-jumping prime editing in rice”的研究论文。作者首先借鉴人类细胞中开发的TJ-PE技术,设计TJ-PE pegRNA(TJ-pegRNA),TJ-pegRNA含一个逆转录模板RTT和两个引物结合位点PBS。再结合Csy4 pre-pegRNA加工系统(Csy4PS)和植物先导编辑器ePPEplus系统,开发出了高效的Csy4PS辅助TJ-PE系统(图7b)。该系统可在水稻中实现长达710bp的大片段精准插入,极大扩展了PE技术在植物中的应用范围,为作物育种提供了强有力的基因编辑工具。此外,先前的研究显示删除植物PE编辑器中的RNase H结构域,能够显著提高常规PE的效率;但作者发现在ePPEplus中引入RNase H结构域,能够显著提升大片段完整插入效率(图7c)。
在T1代纯合植株中,OsC1位点插入207bp pGluB4的植株叶片由紫色转为绿色(图7d),RT-PCR显示目标基因表达显著受抑,证实插入对基因功能的影响。
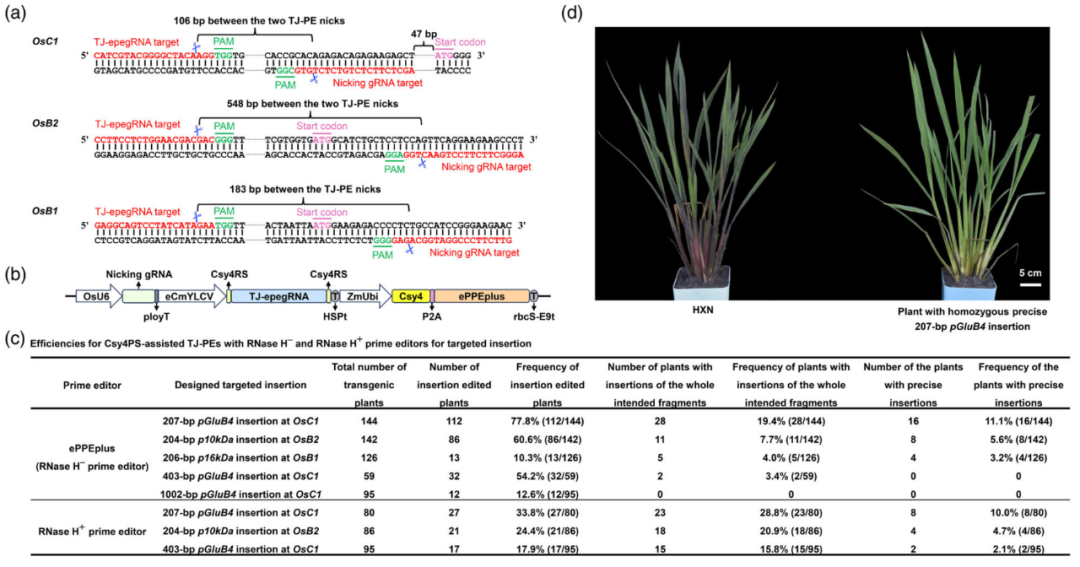
图7 TJ-PE介导在OsC1、OsB2和OsB1位点实现大片段精准插入(Li et al., 2025)。(a)用于插入OsC1、OsB2和OsB1位点的TJ-PE靶标;(b)Csy4PS辅助TJ-PE系统的载体结构示意图。Csy4RS:Csy4识别位点;白框:启动子;灰框:终止子;(c)使用RNase H-ePPEplus系统和RNase H+ePPEplus的Csy4PS辅助TJ-PE的插入效率;(d)野生型粳稻黑香糯(HXN)和精准插入207bp pGluB4的纯合突变体的表型。
2.2.2 PrimeRoot编辑系统
2023年4月,中国科学院高彩霞课题组在Nature Biotechnology杂志上发表了一篇题为“Precise integration of large DNA sequences in plant genomes using PrimeRoot editors”的研究论文。作者开发了一种名为PrimeRoot的编辑系统,能够将高达11.1kb的DNA精准插入植物基因组中,这是植物基因编辑领域迈出的重要一步。
作者首先通过GFP报告系统筛选水稻中位点特异性重组酶(SSR),结果显示Cre和FLP是水稻中最有效的重组酶(图8b、c)。接着,作者评估了PPE+pegRNA、PPE+epegRNA、ePPE+pegRNA和ePPE+epegRNA组合在8个内源性位点精确插入重组酶序列的能力,结果表明ePPE+epegRNA(简称双ePPE系统)的编辑效率最高,但其编辑会随编辑片段大小增大而降低(图8d)。最后,作者将重组酶与双ePPE系统相结合,发展为PrimeRoot编辑系统,可大致分为v1、v2N和v2C这3种类型(图9a)。通过一体化GFP报告系统(图9b)发现PrimeRoot.v2C的编辑效率最高(图9c、d),并进一步证明了PrimeRoot.v2C的实用性。作者对PrimeRoot与基于CRISPR-Cas9的NHEJ修复进行了比较,发现PrimeRoot比基于CRISPR-Cas9的NHEJ修复有更卓越的编辑精度。作者利用PrimeRoot将一个包含PigmR的基因表达框插入到水稻的预测基因组“安全港”位点,该基因表达框由Act1启动子驱动,最终获得了含有预期插入的编辑植株,效率为6.3%,有效提高了水稻对稻瘟病的抗性。
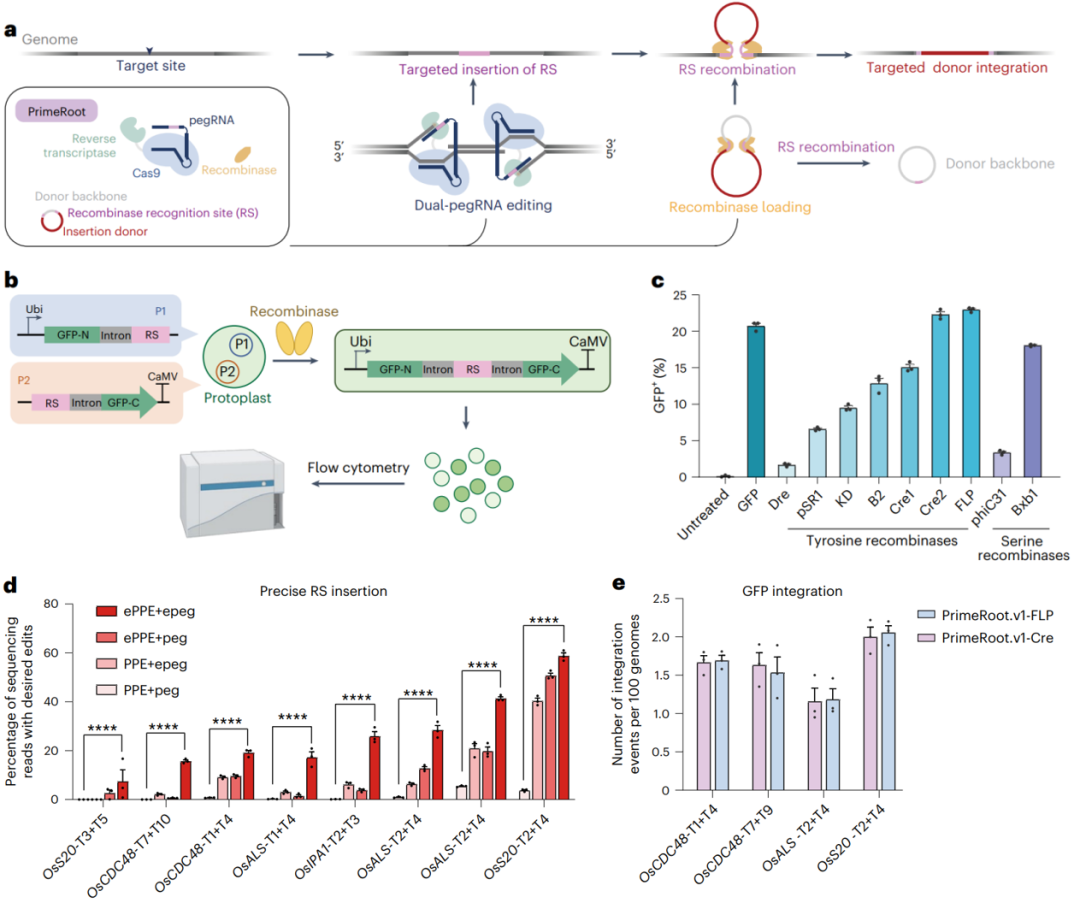
图8 PrimeRoot结合了优化的重组酶和双ePPE系统,以实现大片段精准插入(Sun et al., 2024)。(a)PrimeRoot如何在植物中实现大片段精准插入的示意图。大致原理:先通过双ePPE系统在特定位点精准插入重组酶识别位点,再利用重组酶的重组反应将供体序列插入特定位点上;(b)用于评估植物原生质体中SSR重组活性的GFP报告系统示意图;(c)通过流式细胞术检测原生质体中GFP+的百分比,以反映重组酶活性。评估了7种酪氨酸重组酶和2种丝氨酸重组酶,并将GFP用作阳性对照;(d)PPE+peg、PPE+epeg、ePPE+peg和ePPE+epeg在七个内源性位点精准插入重组酶识别位点的百分比;(e)PrimeRoot.v1-Flp和PrimeRoot.v1-Cre介导四个内源性位点插入GFP的百分比。
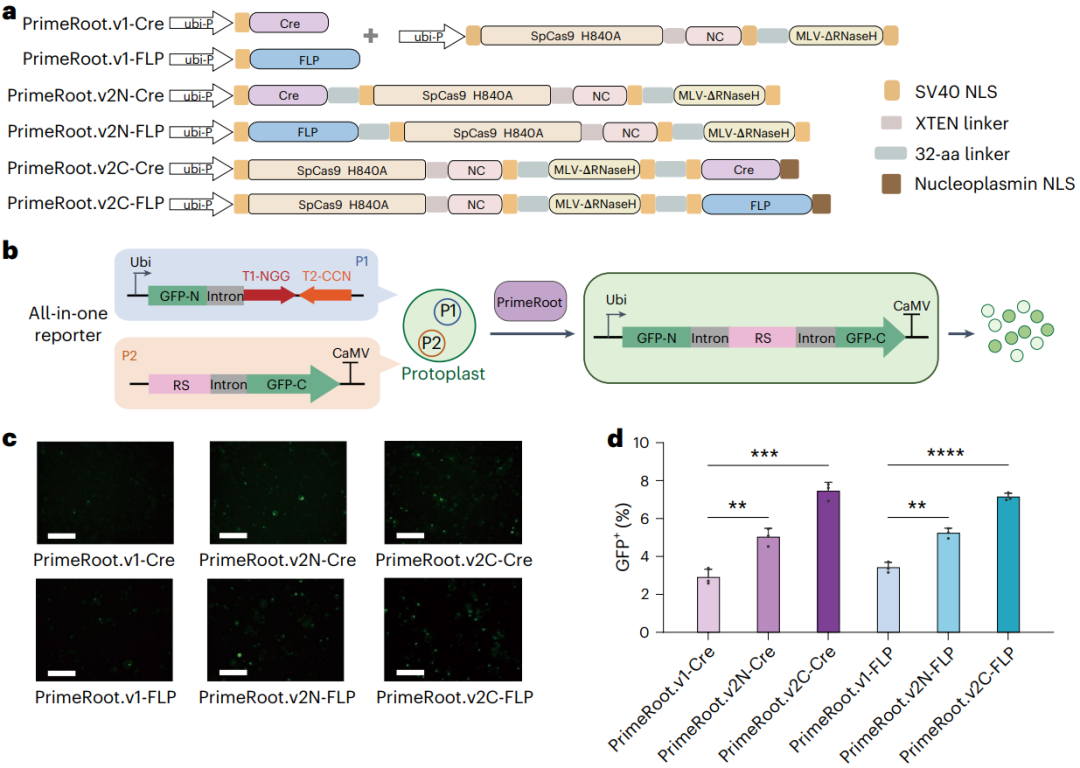
图9 PrimeRoot系统的开发(Sun et al., 2024)。(a)PrimeRoot.v1、PrimeRoot.v2N和PrimeRoot.v2C的示意图。SpCas9 H840A:SpCas9蛋白序列840位的组氨酸(H)被丙氨酸(A)取代,导致HNH核酸酶结构域失活,仅保留RuvC结构域的活性,只能在靶标位点产生单链DNA切口(nick);MLV-ΔRNaseH:RNaseH失活的莫洛尼小鼠白血病病毒逆转录酶突变体;SV40 NLS:猿猴病毒40核定位信号;(b)用于评估植物原生质体中PrimeRoot重组活性的一体化GFP报告系统的示意图;(c)六种不同PrimeRoot编辑系统转化的原生质体的显微荧光图像;(d)六种不同PrimeRoot编辑系统转化的原生质体中GFP+的百分比。
除了前面介绍的大片段基因组删除和精准插入外,大规模基因组结构变异还包括重复、重组、倒位和易位等类型。由于篇幅所限,本期内容就先介绍到这里啦。后续伯小远将继续收集和整理相关研究文献,为大家详细介绍实现这些结构变异的基因编辑工具。如果大家对上述内容感兴趣,建议下载相关文献深入阅读原文,根据各类基因编辑工具的特点,结合自身实验需求,才能更好地加以应用哦!
References:
Gilbertson L, Puchta H, Slotkin R K. The future of genome editing in plants[J]. Nature Plants, 2025: 1-6.
Li F, Hou H, Song M, et al. Targeted insertion of large DNA fragments through template‐jumping prime editing in rice[J]. Plant Biotechnology Journal, 2025, 23(7): 2645.
Liu D, Myers E A, Xuan S, et al. Heritable, multinucleotide deletions in plants using viral delivery of a repair exonuclease and guide RNAs[J]. Plant physiology, 2024, 194(4): 2229-2239.
Saika H, Hara N, Yasumoto S, et al. Heritable Large Deletions Using Type IE CRISPR-Cas3 in Rice[J]. bioRxiv, 2025: 2025.06. 05.658196.
Sun C, Lei Y, Li B, et al. Precise integration of large DNA sequences in plant genomes using PrimeRoot editors[J]. Nature Biotechnology, 2024, 42(2): 316-327.
Tan J, Zhao Y, Wang B, et al. Efficient CRISPR/Cas9‐based plant genomic fragment deletions by microhomology‐mediated end joining[J]. Plant Biotechnology Journal, 2020, 18(11): 2161.
Weiss T, Wang C, Kang X, et al. Optimization of multiplexed CRISPR/Cas9 system for highly efficient genome editing in Setaria viridis[J]. The Plant Journal, 2020, 104(3): 828-838.
Zheng C, Liu B, Dong X, et al. Template-jumping prime editing enables large insertion and exon rewriting in vivo[J]. Nature Communications, 2023, 14(1): 3369.




















 7万+
7万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








