生命活动的奥秘始于基因组DNA中蕴藏的遗传信息,它通过转录和翻译的精密流程,实现从DNA到RNA再到蛋白质的流动。为了确保特定基因在正确的时间和空间表达,调控机制不可或缺。其中,蛋白质与核酸(尤其是DNA)的互作扮演着核心角色——我们不仅需要揭示目标蛋白结合了哪些DNA位点,还需追踪其下游调控途径,以解码疾病或发育的分子基础。那么,研究这类互作主要依赖哪些技术呢?随着组学的发展,若想获取全基因组范围的互作图谱,高通量组学测序技术已成为关键工具。例如,体内方法如ChIP-seq、CUT&Tag和CUT&RUN,能全面捕捉特定蛋白与DNA的相互作用,助力发现组蛋白修饰位点、转录因子靶点或超级增强子;而体外技术如DAP-seq,则提供了一种替代方案来识别转录因子结合位点。如今,无论体内、体外验证还是点对点分析,蛋白-DNA互作的研究手段已相当成熟。本文将带您深入比较三种主流研究已知目的蛋白寻找下游靶点的组学技术:从2007年首次发表的经典ChIP-seq开始,到后续创新的CUT&Tag和DAP-seq,助您精准选择基因组互作研究的利器。
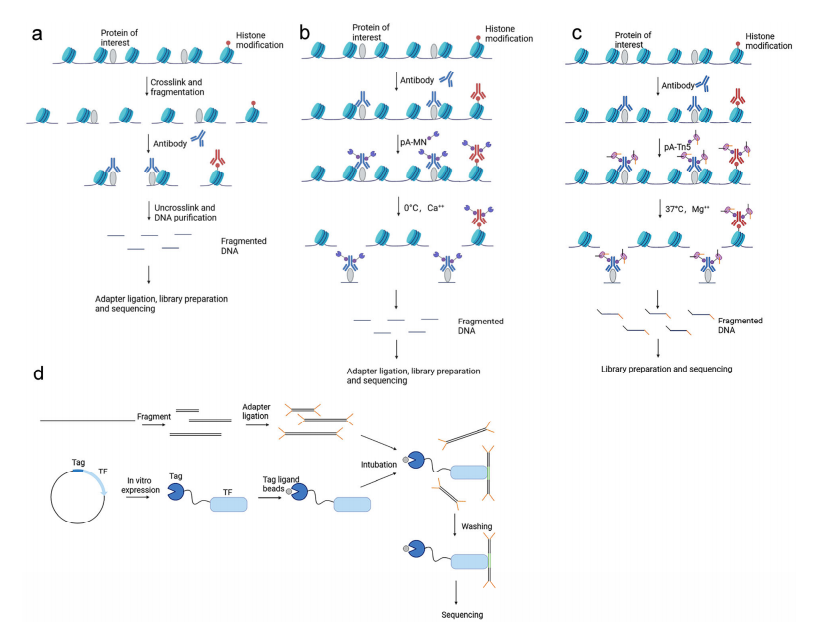
图 已知目的蛋白寻找下游靶点的组学技术(Wang et al., 2023)。
(a)ChIP‐seq;(B)CUT&RUN;(c)CUT&Tag;(d)DAP-seq。
01 ChIP-seq——体内蛋白-DNA结合捕获的金标准
ChIP-seq(Chromatin Immunoprecipitation with highthroughput sequencing,染色质免疫共沉淀高通量测序)是一种用来分析蛋白质与DNA相互作用的研究方法,它将染色质免疫沉淀和高通量测序(NGS)技术结合,可鉴定任意具有DNA结合活性的蛋白在体内的结合位点,免疫沉淀依赖于目的蛋白特异性抗体对蛋白与染色质复合体的富集。
目前的ChIP-seq研究主要包括两大类应用——TF ChIP和Histone ChIP。转录因子作为反式作用因子,与真核基因的顺式作用元件比如启动子、增强子等发生特异性相互作用,从而激活或者抑制基因的转录。ChIP-seq技术可以动态地捕捉到生物体内转录因子在基因启动子区等的结合情况和强度变化,是研究转录调控的有力武器。对于表观遗传学调控的主要方式之一组蛋白修饰,ChIP-seq技术可以获得组蛋白在染色体上的分布情况,从而确定组蛋白修饰相关的特定位点,还可以确定组蛋白修饰酶类的靶标。
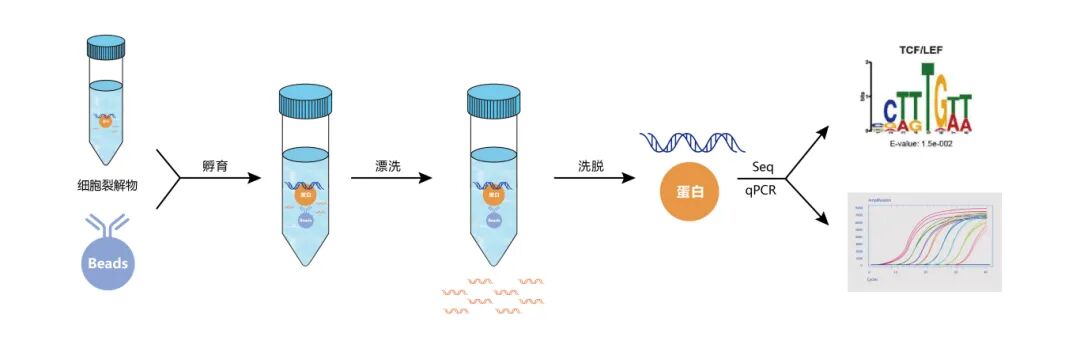
图 染色质免疫沉淀ChIP-seq/qPCR实验流程。
02 CUT&Tag——高效灵敏的蛋白-DNA互作探测新锐
CUT&Tag(Cleavage Under Targets and Tagmentation)是基于NGS的在ChIP-seq和CUT&RUN 基础上进一步优化的新方法。它能在全基因组范围内检测与目的蛋白互作的DNA片段,从而研究组蛋白修饰和转录因子在基因组上结合位置,属于新一代超微量ChIP-seq技术。
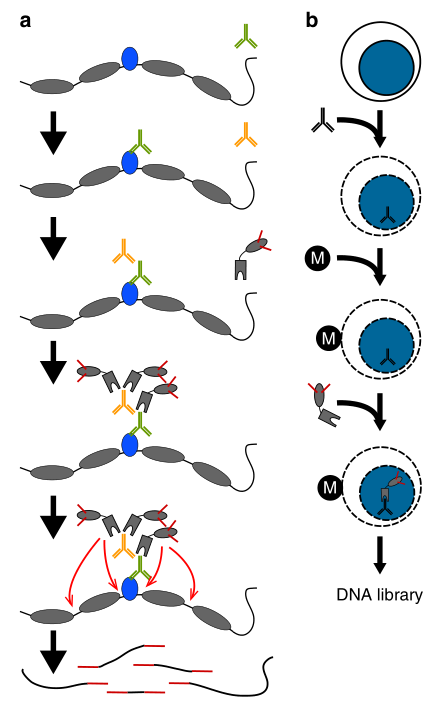
图 CUT&Tag技术原理图(Kaya-Okur et al., 2019)。
03 DAP-seq——体外蛋白-DNA互作分析的开拓者
DAP-seq(DNA affinity purification sequencing,DNA亲和纯化测序)通过体外表达转录因子鉴定在全基因组上的结合位点(Transcription factor binding site,TFBS),是ChIP-seq的重要补充方法。ChIP-seq可以在体内检测TFBS,较为依赖于抗体质量,DAP-seq通过体外表达蛋白解决了这一困扰,并且极大地提高了TFBS富集效率。该技术自问世以来,已被广泛应用于转录调控和表观组学的研究。
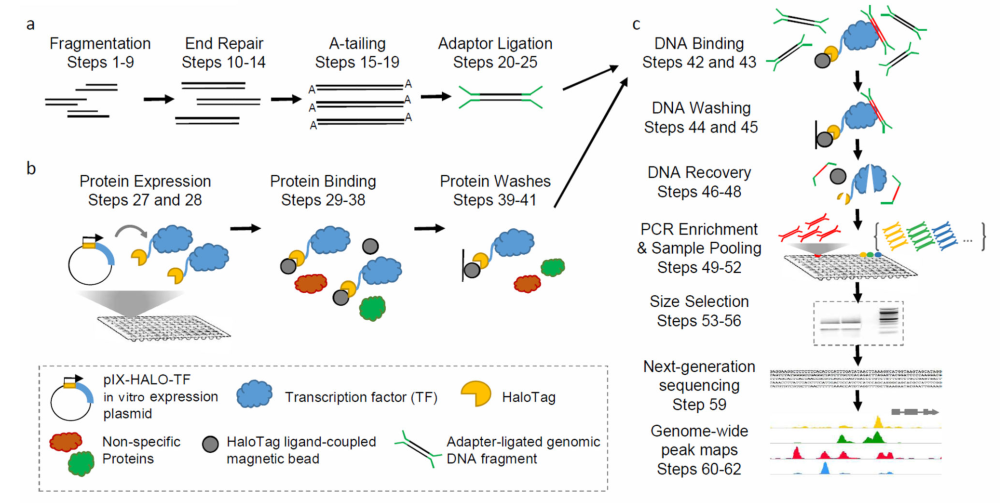
图 DAP-seq实验流程(Bartlett et al., 2017)。
04 技术比较
为直观呈现三种技术的差异,方便大家更好更清晰地进行技术选择,下表分别从物种适用、样本适用、抗体选择、研究范围、样本起始量、信噪比、重复性等不同方面分别进行详细对比说明。


参考文献
[1] Wang M, Li Q, Liu L. Factors and methods for the detection of gene expression regulation[J]. Biomolecules, 2023, 13(2): 304.
[2] Kaya-Okur H S, Wu S J, Codomo C A, et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells[J]. Nature communications, 2019, 10(1): 1930.
[3] Bartlett A, O'Malley R C, Huang S C, et al. Mapping genome-wide transcription-factor binding sites using DAP-seq[J]. Nature protocols, 2017, 12(8): 1659-1672.
[4] Kaya-Okur H S, Wu S J, Codomo C A, et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells[J]. Nature communications, 2019, 10(1): 1930.





















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








