基于SparCC, MENA, LSA, CoNet构建微生物相互作用网络
背景介绍
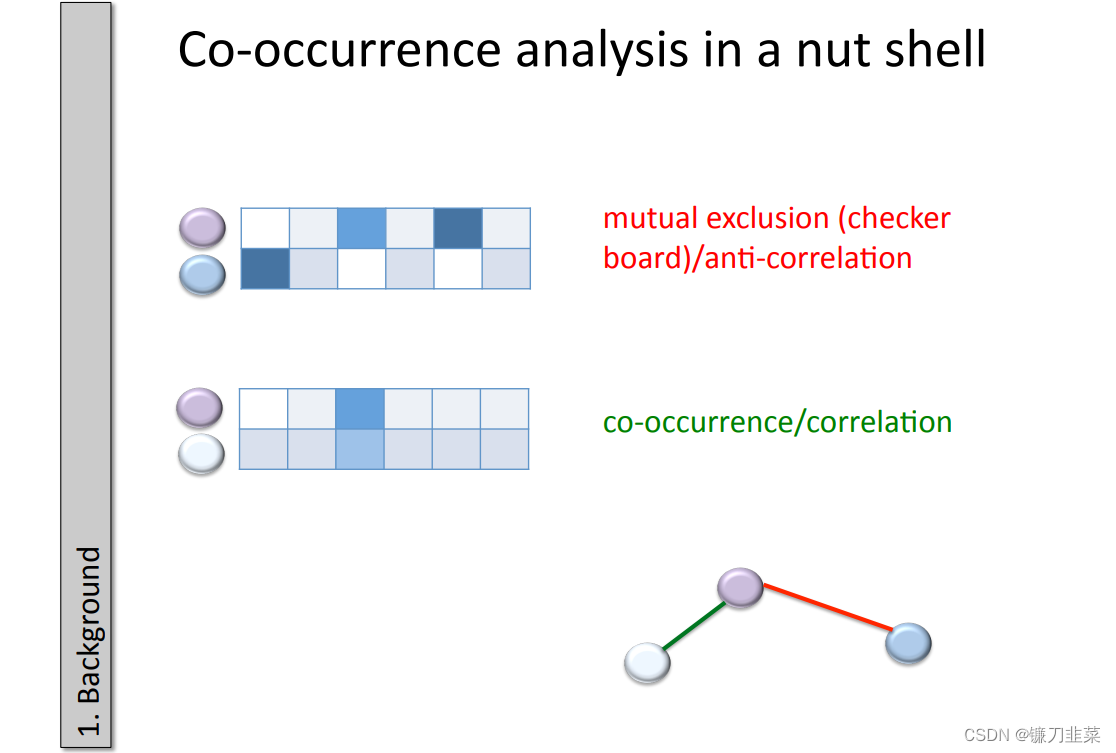
微生物之间的共现(Co-occurrence)可能有以下几种原因,它们可能具有一定的生态关系,或者在生态位上有重叠。
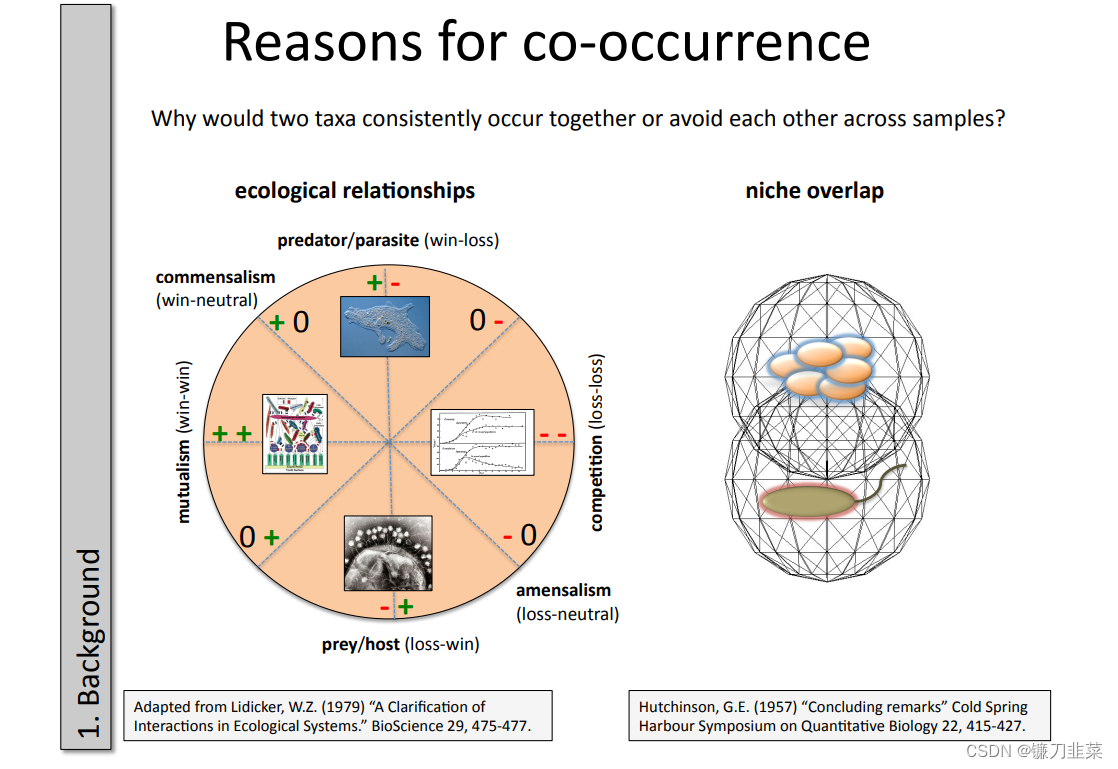
物种的关系可能是此消彼长、或者共增共减。
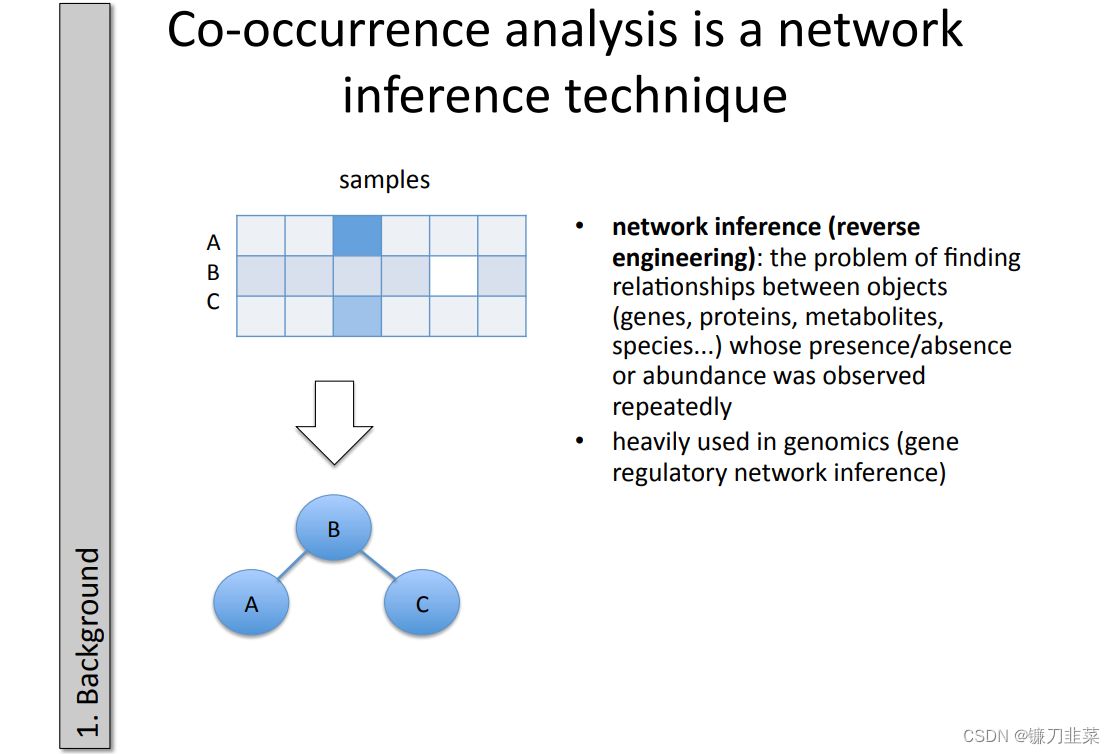
共现分析是一种网络推断技术,可以用来发现不同目标(例如基因、蛋白质、代谢物、物种等)之间的关系,基于可重复的观测中的丰度或者出现/缺失,尤其是应用在基因组学领域,比如基因调控网络推断。
网络推断技术用于宏基因组学及其存在的问题
目前有两种实现网络的技术:
(1)第一种是基于相似性
不管是基于abundance还是incidence的数据,都可以计算成对物种之间的相似度矩阵,并随机化数据反复计算。考察实际相似度与打乱后相似度的显著性差异。最后保留具有意义的相似度并可视化。








 本文介绍了微生物相互作用网络的构建方法,包括SparCC的对数比例相关性计算、MENA的随机矩阵理论、LSA的时间序列分析以及CoNet的组合效应网络推断。这些工具各有特点,如SparCC的迭代计算、MENA的阈值选择、LSA的滞后效应考虑以及CoNet的多种相关性综合。网络推断技术虽能揭示微生物关系,但应注意相似性并不等同于因果性。
本文介绍了微生物相互作用网络的构建方法,包括SparCC的对数比例相关性计算、MENA的随机矩阵理论、LSA的时间序列分析以及CoNet的组合效应网络推断。这些工具各有特点,如SparCC的迭代计算、MENA的阈值选择、LSA的滞后效应考虑以及CoNet的多种相关性综合。网络推断技术虽能揭示微生物关系,但应注意相似性并不等同于因果性。

 订阅专栏 解锁全文
订阅专栏 解锁全文


















 329
329

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










