人工原生动物优化器(Artificial Protozoa Optimizer)是一种受自然启发的群智能优化算法。该算法以原生动物为灵感,提出一种新的人工原生动物优化器(APO)。APO通过模拟原生动物的觅食、休眠和繁殖行为来模拟原生动物的生存机制。从实用性出发,将该方法应用于具有约束条件的连续空间中求解五种常见的工程设计问题。此外,将该算法应用于具有约束条件的离散空间中的多级图像分割任务。该成果于2024年发表在知名一区SCI期刊Knowledge-Based Systems上。
对各种生物现象的研究表明,微生物具有明显的优势。微生物中的细菌、藻类和原生动物具有类似于高等植物和动物体内器官的类似功能,这些器官是通过被称为“强身动物”的特殊结构完成的。这些微生物具有基本的生命特征,包括新陈代谢、繁殖、遗传连续性、变异性和对环境刺激的适应。由于微生物组织简单、复杂度低,通常比较高的生物更有效地利用它们。
1、算法原理
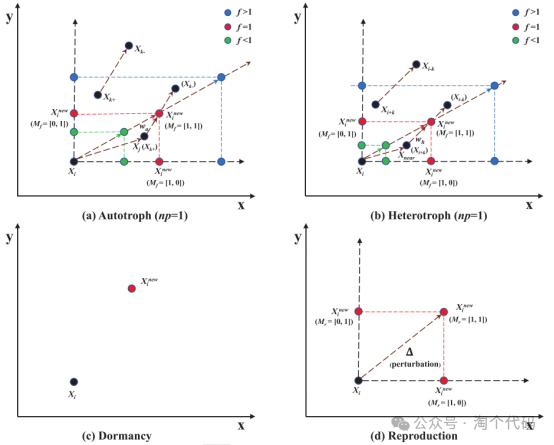
(1)觅食:
原生动物可以通过叶绿体产生碳水化合物来提供营养。如果原生动物暴露在强烈的光强下,它就会离开它的位置,向一个位置移动。如果原生动物周围的光强度较低,则原生动物就会移动到原生动物的位置。对于自养模式,我们提供了以下数学模型:
其中Xinew和Xi分别表示ith原生动物的更新位置和原始位置。Xj是Jth原生动物。Xk表示在Kth配对邻居中随机选择的原生动物。F表示觅食因子,rand表示均匀分布[0,1]区间中的随机数。iter和itermax分别表示当前迭代和最大迭代。NP表示外部因素中邻域对的个数,npmax是NP的最大值。U是自养模式中的一个权重因子。Mf是用于觅食的矢量,其大小为(1×dim)。di代表维度索引
(2)异养方式
在黑暗中,原生动物可以通过吸收周围的有机物来获得营养。假设X近邻是附近食物丰富的地方,原生动物就会向它移动。对于异养模式,我们提出了以下数学模型:
其中Xnear是附近位置,Xi-k表示从配对邻居中选择的第(i-k)个原生动物,其秩指数为i-k。特别是,如果Xi,是Xps,Xi+k也被设为Xps.w是异养模型中的权因子,Rand是[0,1]区间中元素的随机向量。
(3)休眠
为了产生一个新的后代,它将从高斯分布中随机抽取的一个随机值加到单个解向量的每个分量上。这个高斯随机变量控制多少中断被添加到父向量,这有助于算法摆脱局部最优。对于每个父解向量,在第m代高斯变异产生的后代被描述为
其中xmin和xmax分别表示下界向量和上界向量。lbdi和ubdi;分别表示Dith变量的下界和上界。
(4)再生产
在适当的年龄和健康时,原生动物会进行无性繁殖,这是众所周知的。从理论上讲,这种繁殖导致原生动物分裂成两个相同的后代。我们通过产生一个复制的原生动物并考虑一个扰动来模拟这种行为。生殖的数学模型如下:
其中“±”意味着扰动可以是正反向的。M是复制过程中的映射向量,其大小为(1×DIM),每个元素为0或1。
(5)算法参数
APO所涉及的参数如下:
其中PF是原生动物种群休眠和繁殖的一部分。而pfmax是PF的最大值。Pah表示自养和异养行为的概率,Par表示休眠和繁殖的可能性。
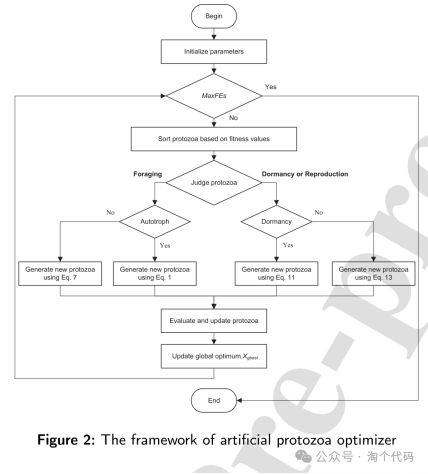
APO优化算法的流程图如下
APO优化算法的伪代码如下

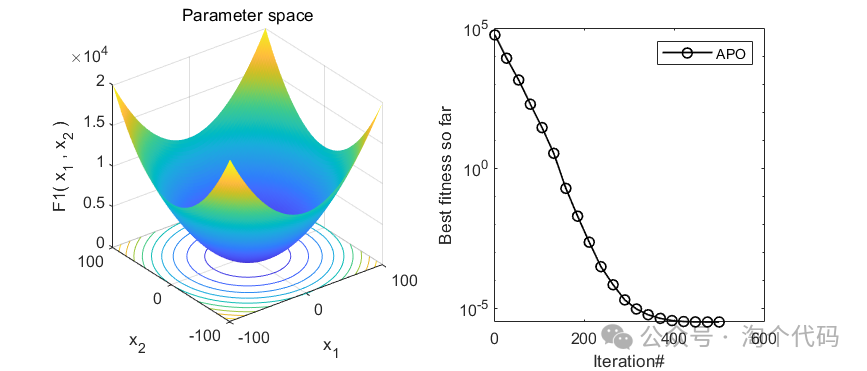
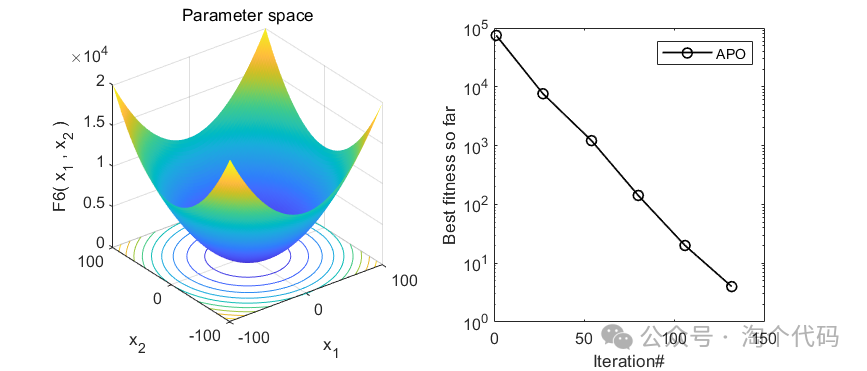
2、结果展示
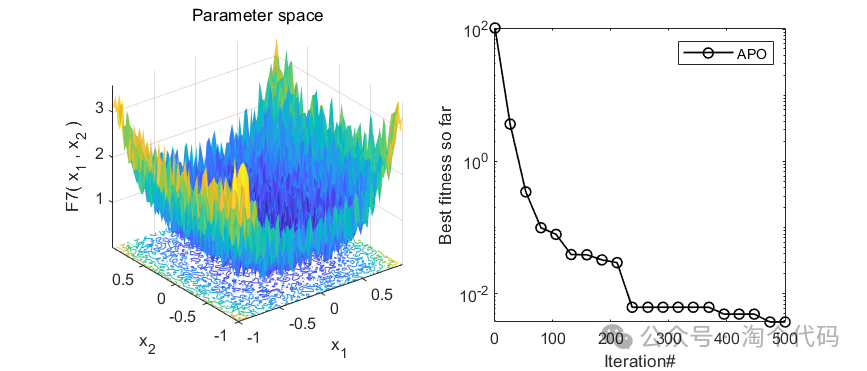
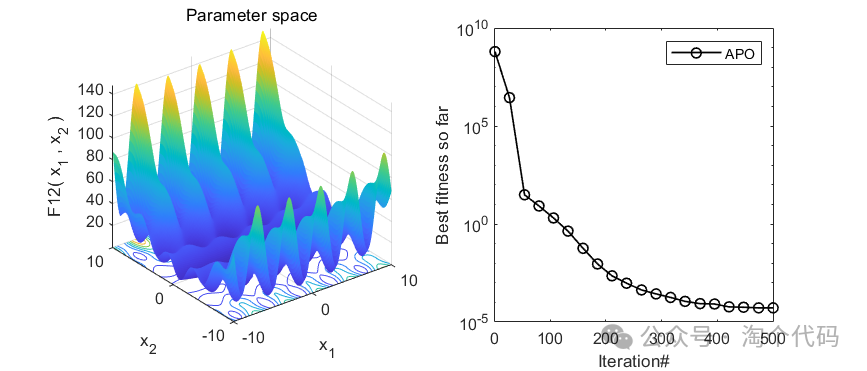
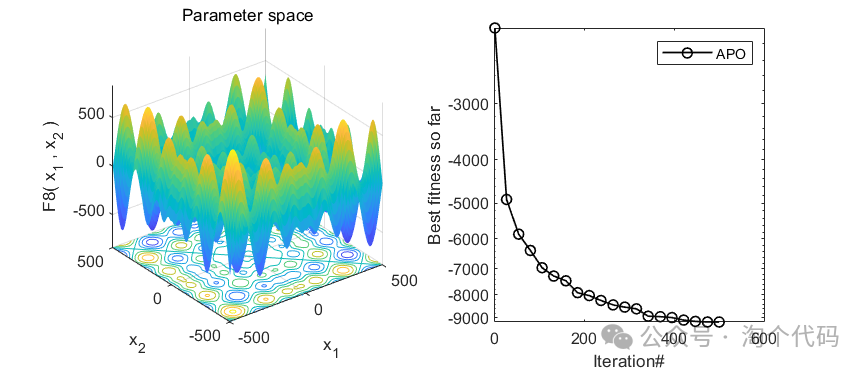
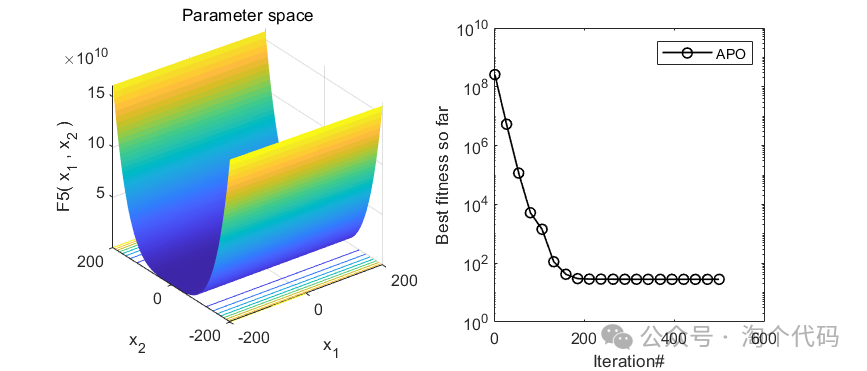
3、MATLAB核心代码
function [bestProtozoa,bestFit,cuver] = APO_func(pop_size,iter_max,Xmin,Xmax,dim,fobj)
% random seeds
stm = RandStream('swb2712','Seed',sum(100*clock));
RandStream.setGlobalStream(stm);
% global best
Xmin = Xmin.*ones(1,dim);
Xmax = Xmax.*ones(1,dim);
ps = pop_size; % ps denotes protozoa size
np = 1; % np denotes neighbor pairs np_max can be set in floor((ps-1)/2)
pf_max = 0.1; % pf_max denotes proportion fraction maximum
%
tic;
protozoa=zeros(ps,dim); % protozoa
newprotozoa=zeros(ps,dim); % new protozoa
epn=zeros(np,dim); % epn denotes effect of paired neighbors
% initilization
for i = 1:ps
protozoa(i,:) = Xmin + rand(1,dim).*(Xmax-Xmin);
protozoa_Fit(i) = fobj(protozoa(i,:));
end
% find the bestProtozoa and bestFit
[bestval,bestid] = min(protozoa_Fit);
bestProtozoa = protozoa(bestid,:); % bestProtozoa
bestFit = bestval; % bestFit
cuver(1) = bestFit;
%% Main loop
for iter=2:iter_max
[protozoa_Fit,index] = sort(protozoa_Fit);
protozoa= protozoa(index,:);
pf = pf_max*rand; % proportion fraction
ri=randperm(ps,ceil(ps*pf)); % rank index of protozoa in dormancy or reproduction forms
for i=1:ps
if ismember(i,ri) % protozoa is in dormancy or reproduction form
pdr=1/2*(1+cos((1-i/ps)*pi)); % probability of dormancy and reproduction
if rand<pdr % dormancy form
newprotozoa(i,:)= Xmin + rand(1,dim).*(Xmax-Xmin);
else % reproduction form
flag=[1,-1]; % +- (plus minus)
Flag=flag(ceil(2*rand));
Mr=zeros(1,dim); % Mr is a mapping vector in reproduction
Mr(1,randperm(dim,ceil(rand*dim)))=1;
newprotozoa(i,:)= protozoa(i,:) + Flag*rand*(Xmin+rand(1,dim).*(Xmax-Xmin)).*Mr;
end
else % protozoa is foraging form
f= rand*(1+cos(iter/iter_max*pi)); % foraging factor
Mf=zeros(1,dim); % Mf is a mapping vector in foraging
Mf(1,randperm(dim,ceil(dim*i/ps)))=1;
pah= 1/2*(1+cos(iter/iter_max*pi)); % probability of autotroph and heterotroph
if rand<pah % protozoa is in autotroph form
j= randperm(ps,1); % j denotes the jth randomly selected protozoa
for k=1:np % np denotes neighbor pairs
if i==1
km=i; % km denotes the k- (k minus)
kp=i+randperm(ps-i,1); % kp denotes the k+ (k plus)
elseif i==ps
km=randperm(ps-1,1);
kp=i;
else
km=randperm(i-1,1);
kp=i+randperm(ps-i,1);
end
% wa denotes weight factor in the autotroph forms
wa=exp(-abs(protozoa_Fit(1,km)/(protozoa_Fit(1,kp)+eps)));
% epn denotes effect of paired neighbors
epn(k,:)=wa*(protozoa(km,:)-protozoa(kp,:));
end
newprotozoa(i,:)= protozoa(i,:)+ f*(protozoa(j,:)-protozoa(i,:)+1/np*sum(epn,1)).*Mf;
else % protozoa is in heterotroph form
for k=1:np % np denotes neighbor pairs
if i==1
imk=i; % imk denotes i-k (i minus k)
ipk=i+k; % ipk denotes i+k (i plus k)
elseif i==ps
imk=ps-k;
ipk =i;
else
imk=i-k;
ipk=i+k;
end
% neighbor limit range in [1,ps]
if imk<1
imk=1;
elseif ipk>ps
ipk=ps;
end
% denotes weight factor in the heterotroph form
wh=exp(-abs(protozoa_Fit(1,imk)/(protozoa_Fit(1,ipk)+eps)));
epn(k,:)=wh*(protozoa(imk,:)-protozoa(ipk,:));
end
flag=[1,-1]; % +- (plus minus)
Flag=flag(ceil(2*rand));
Xnear=(1+Flag*rand(1,dim)*(1-iter/iter_max)).* protozoa(i,:);
newprotozoa(i,:)=protozoa(i,:)+f*(Xnear-protozoa(i,:)+1/np*sum(epn,1)).*Mf;
end
end
newprotozoa(i, : ) = Bounds( newprotozoa(i, : ), Xmin, Xmax );
newprotozoa_Fit(i)= fobj(newprotozoa(i, : ));
end
bin = (protozoa_Fit > newprotozoa_Fit)';
protozoa(bin==1,:) = newprotozoa(bin==1,:);
protozoa_Fit(bin==1) = newprotozoa_Fit(bin==1);
[bestFit,bestid] = min(protozoa_Fit);
bestProtozoa = protozoa(bestid,:);
cuver(iter)=bestFit;
end
recordtime = toc;
end
% Application of simple limits/bounds
function s = Bounds( s, Lb, Ub)
% Apply the lower bound vector
temp = s;
I = temp < Lb;
temp(I) = Lb(I);
% Apply the upper bound vector
J = temp > Ub;
temp(J) = Ub(J);
% Update this new move
s = temp;
end参考文献
[1]Wang X, Snášel V, Mirjalili S, et al. Artificial Protozoa Optimizer (APO): A novel bio-inspired metaheuristic algorithm for engineering optimization[J]. Knowledge-Based Systems, 2024: 111737.
完整代码获取
后台回复关键词:
TGDM900




 文章介绍了一种名为ArtificialProtozoaOptimizer(APO)的新型群智能算法,它模仿原生动物的觅食、异养、休眠和再生产行为解决工程设计问题及图像分割任务。算法详细介绍了其数学模型和MATLAB实现,展示了在约束条件下优化的效果。
文章介绍了一种名为ArtificialProtozoaOptimizer(APO)的新型群智能算法,它模仿原生动物的觅食、异养、休眠和再生产行为解决工程设计问题及图像分割任务。算法详细介绍了其数学模型和MATLAB实现,展示了在约束条件下优化的效果。

















 1054
1054

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










