mnoai 我用的1.2.0的版本,对比代码和效果,明显比opencv变形搞得更好 。而且使用上超级简单
import numpy as np
import pydicom
from monai.transforms import Rand2DElastic
import torch
import matplotlib.pyplot as plt
# 读取DICOM文件
ds = pydicom.dcmread(r"D:\workspace_py\deeplean\Anonymized_20250720\series-00002\image-00043.dcm")
img = ds.pixel_array.astype(np.float32) # 转换为浮点数
# 添加通道维度 (H, W) -> (C, H, W),注意不要添加批次维度
img_tensor = torch.tensor(img)[None, ...] # 形状变为 (1, H, W)
# 创建弹性变形变换
elastic_transform = Rand2DElastic(
prob=1.0, # 应用概率100%
spacing=(20, 20), # 控制网格点间距(像素)
magnitude_range=(1, 3), # 变形幅度范围
rotate_range=None, # 不添加旋转
shear_range=None, # 不添加剪切
scale_range=None, # 不添加缩放
spatial_size=img.shape, # 输出尺寸保持原图大小 (H, W)
mode="bilinear", # 双线性插值
padding_mode="zeros" # 边界填充0
)
# 应用弹性变形
np.random.seed(42) # 固定随机种子
torch.manual_seed(42)
deformed_img = elastic_transform(img_tensor)[0].numpy() # 只需要移除通道维度
# 可视化结果
plt.figure(figsize=(12, 6))
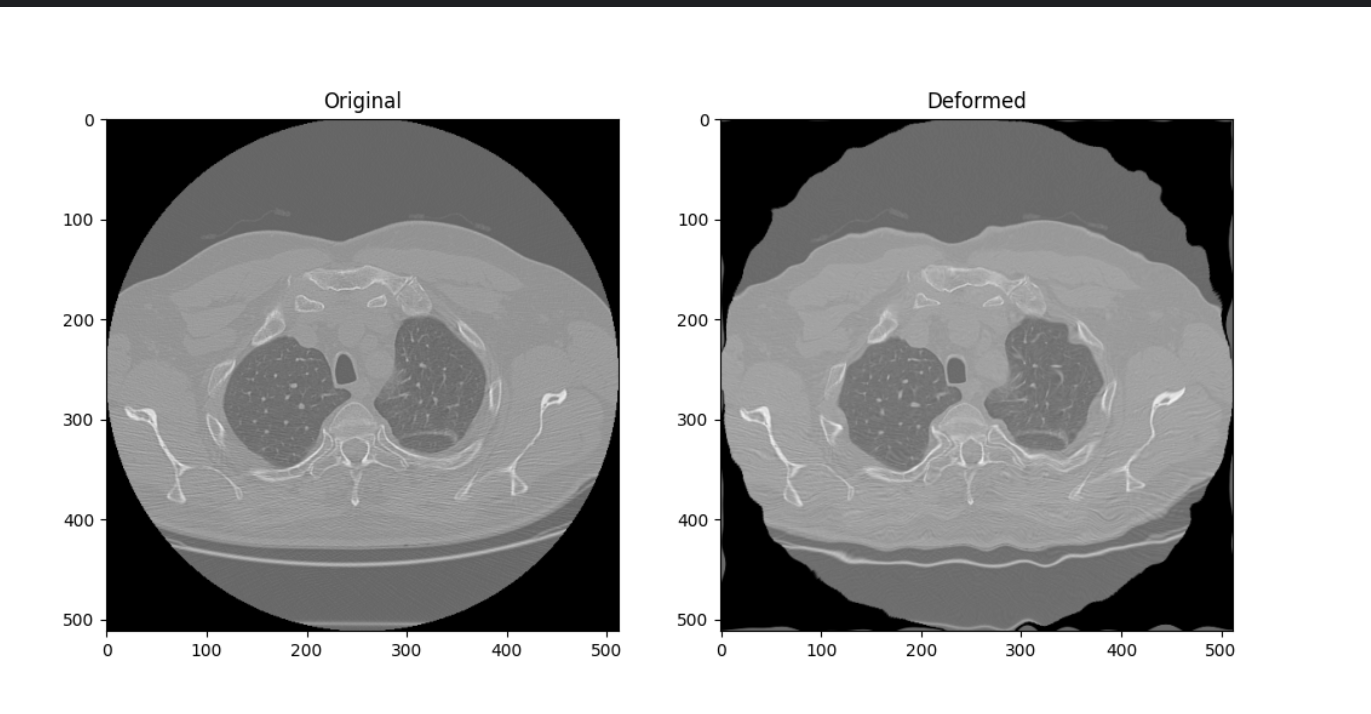
plt.subplot(121), plt.imshow(img, cmap="gray"), plt.title("Original")
plt.subplot(122), plt.imshow(deformed_img, cmap="gray"), plt.title("Deformed")
plt.savefig("elastic_deformation_result.png", bbox_inches="tight")
plt.show()
这段代码的作用很简单:就是把一张医学图像(DICOM 格式)做一些扭曲变形,然后展示原图和变形后的图。
用大白话一步步解释:
- 准备工具:先加载需要的 "工具箱"(各种库),有处理数字的、读医学图像的、做变形的、画图的。
- 读医学图像:找到你电脑里的那个 DICOM 文件(就是你给的那个长路径文件),把它读进来变成电脑能处理的数字格式。
- 调整图片格式:医学图像是二维的(像张照片),但处理工具要求必须多加一个 "通道" 维度(就像给照片套个信封),所以这里加了一个维度,让图片格式符合工具的要求。
- 设置变形参数:
- 肯定要变形(prob=0)
- 变形的网格大小是 20x20(spacing)
- 变形幅度不大(1 到 3 之间)
- 只做扭曲,不旋转、不拉伸(后面几个设为 None)
- 变形后保持原图大小
- 执行变形:固定随机种子(这样每次变形效果一样,方便对比),然后对图片应用刚才设置的变形。
- 展示结果:画两张图,左边是原图,右边是变形后的图,还保存了一张图片到电脑里。
简单说就是:读入医学图像→按工具要求调整格式→设置变形程度→执行变形→看效果。
运行后会弹出一个窗口显示两张图,你能清楚看到原图和扭曲变形后的区别,同时文件夹里会多一个叫 "elastic_deformation_result.png" 的图片文件。

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








