 点击上面“蓝字”关注我们
点击上面“蓝字”关注我们 多位点序列分型(Multilocus sequence typing, MLST)[1]是近年来针对细菌分型发展很快的一种分子生物学分析方法,具有很高的分辨力,既适用于分子流行病学研究,也可用于分子进化学的研究。该方法被广泛应用于病原菌、环境菌和真核生物中。
多位点序列分型(Multilocus sequence typing, MLST)[1]是近年来针对细菌分型发展很快的一种分子生物学分析方法,具有很高的分辨力,既适用于分子流行病学研究,也可用于分子进化学的研究。该方法被广泛应用于病原菌、环境菌和真核生物中。MLST主要是基于测定核苷酸序列的一种细菌分型方法,通过PCR扩增6~10个管家基因内部400~600bp的核苷酸序列,每个位点的序列根据其发现的时间顺序赋予一个等位基因编号,每一株菌的等位基因编号按照指定的顺序排列就是它的等位基因谱,也即是这株菌的序列型(sequence type, ST),每个ST均代表一组单独的核苷酸序列信息。通过比较ST可以发现菌株的相关性,即密切相关菌株具有相同的ST或仅有极个别基因位点不同的ST,而不相关菌株的ST至少有3个或3个以上基因位点不同。
 关于如何获得目标菌株的ST型呢?接下来就教大家如何使用相关工具获得相应菌株的ST型,以便后续的分析和研究。
关于如何获得目标菌株的ST型呢?接下来就教大家如何使用相关工具获得相应菌株的ST型,以便后续的分析和研究。 01选择目标菌株,获取目的基因
01选择目标菌株,获取目的基因 以鲍曼不动杆菌[2](Acinetobacter baumannii )为例。02管家基因和相应引物设计方案的确定
以鲍曼不动杆菌[2](Acinetobacter baumannii )为例。02管家基因和相应引物设计方案的确定
对于数据库中已有相关方案的菌株,直接通过网站获取相应管家基因和引物设计方案[3](https://www.ncbi.nlm.nih.gov/或http://pubmlst.org/); 选择鲍曼不动杆菌 (https://pubmlst.org/abaumannii/info/primers_Pasteur.shtml)。

整理序列为.txt格式,提交网站(https://pubmlst.org/bigsdb?db=pubmlst_abaumannii_pasteur_seqdef&set_id=2&page=sequenceQuery)进行序列比对,获得每个管家基因的等位基因编号。

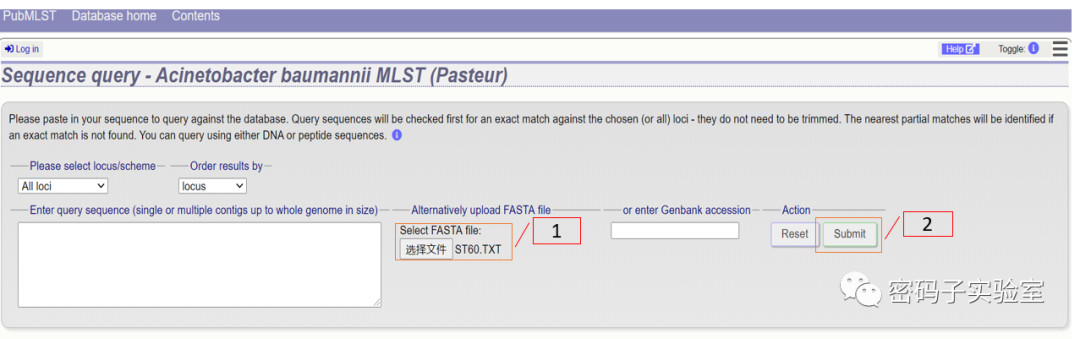

获得目标菌株的ST型,就可以进行下一步的分析,如BLAST比对和MEGA构建系统发育树分析。
 05参考文献
05参考文献[1]. Maiden MC, Bygraves JA, Feil E, et al. Multilocus sequence typing: a portable approach to the identification of clones within populations of pathogenic microorganisms. Proc Natl Acad Sci U S A. 1998;95(6):3140-3145. doi:10.1073/pnas.95.6.3140
[2].Castillo-Ramírez S, Graña-Miraglia L. Inaccurate Multilocus Sequence Typing of Acinetobacter baumannii. Emerg Infect Dis. 2019;25(1):186-187. doi:10.3201/eid2501.180374
[3].Jolley, K.A., Maiden, M.C. BIGSdb: Scalable analysis of bacterial genome variation at the population level. BMC Bioinformatics 11, 595 (2010). https://doi.org/10.1186/1471-2105-11-595
 关于MLST分子分型小伙伴们会了吗?获得更多咨询或资料,请关注下方微信公众号。
关于MLST分子分型小伙伴们会了吗?获得更多咨询或资料,请关注下方微信公众号。

 微信号:mimazilab生物信息学实操 实验操作技能
微信号:mimazilab生物信息学实操 实验操作技能科研绘图技巧 行业动态播报
-这里只有干货 扫码关注我们-
 (添加小编微信,邀您入群)微信群:科研绘图互助交流-科研问题互助 经验分享-
(添加小编微信,邀您入群)微信群:科研绘图互助交流-科研问题互助 经验分享-

QQ群号:831027917
生物信息互助交流- 生信互助 脚本共享-



 本文介绍了MLST方法在细菌分型中的应用,通过PubMLST网站获取管家基因信息,进行序列比对,确定菌株的序列型(ST),并利用ST进行相关性分析,包括BLAST比对和系统发育树构建。提供了一个具体示例——鲍曼不动杆菌的分型步骤。
本文介绍了MLST方法在细菌分型中的应用,通过PubMLST网站获取管家基因信息,进行序列比对,确定菌株的序列型(ST),并利用ST进行相关性分析,包括BLAST比对和系统发育树构建。提供了一个具体示例——鲍曼不动杆菌的分型步骤。

















 691
691

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








