题目分析
先打一张碱基之间的相似度的表
int c[5][5]={{5, -1, -2, -1, -3},
{-1, 5, -3, -2, -4},
{-2, -3, 5, -2, -2},
{-1, -2, -2, 5, -1},
{-3, -4, -2, -1, 0}};
设两个序列分别为 a, b
用 f[i][j] 表示两个序列中 a[i] 与 b[j] 配对时最大的相似度
发现f[i][j]可以由三种状态转移过来
1、a[i] 与 b[j] 对应

如图所示,可以看出此时 f[i][j] = f[i-1][j-1] + c[a[i]][b[j]]; //此处a[i] 和 b[j] 指字母对应的数字
2、a[i] 与 ‘ ’ 对应
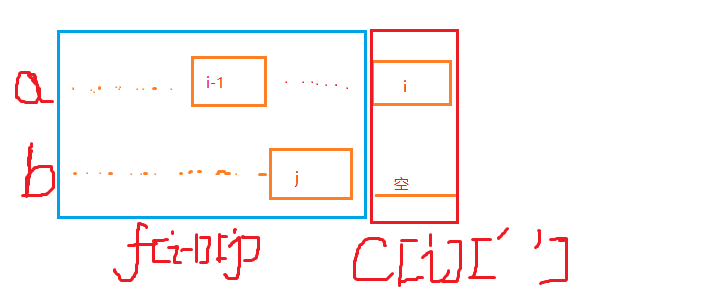
此时 f[i][j] = f[i-1][j] + c[a[i]][‘ ’];
3、b[j] 与 ' '对应
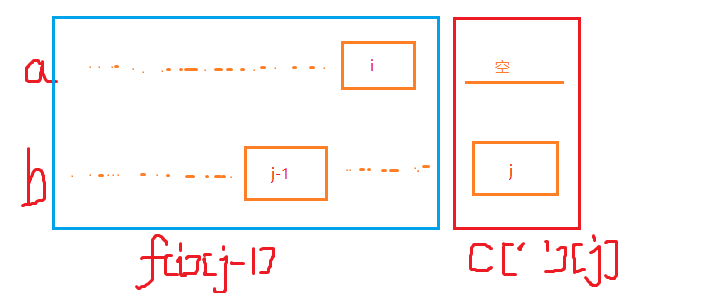
f[i][j] = f[i][j-1] + c[‘ ’][b[j]];
注意
1、在转移前要判断 i 和 j 的值 > 0 ,避免一些问题
2、因为相似值可能 < 0, 所以初值赋为 -无穷大 (0xcf),f[0][0]赋为0;
代码
#include <bits/stdc++.h>
using namespace std;
int l1, l2, f[105][105], q[100];
int c[5][5]={{5, -1, -2, -1, -3},
{-1, 5, -3, -2, -4},
{-2, -3, 5, -2, -2},
{-1, -2, -2, 5, -1},
{-3, -4, -2, -1, 0}};
char a[105], b[105];
int main() {
q['A'] = 0, q['C'] = 1, q['G'] = 2, q['T'] = 3; //标记各种字母对应的数值,‘’对应4
scanf ("%d %s %d %s", &l1, a + 1, &l2, b + 1);
memset (f, 0xcf, sizeof(f)); f[0][0] = 0;
for (int i = 0; i <= l1; i++)
for (int j = 0; j <= l2; j++){
if (i and j) f[i][j] = f[i-1][j-1] + c[q[a[i]]][q[b[j]]];
if (j) f[i][j] = max (f[i][j], f[i][j-1] + c[4][q[b[j]]]);
if (i) f[i][j] = max (f[i][j], f[i-1][j] + c[q[a[i]]][4]); //括号有点多
}
printf ("%d\n", f[l1][l2]);
return 0;
}







 本文介绍了一种基于相似度表的DNA序列比对算法,通过动态规划方法计算两个DNA序列的最大相似度。算法使用5x5矩阵存储碱基间的相似度,并通过三种状态转移计算最大相似度。
本文介绍了一种基于相似度表的DNA序列比对算法,通过动态规划方法计算两个DNA序列的最大相似度。算法使用5x5矩阵存储碱基间的相似度,并通过三种状态转移计算最大相似度。
















 1028
1028

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








